
#本教程部分参考B站15天入门生物信息教程,在开启以下教程前,请务必看看我前面两个教程,Linux系统上安装R语言(https://www.bilibili.com/read/cv24718269)和下载好转录组(https://www.bilibili.com/read/cv24719254)。
#1,我们对上次下载的转录组进行实战分析,首先进行质量控制,使用fastp软件。
conda list#查看目前已经安装的包
conda search fastp#查看版本
conda install fastp=0.23.2#建议装个偏旧的版本
fastp-h#检查是否安装成功,有东西出来就行
#2,对文件重命名,回看NCBI,SRR22954651- SRR22954653是野生型,另外3个就是过表达了。
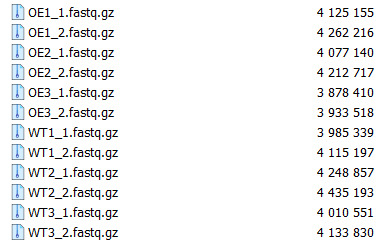
#3,生成sample.txt文件,每行后面有个空格,最后一行是空行:
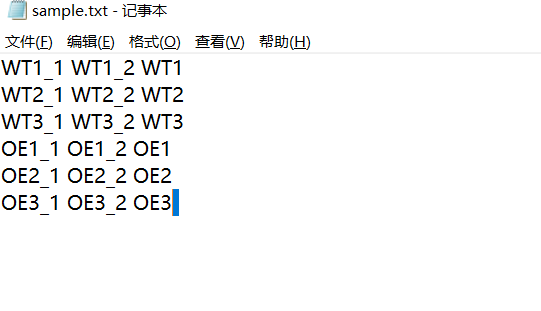
#4,生成批量命令脚本
awk'{print "fastp -i "$1".fastq.gz -I "$2".fastq.gz -o "$1".clean.fq.gz -O "$2".clean.fq.gz -h "$3".html &"}'sample.txt>command_fastp.sh
#5,看看command_fastp.sh文件
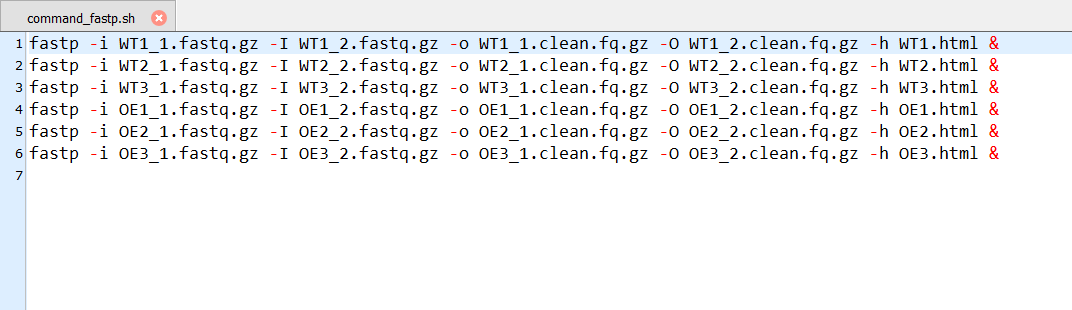
#6,运行脚本
sh command_fastp.sh
#7,或者将步骤3-6改为使用for循环:
for i in {1..3};do fastp -i WT${i}_1.fastq.gz -I WT${i}_2.fastq.gz -o WT${i}_1.clean.fq.gz -O WT${i}_2.clean.fq.gz -h WT${i}.html;done;
for i in {1..3};do fastp -i OE${i}_1.fastq.gz -I OE${i}_2.fastq.gz -o OE${i}_1.clean.fq.gz -O OE${i}_2.clean.fq.gz -h OE${i}.html;done;
#8,等到html文件出来就行,这里大概用了15min,查看结果,有clean data,还有HTML的报告,报告解读主要看Q30>85%,它这个上传文件就是质控过的,所以质控前后的数据基本一致。目前生物公司测序后的数据基本也是像这样质控完了,才会给客户,而且质量基本都不错。但是以防万一,还是要自己质控一遍的。
#eva
#加个尾图
