氨基酸,碱基,元素的增删改
使用内置的Builder插件即可。
实战注意事项:在添加氨基酸的时候,如果C末端残基是以OXT结尾的氨基酸,就无法添加,应该把OXT去掉或者采取其他方案。
窗口拆分和合并
在Display->Toggle Floating 可以拆分这2个窗口,快捷键是Ctrl+E,
设置和查看工作路径
点击1.1菜单窗口 File->Working Directory->Change 可以查看现在的工作目录在哪里,也可以设置新的路径作为工作目录。
下载蛋白
从PDB网站上下载蛋白,如 4hbk.pdb, 也可以直接通过PyMOL 下载蛋白,点击菜单栏中的 File->Get PDB,如下图图所示, 在PDB ID 对应的框中填入PDB的编号就可以了,不要包含后缀.pdb
下面对Show 着重讲解: show 有2类操作方法:
- show as 分别点击S->as->cartoon 和S->as->stick,
我们可以观察到AS模式是把原有的渲染模式抹除后再重新渲染,经过上述操作后仅仅显示stick形式
- show 点击S->as->cartoon 再点击S->stick;
我们可以观察到SHOW方法,是保留原有的渲染,再添加新的渲染。
蛋白对象的action操作
- 点击 A->preset->b-factor putty 基于bfactor数值显示蛋白的柔性
- 点击 A->preset->publication 高质量出版标准
第四部分 Action->generate 操作
- 显示蛋白的静电势图 Action->generate->vacuum_electrostatics->protein contact potential 就可以了
对象的Label操作
添加label
调整配体的位置
pymol中包含配体和蛋白,如何调整配体和蛋白的相对位置。
命令相关
下载蛋白(fetch)
fetch 命令可以根据PDB ID编号下载蛋白结构到工作路径,并载入到PyMOL中显示,在PyMOL中的object的名字默认是PDB ID,可通过name参数进行修改。 从PyMOL 1.8开始,默认的格式是cif,可通过type参数修改文件的格式,也支持根据配体ligand ID的编号下载小分子。
示例:
- fetch 6FYZ # 下载编号是6FYZ的蛋白,文件格式是cif。
- fetch EBE # 下载编号是EBE的配体小分子,文件格式是cif。
- fetch 6MVD,name=”test”,type=pdb # 下载文件格式为pdb的6MVD的蛋白,载入PyMOL中设置名字为test.
type后面的参数不是字符串,不能加引号
type 支持的文件格式有:
type = cif|mmtf|pdb|pdb1|2fofc|fofc|emd|cid|sid|cc
载入amber轨迹文件 load
这里我以rst7文件为例
pymol 支持amber的trj(rst7) 格式和top格式 。
load in.prmtop,mol
load in.rst7,mol
实战:
load 6z5r_wm5_final_U2H.prmtop,mmm
load_traj 6z5r_wm5_final_U2H.inpcrd,mmm,format=rst7
设置背景颜色(bg_color)
设置背景颜色为白色:
bg_color white
设置surface的颜色(cartoon_color)
设置surface的颜色为red
set surface_color,red
注解
注意颜色是有优先级的,默认设置的是atom的颜色,如果没有设置surface的颜色,则继承atom的颜色
体验下
set surface_color,red
color blue
unset surface_color
设置surface的大小(solvent_radius)
设置surface的大小,把surface设置细一点
set solvent_radius, 1.6
set solvent_radius, 0.8
绘制蛋白截面图clip
需要ray后才能看到截面的效果。
as surface
clip near,-10
ray
注解
为了获得想要的效果,多次尝试clip的数值。 也可以通过滚动数据获得切面
设置截面的颜色
set ray_interior_color, grey
set ray_interior_color, grey,obj01
开关单双键模式 valence
set valence,1 开启双键模式。
set valence,0 关闭双键模式。
示例
fetch BUH
set valence,1
set valence,0
效果如下:
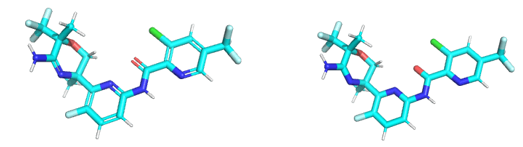
开启球棍模型ball-stick
set stick_ball,1
set stick_ball_ratio,1.7
as stick
效果如下图所示:
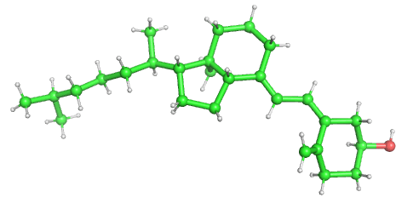
开启球棍模型ball-stick
点击 Action -> preset -> ball and stick
效果如下图所示:
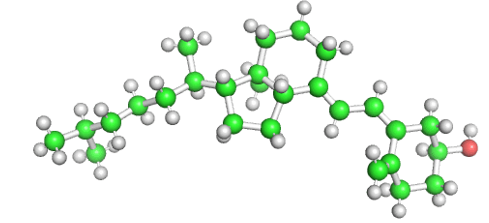
设置相互作用的样式
- 怎么像这样把相互作用的线调粗?
set dash_radius, 0.3
- 把虚线变成实线?
set dash_gap,0
- 设置实线区域的长度
set dash_length, 0.8
保存图片(png)
png filename.png 可以使用该命令进行快捷保存图片
高清图片(ray)
制作高清图片, 制作完图片后输入ray命令,然后保存图片。
保存蛋白序列
save xxx.fasta,xxx
复制pymol 中特定的序列
fetch 4bhk
save 1.fasta, 4bhk and chain A and resi 200-210
设置两个光源 two_sided_lighting
set two_sided_lighting,1
当蛋白表面有破损的时候,ray的时候可能会出现黑洞现象,如下图所示。
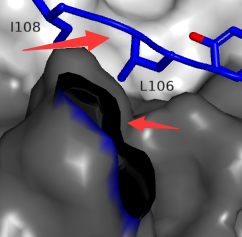
使用上述命令,设置两个光源就可以消除黑洞现象。
设置label的小数位数
默认距离和角度是1位,通过下面命令可以修改,显示的位数。
set label_digits,2
set label_distance_digits,2
set label_dihedral_digits,2
set label_angle_digits, 2
设置label的字体大小
label字体的默认大小是14pt, 单位是pt。
set label_size,20
除了使用pt单位,也可以使用Angstrom 单位,如设置字体大小为 1.2 Angstrom。
set label_size,-1.2
删除object
delete objectName
delete all
保存object
cmd.save(filename[, selection[, state[, format]]])
save file [,(selection) [,state [,format]] ]
PyMOL>save 01.pdb, ONLYprotein,
Save: wrote “01.pdb”.
PyMOL>cmd.save(“0.pdb”,”ONLYprotein”)
掩藏object-disable
disable objectName
掩藏representation
hide stick hide cartoon
蛋白的展现形式(show,as)
命令如下:
as cartoon
show cartoon,objectname
as stick,all
as stick,objectname
as stick,selection express
as stick,resn arg
设置双键的显示形式 valence
set valence, 0 #关闭双键显示
set valence, 1 #开启双键显示
设置stick的粗细
set stick_radius, 0.12
设置小球的大小 sphere_scale
set sphere_scale,0.5
重新计算二级结构(dss)
dss
选择中的关键词和缩略
- organic org. Non-polymer organic compounds (e.g. ligands, buffers)
- inorganic ino. Non-polymer inorganic atoms/ions
- solvent sol. Water molecules
- polymer pol. Protein or Nucleic Acid
- polymer.protein Protein (New in PyMOL 2.1)
- polymer.nucleic Nucleic Acid (New in PyMOL 2.1)
- guide Protein CA and nucleic acid C4*/C4’
- hetatm Atoms loaded from PDB HETATM records
- hydrogens h. Hydrogen atoms
- backbone bb. Polymer backbone atoms (new in PyMOL 1.6.1)
- sidechain sc. Polymer non-backbone atoms (new in PyMOL 1.6.1)
- metals Metal atoms (new in PyMOL 1.6.1)
- donors don. Hydrogen bond donor atoms
- acceptors acc. Hydrogen bond acceptor atoms
cartoon 模式
pymol 中内置了10种cartoon 模式:
- skip (-1) 不显示
- automatic (0) (use ss property) 默认模式
- loop (1)
- rectangle (2)
- oval (3)
- tube (4)
- arrow (5)
- dumbbell (6) (see also cartoon_fancy_helices)
- putty (7) (b-factor scaling)
- dash (8) (new in PyMOL 1.8.2)
在命令行窗口输入下面的命令对各种模式进行感受。
cartoon loop cartoon tube cartoon putty
cartoon helix 模式
在cartoon automaic 模式下 pymol 中内置了3种cartoon helix模式:
- 默认default模式
- Fancy Helices 模式 两端是尖尖
- Cylindrical Helices 模式 圆柱体
开启 Fancy 模式
set cartoon_fancy_helices, 1
开启 cylindrcal 模式
set cartoon_cylindrical_helices, 1
设置圆柱的粗细:
set cartoon_helix_radius,0.9
set cartoon_helix_radius,2.5
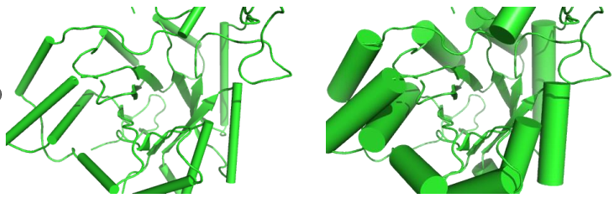
启用默认 default 模式
set cartoon_fancy_helices, 0
set cartoon_cylindrical_helices, 0

定义fancy 和default模式下的α-helix
命令:
set cartoon_oval_length , 0.8 # default is 1.20
set cartoon_oval_width , 0.2 # default is 0.25
着色(color)
color red,object name
选择,残基(select)
选择所有的碳原子: select catoms, elem C
选择蛋白:select protein, (byres polymer & name CA)
选择核酸: select nucleic, (byres polymer & name P)
选择RNA: select rna, (byres polymer & name O2’)
选择DNA: select dna, (nucleic & !rna)
实战:
select L,(chain L and polymer)
删除配体分子5A以外的水分子
remove resn hoh and (resi 307 gap 5)
叠合两个object(super)
参考: https://pymolwiki.org/index.php/Super
自动进行全局叠合
super 会把两个object进行叠合,和align的区别主要是
- super 主要是基于结构进行叠合;
- align 主要是基于序列进行叠合。
当两个蛋白的序列相似度较低的时候,使用super叠合的效果比align叠合的效果好。
super p1, p2
指定叠合部分
super p1 & alt A+'', p2 & alt B+''
实战
super obj03 & resi 2-191 & chain K, obj04 & resi 2-191 & chain C
注:在PyMOL中,还有多种结构叠合的方法,可通过“cealign”、“align”、“super”、“pairfit”、“fit”等命令实现。常用的就是align和super
设置二级结构的颜色
PyMOL中内置了3种二级结构着色策略。
helix sheet loop
red yellow green (策略1)
cyan magenta salmon (策略2)
cyan red magenta (策略3)
除了上述内置的着色策略,我们还可以自定义二级结构的颜色。
方法一:
选定二级结构对应的氨基酸残基,然后设置颜色。
方法二(推荐):
color red, ss h
color yellow, ss s
color green, ss l+''
推荐一套配色策略,感觉比内置的好看一点。(实测不好看)
PyMOL>color yellow,ss s
PyMOL>color grey,ss l+''
PyMOL>color teal,ss h
命令行设置不同的光照模式
鼠标模式点击 Plugin->light settings进行设置。
命令行模式
set ambient,0.4
set reflect,0.5
设置颜色 (set xx_color)
设置背景颜色
bg_color white