大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
痤疮表皮杆菌(Cutibacterium acnes,C.acnes)是一种革兰氏阳性细菌,是人类皮肤微生物组成员。尽管是最丰富的皮肤共生体,但某些成员与常见的炎症性疾病(如痤疮)有关。各种C.acnes分支的完整基因组序列可以鉴定推定的甲基转移酶,其中一些可能属于保护入侵DNA宿主的限制性修饰(restriction-modification,R-M)系统。然而,关于这些系统是否在不同的C.acnes菌株中发挥作用,目前知之甚少。
2022年3月18日,西班牙庞培法布拉大学Marc Güell团队在《PLOS PATHOGENS》杂志发表题为“Engineering selectivity of Cutibacterium acnes phages by epigenetic imprinting”的研究论文,该研究以痤疮表皮杆菌(C. acnes)为对象,通过Oxford Nanopore Technologies(ONT)测序分析了六种代表性痤疮C.acnes菌株的甲基化水平。揭示了C. acnes KPA171202的R-M系统IIIB影响PAD20噬菌体感染特性并保护细菌免于裂解。

标题:Engineering selectivity of Cutibacterium acnes phages by epigenetic imprinting(痤疮表皮杆菌噬菌体表观遗传印迹的工程选择性研究)
时间:2022-03-18
期刊:PLOS PATHOGENS
影响因子:IF 6.7
技术平台:三代原核甲基化ONT测序、RNA-seq
研究摘要:
细菌有多种对噬菌体感染的防御机制。其中一种防御机制是对自身DNA进行修饰,以区分自体或非自体。对皮肤细菌C. acnes菌株将修饰引入自己基因组的能力进行分析,发现其中一种菌株能够做到这一点。分析结果表明,自身DNA无修饰的细菌会被噬菌体强感染,而修饰后的细菌对感染具有免疫力。皮肤重某些痤疮C. acnes菌株的成分高度多样化,某些菌株的丰度变化会导致疾病。例如,抗生素的使用通常会杀死细菌,无论其菌株类型如何,因此不能保护人类与其微生物组的共生关系。因此本研究检测是否可以通过使噬菌体仅针对没有能力修饰自身DNA的菌株来选择性地杀死特定的菌株亚群。结果表明,随着时间的推移,可以调控菌株组成,挽救具有有益性状的菌株,同时减少引发痤疮的菌株。
为了研究假定的R-M活性及其在宿主保护机制中的相关性,本研究通过三代原核甲基化ONT测序(ONT)测序分析了六种代表性痤疮杆菌菌株的甲基化图谱。在菌株KPA171202中检测到在确定的DNA共有序列上6-甲基腺嘌呤(6mA)修饰,且该R-M系统的重组表达证实了其甲基化活性。而R-M敲除突变体验证了该菌株甲基化特性的缺失。此外还研究了C. acnes噬菌体(PAD20)杀死各种痤疮表皮杆菌(C. acnes)菌株的潜力,并将其特异性的增加与甲基化活性菌株感染后获得的噬菌体DNA甲基化联系起来。研究证明了在R-M缺失菌株中繁殖的噬菌体选择性地杀死R-M缺失痤疮敏感分支,而益生菌维持对噬菌体感染抗性。
研究结果
(1)C. acnes菌株中的限制性甲基化(Restriction-methylation,R-M)
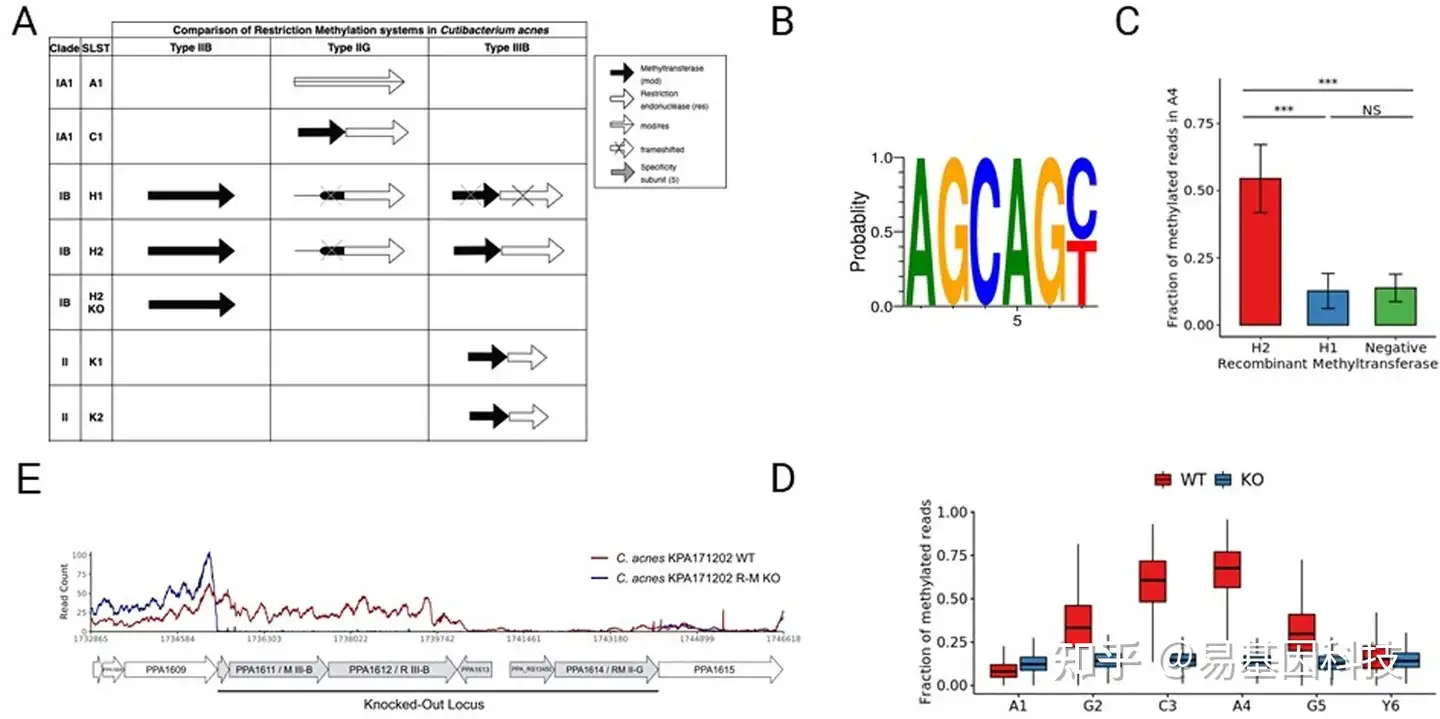
图1:C.acnes KPA171202(SLST H2)具有在AGCAGY序列甲基化motif的功能性R-M系统。
(A)使用REBASE中可获得的组装并对选定痤疮C.acnes菌株中潜在R-M系统的基因组比较,并比较菌株和分支水平。黑色表示MTase,而白色表示REase。
(B) 甲基化评分最高的reads甲基化motif的序列标志。
(C) 重组表达H1或H2 IIIB甲基转移酶后,pMW535质粒中检测的AGCAGY motif甲基化腺嘌呤中的甲基化reads片段。
(D) 野生型(红色)和IIIB型R-M系统敲除型(蓝色)KPA171202(H2)菌株的AGCAGY motif上的甲基化reads片段。
(E) C.acnes KPA171202野生型(SLST H2 WT)和C.acnes限制性甲基化敲除(SLST H2R-MKO)中限制性甲基化位点和周围环境的转录谱。
表达水平通过每个基因组位点的RNA-seq reads数计算(两次重复的平均值)。R-M IIG对应于PPA1614,R-M IIIB对应于PPA1161。
(2)代表性痤疮C. acnes菌株的De novo motif
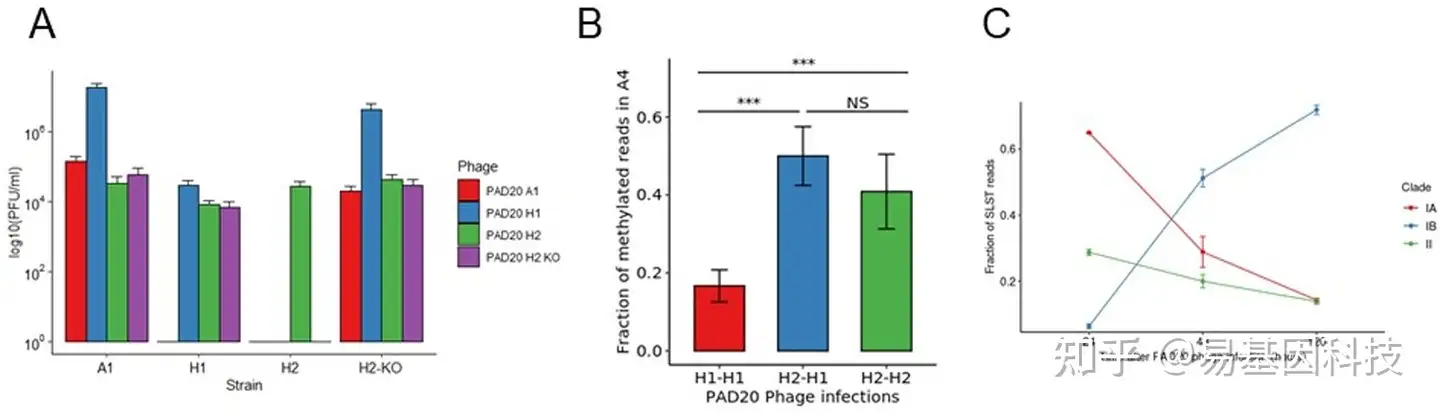
图2:C.acnes KPA171202的R-M系统IIIB影响PAD20噬菌体感染性状并保护细菌免受裂解。
(A)在SLST A1、H1、H2或H2 KO上繁殖的PAD20噬菌体的感染性状变化。
(B) PAD20噬菌体基因组中AGCAGY motif第4位甲基化腺嘌呤的甲基化reads片段。样品首先以噬菌体测序的感染菌株命名,其次以噬菌体先前繁殖的菌株命名(即H2-H1表示H1中产生的噬菌体用于感染H2,然后从H2中提取用于测序)。使用ANOVA分析测试显著性(***,p<0.001)。
(C)选择性杀灭整个微生物组样品在3个不同时间点感染PAD20后在SLST A1上传播。在持续PAD20感染24、48、120小时后采集样本。使用SLST方法扩增样品,并进行Illumina测序。与SLST等位基因的相似性用于将测序reads分类为分支IA、IB和II。Clade IA包含以下SLST类型:A1、A2、A3、A4、A7、A11、A13、A16、A17、A18、A21、A24、A27、A28、A29、A30、A31、A33、A34、A35、A38、A40、A42、A46、A47、A48、A49、A51、A52、C1、C5、F4、F6、F10、F15、F16、F17、F22、F24;Clade IB包含:H1、H2、H3、H4、H7、H8、H10、H11、H12;Clade II包含:K1、K2、K7、K9、K17、K19。
关于易基因三代原核甲基化纳米孔测序(ONT)
表观修饰不需要改变DNA序列便能实现对性状的改变,表观修饰的改变与基因功能乃至细胞状态、发育、衰老、疾病等存在重要的关联。在众多的表观遗传修饰中,最为重要且研究最为广泛的修饰之一是DNA甲基化。
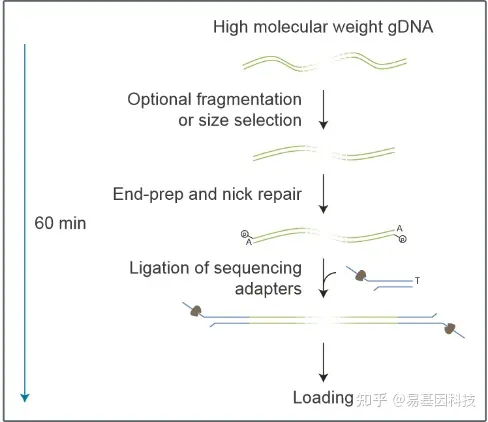
由于PCR的扩增过程会消除碱基修饰信息,通过传统测序技术对其进行检测需要使用特殊的文库制备步骤,从而造成核酸损坏,最终导致测序读长序列非常短。
Nanopore测序是Oxford Nanopore Technologies(ONT)公司开发的基于纳米孔电信号的单分子实时测序技术,DNA双链在马达蛋白的作用下与锚定在生物膜上的八聚体纳米孔蛋白结合并解螺旋。由于生物膜两测存在电势差,解旋后的单链DNA以一定速率通过纳米孔。由于化学性质不同,不同碱基通过纳米孔时,会引起不同电信号的变化。通过对这些电信号变化进行检测和解析,完成序列的实时测定。Nanopore的碱基判读是依据其电流信号而产生,因此其判定过程比较复杂。目前Nanopore根据电流的大小及电流大小的变化情况,通过“递归神经网络(Recurrent Neural Network)”的复杂算法对碱基进行判读。
ONT全基因组甲基化测序原理示意图如下:
技术优势:
- 长读长:平均读长>10Kbp,最长可达2M左右,直接跨越重复序列、高度多态性区域等特殊区域;
- 直接测序:无需PCR扩增,可直接保留并检测DNA甲基化修饰;
- 全基因组覆盖:可同时检测全基因组范围单碱基的5mC与6mA修饰位点,并给出单碱基的甲基化水平;
- 性价比高:ONT测序成本相对于二代NGS测序技术大幅降低。
实验策略:
易基因提供全面的表观组学、微生物组学、转录组学等多组学测序方案,详询易基因:0755-28317900.

参考文献:
Knödlseder N, Nevot G, Fábrega MJ, Mir-Pedrol J, Sanvicente-García M, Campamà-Sanz N, Paetzold B, Lood R, Güell M. Engineering selectivity of Cutibacterium acnes phages by epigenetic imprinting. PLoS Pathog. 2022 Mar;18(3):e1010420.
相关阅读: