大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
双生病毒(Geminiviruses)是一种DNA植物病毒,可引起影响全球作物的高度破坏性疾病。在感染过程中,双生病毒调控细胞过程,抑制植物防御,并导致感染细胞的大量重编程,导致整个植物稳态重大变化。测序技术进步允许大规模同时分析病毒感染的多个方面,为植物-病毒互作的分子机制产生新的见解。然而尚未对双生病毒感染期间宿主转录组、sRNA谱和甲基化组的变化进行综合研究。
2023年12月18日,西班牙亚热带和地中海园艺研究所Araceli G. Castillo团队在《BMC Plant Biology》杂志发表题为“Transcriptional and epigenetic changes during tomato yellow leaf curl virus infection in tomato”的研究论文,该研究使用短读长测序,通过全基因组重亚硫酸盐测序(WGBS)分析了TYLCV感染的番茄(Solanum lycopersicum)植株在四个时间点(TYLCV感染接种后第2、7、14和21天)(days post-inoculation, dpi)的mRNA和sRNA转录组变化,以及14dpi的番茄甲基化组变化。这些数据的分析和整合研究了TYLCV感染期间宿主基因表达的变化及其对sRNA调控(miRNA和阶段性次级小干扰RNA(phasiRNA))和DNA甲基化的依赖性。本研究首次对双生病毒感染后番茄mRNA、sRNA转录组和甲基化组变化进行了全面分析。

标题:Transcriptional and epigenetic changes during tomato yellow leaf curl virus infection in tomato(番茄黄化曲叶病毒感染过程中的转录和表观遗传学变化)
时间:2023-12-18
期刊:BMC Plant Biology
影响因子:5.3
技术平台:WGBS、RNA-seq、sRNA-seq等
研究摘要:
本研究使用时间节点方法,旨在分析番茄对双生病毒番茄黄化曲叶病毒(tomato yellow leaf curl virus,TYLCV)感染的基因调控。研究结果揭示番茄在TYLCV感染后经历了大量的转录和转录后变化,并鉴定出主要变化的调控通路。尽管诱导了主要的植物防御相关过程、基因沉默和免疫反应,但无法阻止感染建立。将细胞外和细胞内免疫受体鉴定为失调的microRNA(miRNA)靶标,并为也产生阶段性次级小干扰RNA(phasiRNA)受体建立了网络。此外感染后14天,在症状普遍出现的时间点,病毒DNA量达到最大水平,但番茄甲基化组没有显著的全基因组变化,但能够鉴定出差异甲基化区域,这些区域可能参与一些差异表达基因的转录调控。
本研究对TYLCV感染过程中番茄转录、转录后和表观遗传水平变化进行了全面可靠的研究。所产生的基因组信息对于了解番茄TYLCV感染引起的遗传、分子和生理变化具有重要意义。
研究结果:
(1)番茄TYLCV感染过程中的转录变化
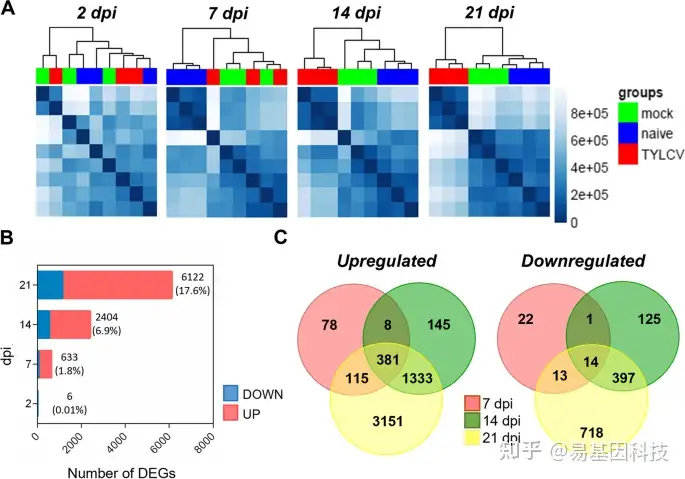
图1:番茄植株TYLCV感染期间的转录变化。
A. 在2dpi、7dpi、14dpi和21 dpi的naïve(蓝色)、mock(绿色)和TYLCV感染样品(红色)中番茄基因表达谱在分层聚类的热图中显示。右侧的蓝色条表示归一化reads数。
B. 堆叠条形图显示TYLCV感染与mock样品在2dpi、7dpi、14dpi和21 dpi的上调DEG(红色)和下调DEG(蓝色)数量。
C. 维恩图显示2dpi、7dpi、14dpi和21 dpi共有和特异性上调DEG和下调DEG。
B和C,只显示FDR矫正p值≤0.05和≥1.5倍诱导或≤0.75倍抑制的的DEG
(2)番茄DEGs对TYLCV感染的功能富集
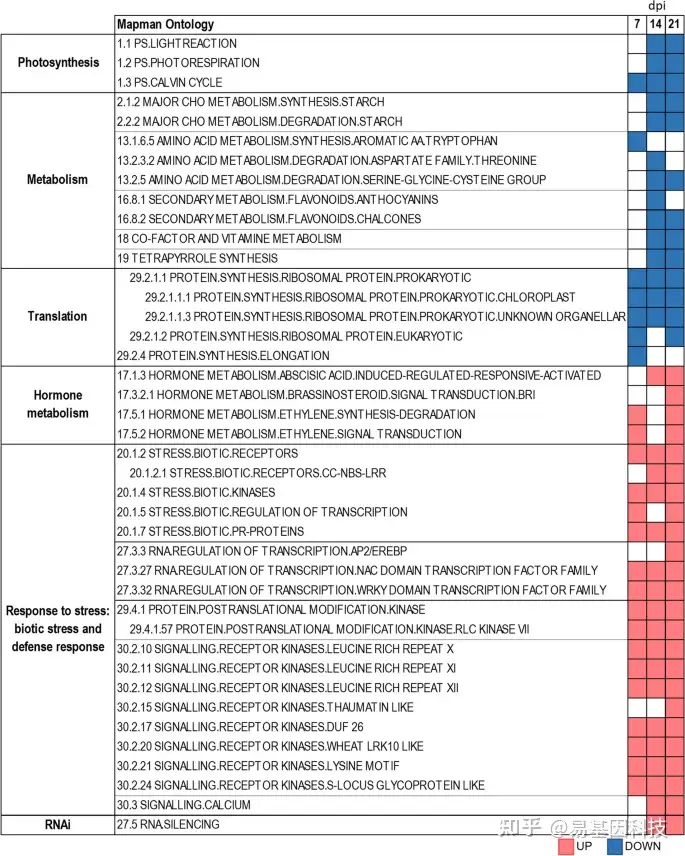
图2:番茄对TYLCV感染响应的生物过程转录失调。使用基因组富集分析(GSEA)计算方法和MapMan本体论作为基因组来源,用于鉴定在7dpi、14dpi和21 dpi显著富集上调(红色)或下调基因(蓝色)的生物过程。没有颜色表示没有统计学上的显著富集(FDR校正p值 ≤ 0.05)
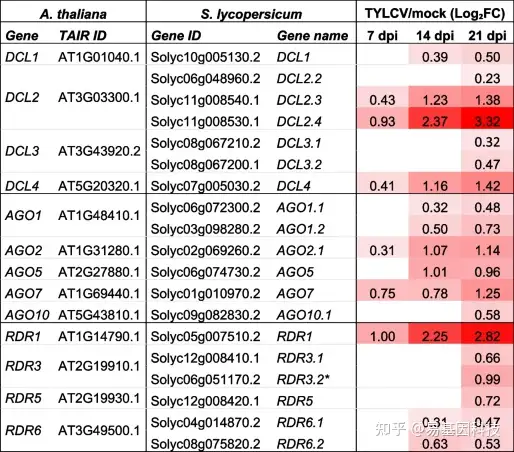
表1:TYLCV感染期间参与转录后基因沉默的差异表达番茄基因
(3)TYLCV感染过程中转录变化动态
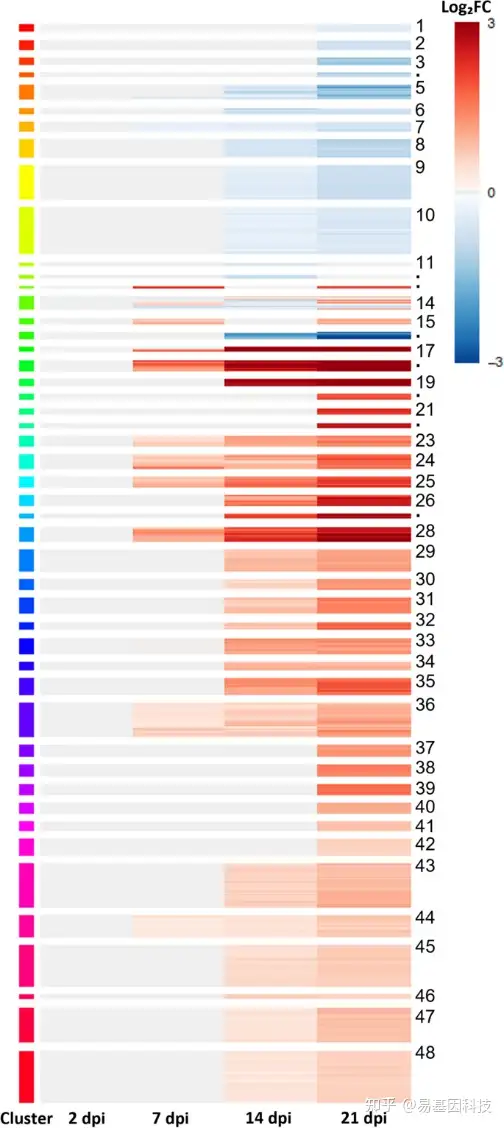
图3:番茄TYLCV感染期间转录调控动态。SplineCluster算法对番茄DEG进行聚类响应TYLCV感染(2,7,14和21 dpi)。
(4)TYLCV感染期间的番茄小RNA谱
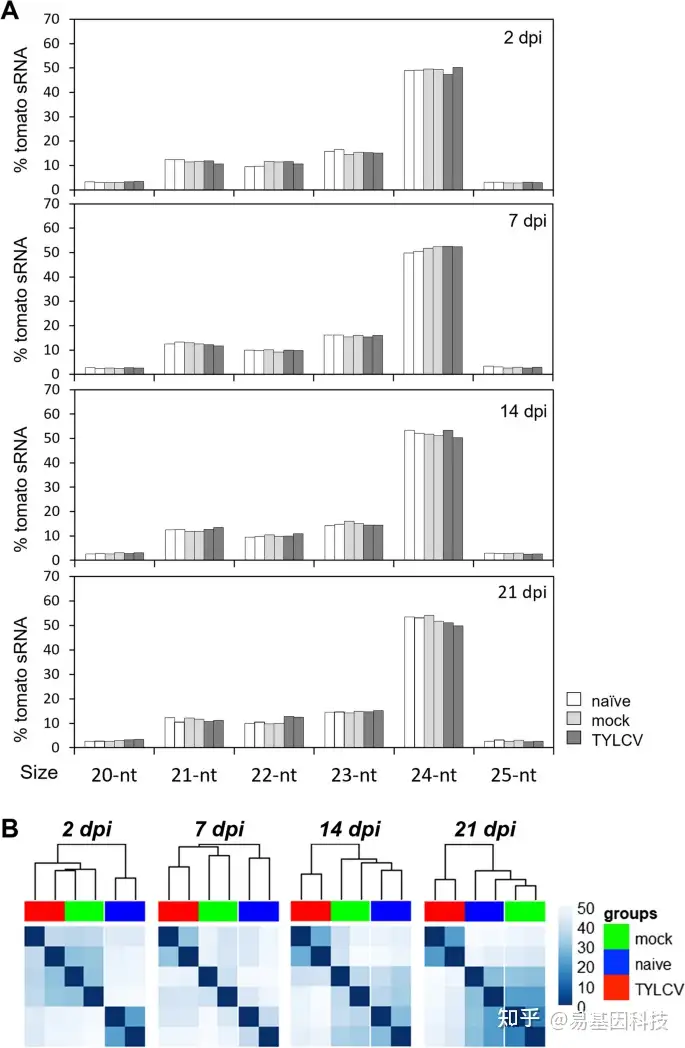
图4:TYLCV感染过程中的番茄sRNA谱。
A. 在2、7、14和21 dpi的naïve、mock和TYLCV感染的番茄样品中,大小类别20-25 nt sRNA reads比对到番茄基因组(18-26 nt)的总sRNA reads百分比。每条对应于一个生物学重复。
B. 在2、7、14和21 dpi的naïve(蓝色)、mock(绿色)和TYLCV感染(红色)样品中番茄sRNA的表达用分层聚类热图显示。右侧的蓝色条表示归一化CPM
(5)TYLCV感染过程中番茄microRNA表达谱的变化

表2:sRNA-seq数据集中鉴定的番茄miRNA分类
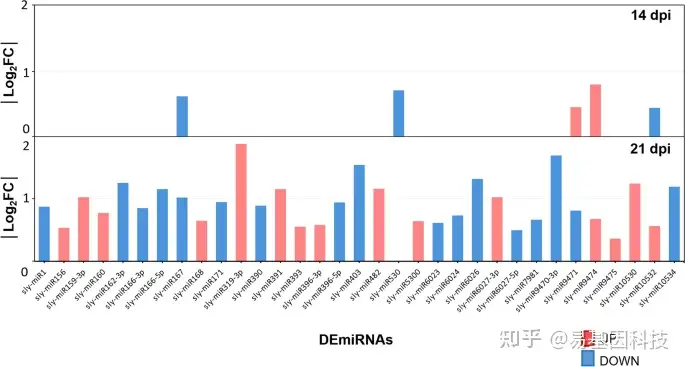
图5:番茄TYLCV感染过程中miRNA表达变化。与14和21 dpi的mock感染相比,TYLCV感染样品中的miRNA表达变化(| log2FC |,log2FC的绝对值)。上调miRNA以红色显示,下调miRNA以蓝色显示
(6)TYLCV感染过程中miRNA靶基因的转录后调控
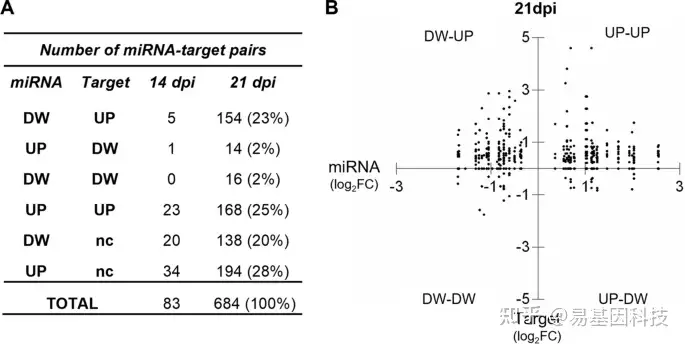
图6:在TYLCV感染的番茄中,demiRNA及其预测的靶基因表达水平。
A、 基于表达水平对14和21 dpi推测miRNA靶基因分类。UP:上调,DW:下调,nc(no change):靶基因无差异表达。
B、 DEmiRNA(x轴)及其靶基因(y轴)在21 dpi时的表达水平(log2FC为TYLCV/mock比)
(7)TYLCV感染过程中phasiRNA及其预测靶基因和PHAS位点的比较表达
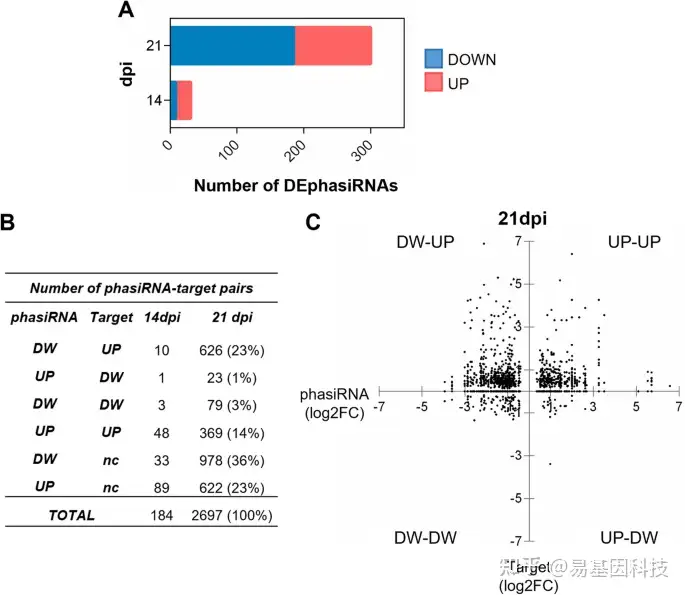
图7:在TYLCV感染的番茄植株中,DEphasiRNA及其预测的靶基因表达水平。
A、 堆叠条形图显示了TYLCV感染与mock样品在14dpi和21 dpi的DEphasiRNA数量、上调(红色)和下调(蓝色)的phasiRNA。
B、 基于miRNA和靶基因表达水平,在14和21 dpi时预测phasiRNA-target对数。
C、 DEphasiRNAs(x轴)及其靶基因(y轴)在21 dpi时的表达水平(log2FC为TYLCV/mock比)
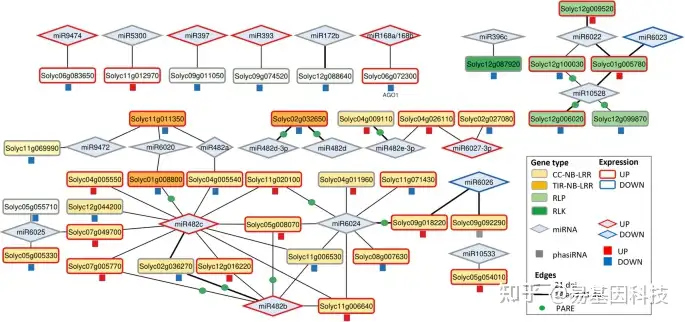
图8:番茄miRNA PHAS位点phasiRNA网络对TYLCV感染21 dpi的影响。miRNA(21和22 nt,菱形)的网络表示,其在21 dpi时从其靶PHAS位点转录本(矩形)触发dephasirna(正方形)形成。每个几何形状都被一条彩色线包围,表示它们的差异表达模式:红色表示诱导,蓝色表示抑制,灰色表示无差异表达。

表3:在14和21 dpi产生DEphasiRNA的番茄PHAS位点及其miRNA触发因子
(8)番茄基因组DNA甲基化与TYLCV感染的关系
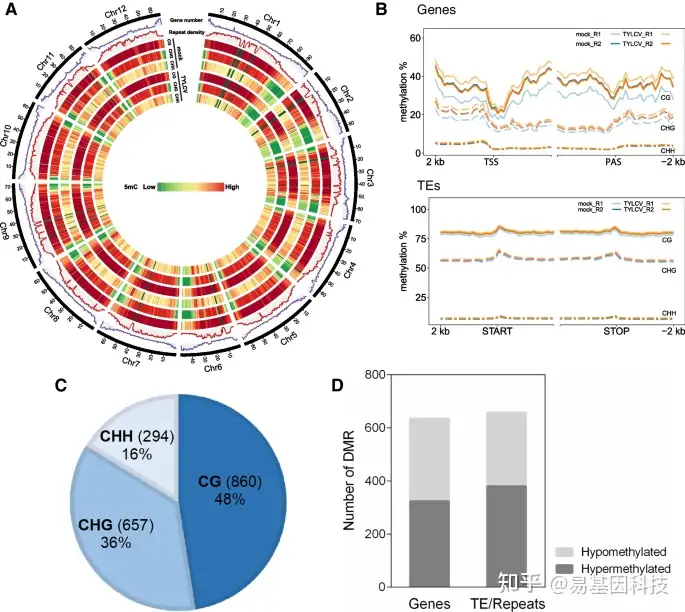
图9:TYLCV感染后的番茄表观基因组图谱。
A、 TYLCV感染和mock样品在不同环境(CG、CHG和CHH)染色体上5-甲基胞嘧啶的分布图。染色体名称显示在外边缘。
B、 mock和感染植株的两个生物重复(R1和R2)的基因甲基化率和TEs/重复序列。转录起始位点(TSS)和多聚腺苷酸化位点(PAS)。
C、 每个甲基化环境(CG、CHG和CHH)上DMR(TYLCV/mock)的总数和百分比。
D、 基因和TEs/重复序列中低甲基化和高甲基化区域的数量。
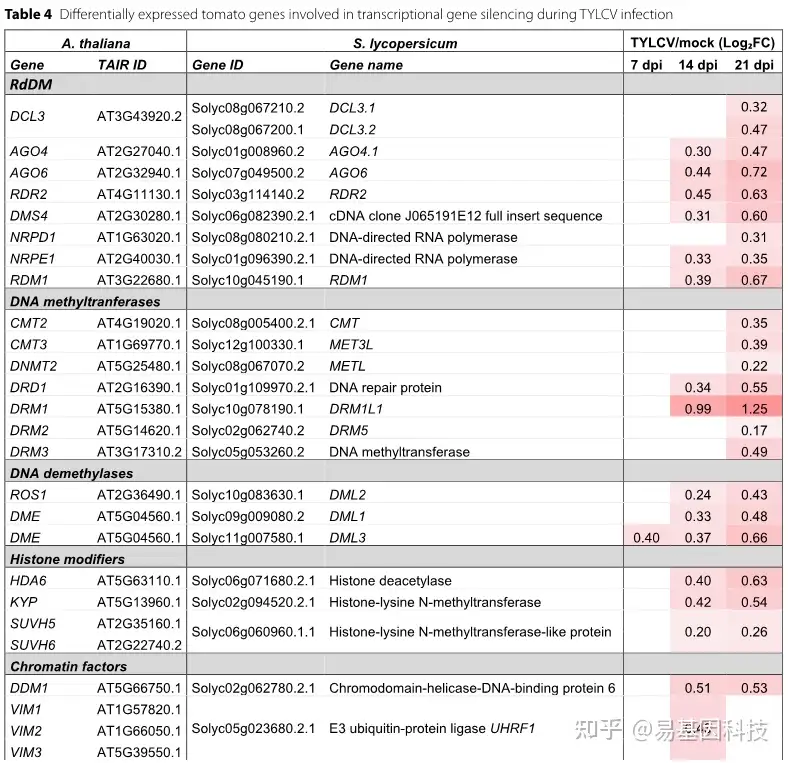
表4:TYLCV感染过程中番茄转录基因沉默的差异表达基因
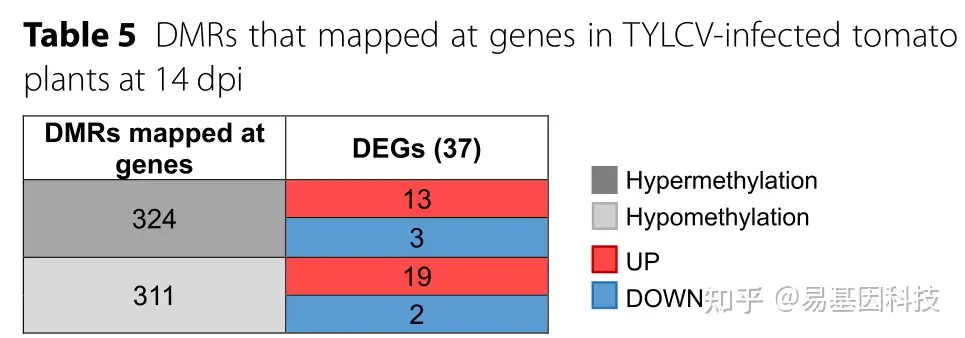
表5:TYLCV感染的番茄植株中14 dpi比对基因的DMRs
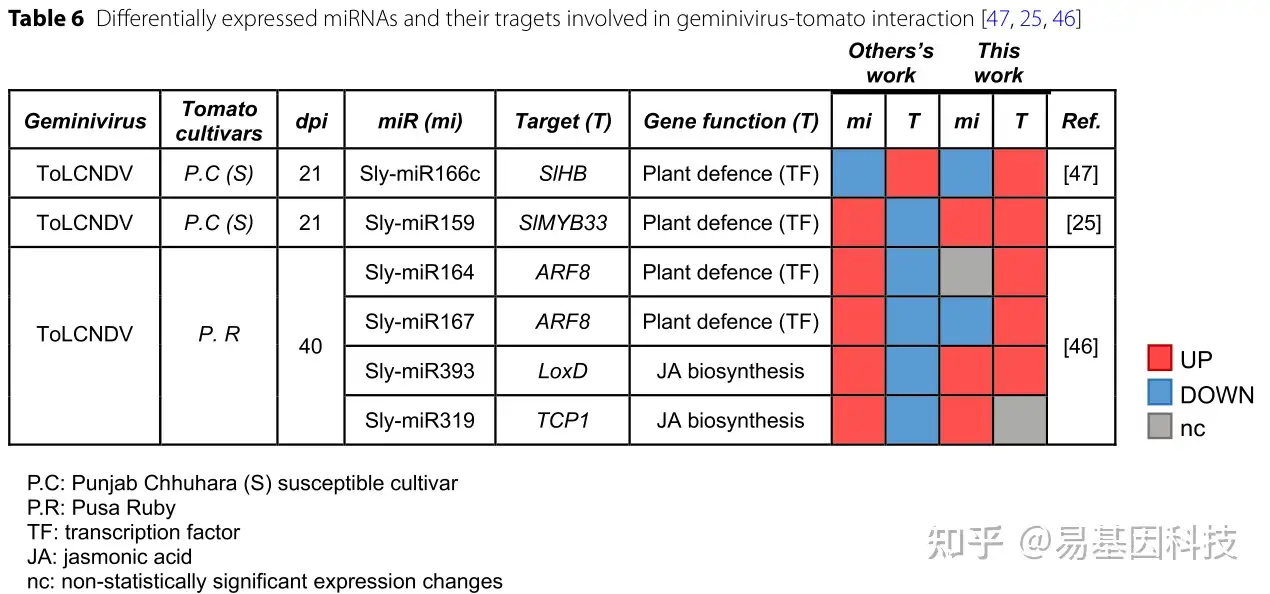
表6:双生病毒-番茄互作中差异表达的miRNA及其转录因子
研究结论
本研究结果表明,TYLCV诱导番茄发生剧烈的转录变化,这种变化在整个感染过程中增加,且在初始阶段依赖于病毒DNA量,但一旦病毒滴度达到其最大水平,就不会发生。属于植物两种主要防御机制的基因,即基因沉默和免疫应答,在症状建立之前被诱导。在整个感染过程中,这些基因的诱导强度和/或数量增加。另一方面,番茄miRNA和phasiRNA失调不依赖于病毒DNA,病毒转录本或病毒sRNA量,并且在蛋白质编码基因显著失调后出现显著诱导或抑制。对差异表达miRNA的分析表明,它们主要靶向参与生长素反应,基因沉默或编码转录因子和免疫受体(RLK和NLR)的基因,其中许多是产生失调的phasiRNA的PHAS位点。当考虑miRNA-target对时,miRNA与其靶标之间的预期反比关系并不一致。来自DNA甲基化机制的大多数主要基因表达在TYLCV感染期间被轻微诱导,并且已经鉴定出可能参与一些差异表达基因转录调控的差异甲基化区域。综上所述,这项研究提供了对TYLCV和番茄相互作用的复杂互作的见解,并代表了对双生病毒感染期间番茄mRNA转录组、sRNA谱和甲基化组变化的首次综合和全面分析。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因科技提供全面的DNA甲基化研究整体解决方案,详询易基因:0755-28317900。
参考文献:Romero-Rodríguez B, Petek M, Jiao C, Križnik M, Zagorščak M, Fei Z, Bejarano ER, Gruden K, Castillo AG. Transcriptional and epigenetic changes during tomato yellow leaf curl virus infection in tomato. BMC Plant Biol. 2023 Dec 18;23(1):651. pii: 10.1186/s12870-023-04534-y. doi: 10.1186/s12870-023-04534-y. PubMed PMID: 38110861.
相关阅读:
年终盘点 | 易基因2023年度DNA甲基化研究项目文章精选
深度综述:人早期胚胎发育的表观遗传调控(染色质重塑+组蛋白修饰+DNA甲基化)