今天简单回顾下中国农业大学王向峰教授团队2023年上半年发表在Trends in Plant Sicence的综述文章。该文阐释系统全面,值得赏析。

摘要
一些从基础研究中获得的生物学知识将被应用于实际的植物育种中。为了弥合基础研究与育种实践之间的差距,机器学习(ML)具有巨大的潜力,将生物学知识和组学数据转化为精准设计的植物育种。在这里,我们回顾了在植物中用于多组学分析的机器学习,包括数据降维、基因调控网络的推断,以及基因发现和优先排序。这些应用将有助于理解性状调控机制,并识别可能适用于知识驱动分子设计育种的目标基因。我们还强调了深度学习在植物表型组学中的应用,以及机器学习在基因组选择辅助育种中的应用,例如各种机器学习算法,它们模拟了基因型(基因)、表型(性状)和环境之间的相关性,最终实现数据驱动的基因组设计育种。
将生物数据和知识转化为植物的精准设计育种
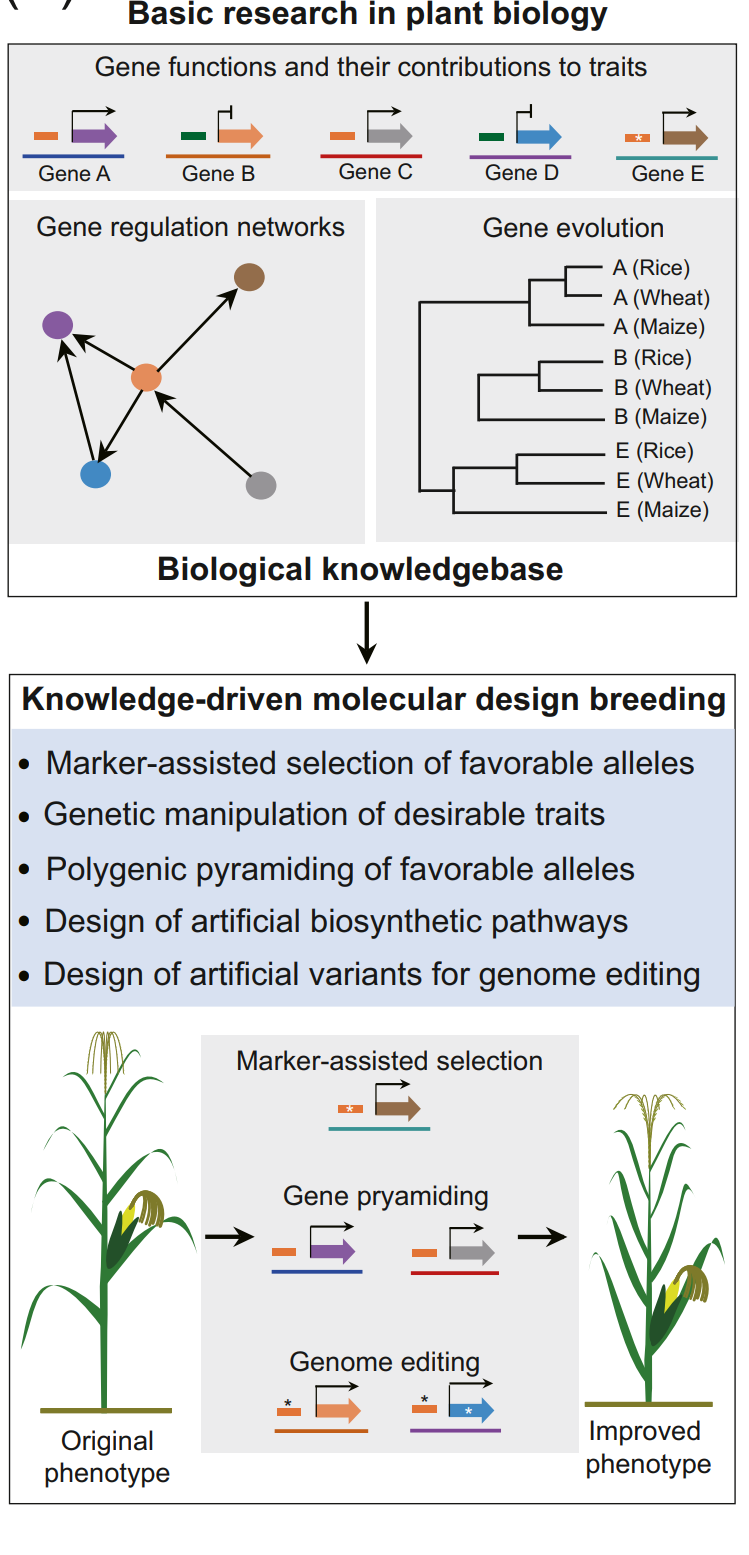
从植物生物学的基础研究中理解基因功能和调控机制。一个生物学知识库将通过多种技术促进知识驱动的分子设计育种。图中展示了通过标记辅助选择、多基因的优良等位基因堆叠以及基因组编辑,改善玉米品种性状的一个例子。
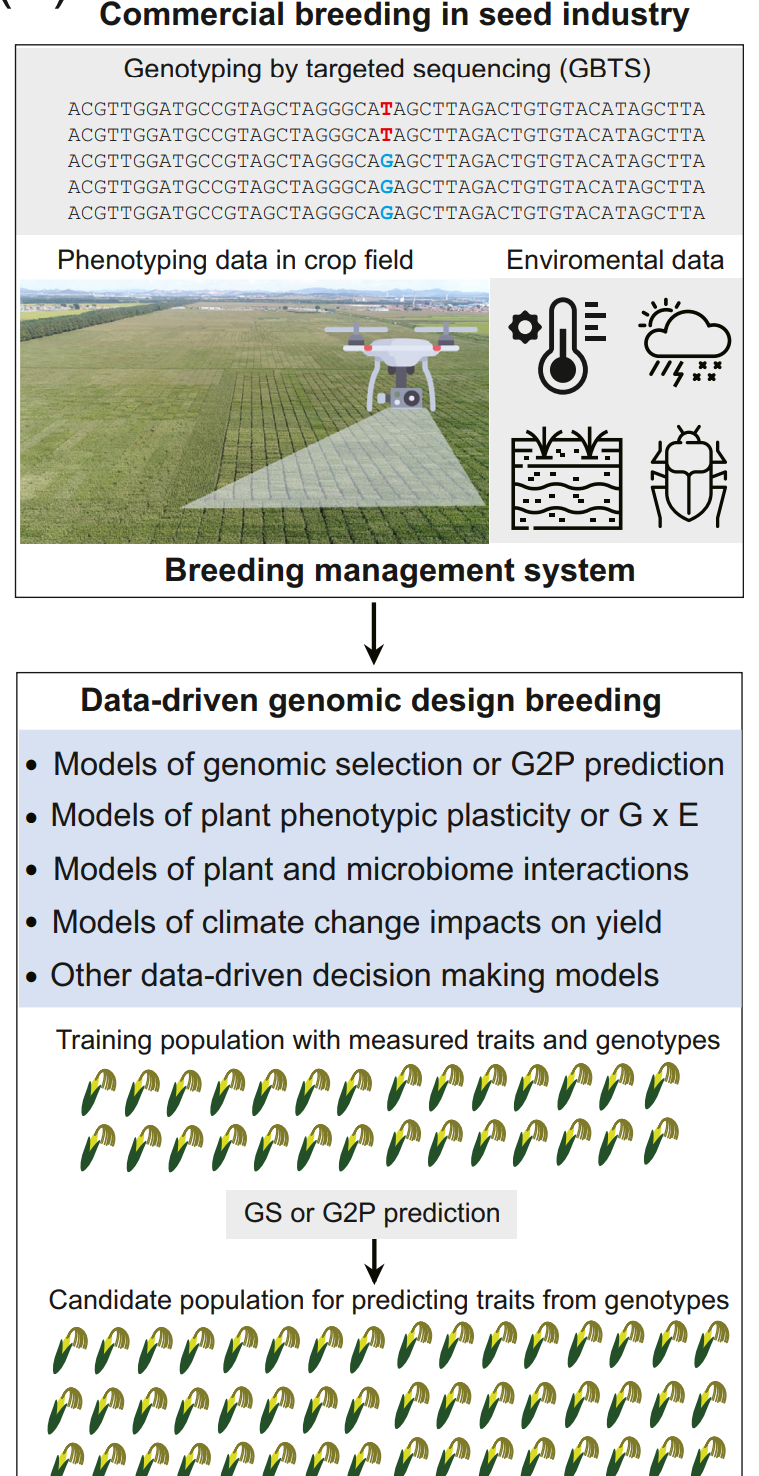
从商业育种项目中积累的基因型、表型和环境数据将通过构建各种决策模型促进数据驱动的基因组设计育种。图中展示了使用基因组选择(GS)模型从基因型预测表型的一个例子。
机器学习(ML)在植物生物学中的应用
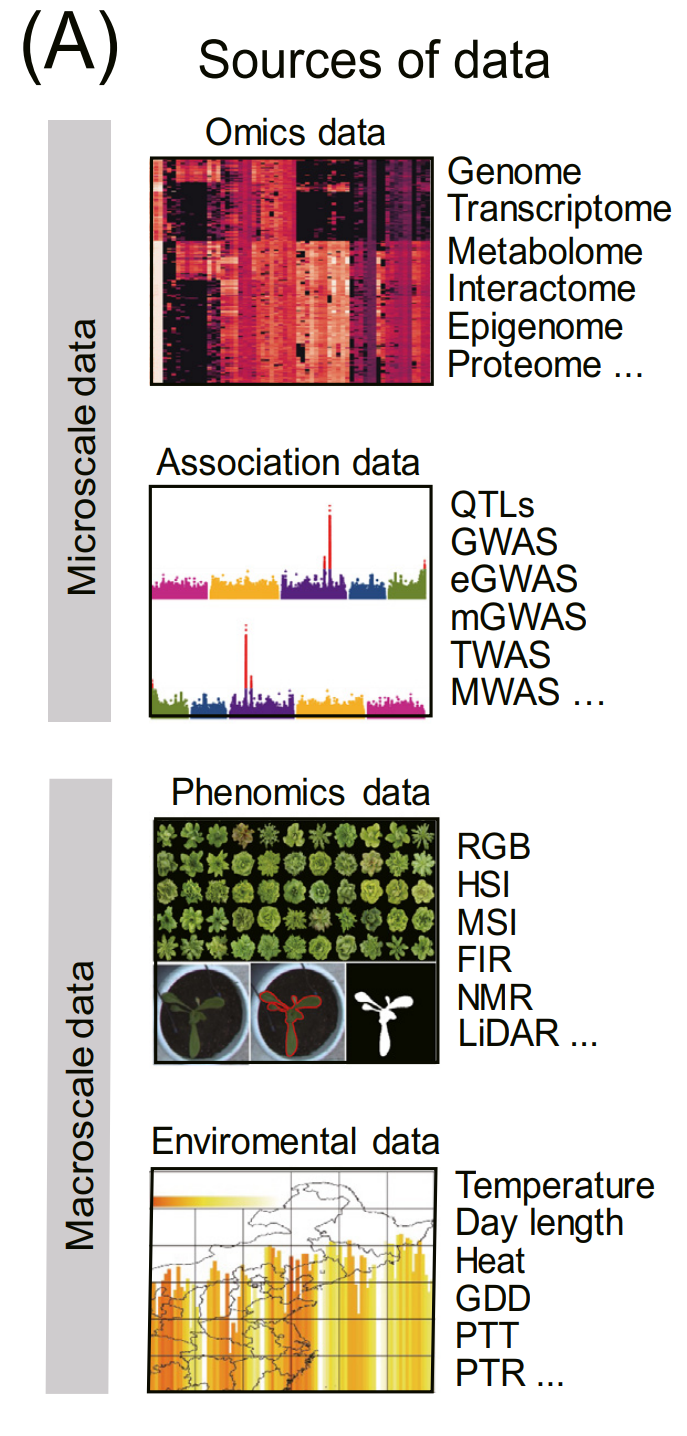
从微观到宏观测量产生的各种生物数据类型。
在生物学中应用的监督学习和无监督学习方法。四个框表示了使用LightGBM进行基因优先排序的特征重要性分析、图卷积网络(GCN)模型整合多组学数据、使用基因型对玉米品系进行分类的OPTICS算法,以及使用t-SNE可视化玉米种群结构的应用。
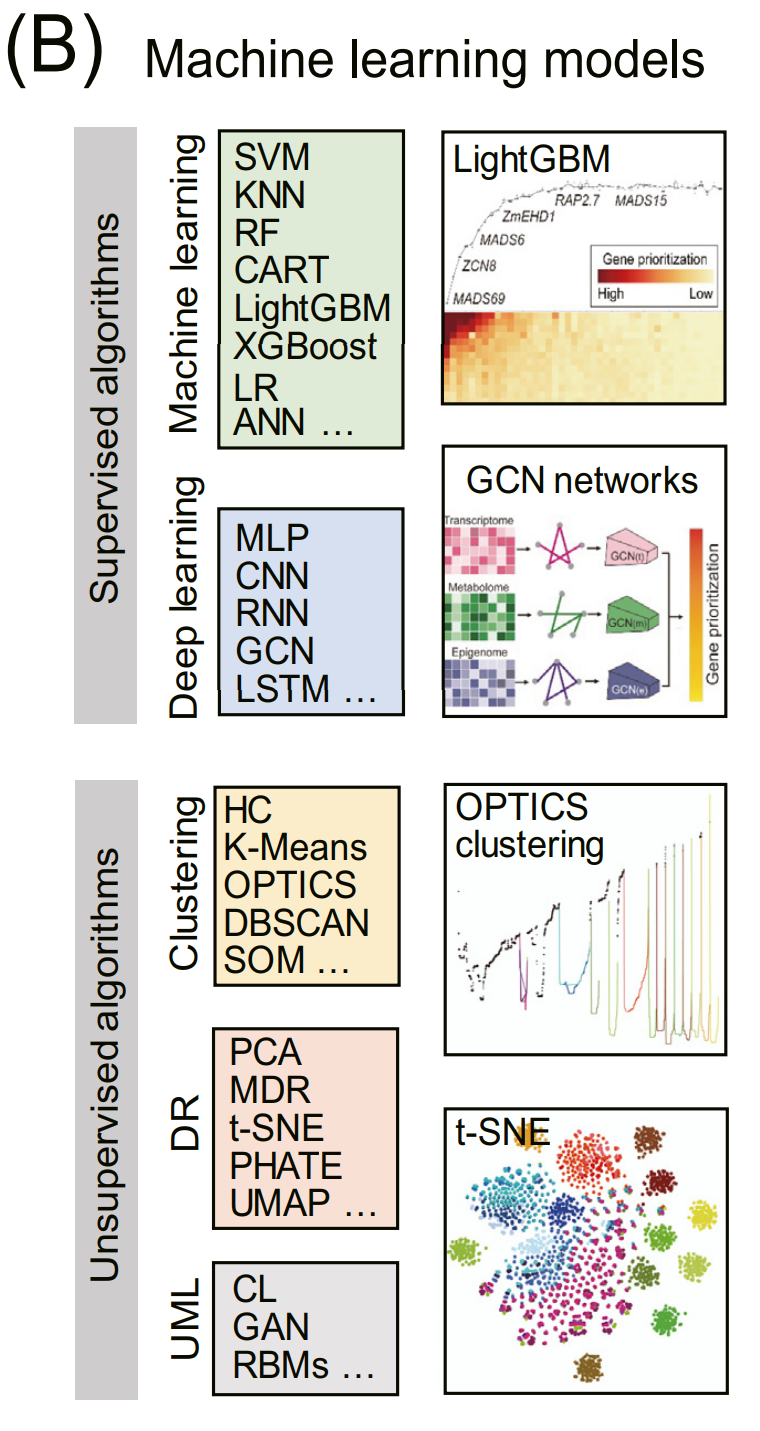
缩写词:ANN,人工神经网络;CART,分类和回归树;CL,对比学习;CNN,卷积神经网络;DBSCAN,基于密度的带有噪声的应用空间聚类;eGWAS,表达基因组关联研究;FIR,远红外;GAN,生成对抗网络;GCN,图卷积网络;GDD,生长度日;GWAS,全基因组关联研究;HC,层次聚类;HIS,高光谱成像;KNN,K最近邻算法;LiDAR,光检测和测距;GBM,轻梯度提升机;LR,逻辑回归;LSTM,长短期记忆;MDR,多因素维度缩减;mGWAS,代谢组基因组关联研究;MLP,多层感知器;MSI,多光谱成像;MWAS,代谢组宽关联研究;NMR,核磁共振;OPTICS,用于识别聚类结构的点排序;PCA,主成分分析;PHATE,基于热扩散亲和力轨迹嵌入的潜力;PTR,光热比;PTT,光热时间;QTLs,数量性状位点;RBMs,限制性玻尔兹曼机;RF,随机森林;RGB,红绿蓝通道摄像头;RNN,循环神经网络;scRNA-seq,单细胞RNA测序;SOM,自组织映射;SVM,支持向量机;t-SNE,t分布随机邻居嵌入;TWAS,转录组宽关联研究;UMAP,统一流形近似和投影;UML,无监督机器学习;XGBoost,极端梯度提升机。
机器学习在生物学应用的例子。

案例研究:多组学数据关联研究(MODAS)中的数据降维(DR)
通过对种质资源参考群体进行多组学分析,可以大大提高寻找致病基因的映射分辨率。然而,多组学数据集通常具有高维性。对包含数百个样本的群体进行全基因组重测序可能会生成数千万个SNP。单个转录组每个样本包含数万个基因的表达数据。如果在单细胞水平上生成,样本数量将乘以数千个细胞计数。如果包括多种条件,数据维度将进一步呈指数级扩展。因此,"维度诅咒"不可避免。为了解决这个问题,MODAS(多组学数据关联研究工具)利用多种无监督学习技术加速群体规模的多组学分析。
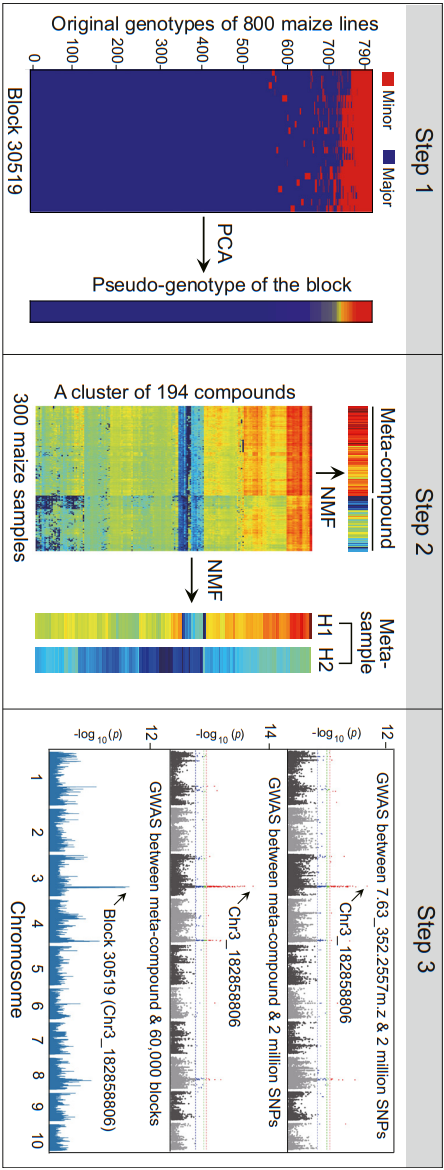
第一步,对基因型数据进行降维:以玉米群体为例,MODAS首先使用Jaccard指数计算任意两个SNP的基因型相似度,然后使用DBSCAN算法将在基因型上具有高相似性的SNP聚类为一个基因组块。接着,对每个聚类的SNP块应用PCA(主成分分析),生成一个伪基因型索引文件。该文件包含约60,000个基因组块,作为一个高度简化的变异图谱,代表玉米原始200万个SNP的基因型。
第二步,对组学数据进行降维:以代谢组数据为例,一个代谢组数据集大约包含30,000种化合物。然而,数据中相当一部分是冗余和噪声,在进行关联分析之前必须移除。MODAS首先使用互信息技术对表现出跨样本相似模式的冗余化合物进行聚类,然后在每个聚类内使用NMF(非负矩阵分解)算法进行DR。NMF将化合物×样本矩阵映射到一个“元化合物”维度和一个“元样本”维度。元化合物对样本的权重可以将300个玉米样本分类为两个主要的单倍型(H1和H2)群体,这与基因组块30519一致。
第三步,关联分析:元化合物的权重可以被视为表型特征,用于进行GWAS,不论是使用200万个SNP的基因型还是60,000个块的伪基因型。两种方法都可以映射到相同的QTL,并识别出峰值SNP Chr3_182858806。与194种化合物和200万个SNP之间的GWAS相比,元化合物和伪基因型之间的关联分析可以将时间从45.6小时减少到仅0.63分钟,并且产生相同的结果。这种策略适用于任何类型的组学数据,用于群体规模的基因挖掘。
亮点总结
为了弥合基础研究与植物育种实践之间的差距,机器学习具有巨大的潜力来整合生物学知识和组学数据,最终实现精准设计的植物育种。
目前,机器学习在植物研究和育种中的应用包括数据降维、基因调控网络的推断、基因发现和优先排序、植物表型组学分析,以及植物表型的基因组预测。
高维生物学指的是从宏观到微观层面生物数据的整合与分析,提高了可用于知识驱动分子设计育种的性状关联基因的机会。
在大数据时代,机器学习能够建模育种实践中收集的基因型、表型和环境数据之间的复杂关系,以实现数据驱动的基因组设计育种。