大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
胎儿肠道经历巨大的扩张和重塑,在发育过程中形成初级绒毛和连续的绒毛间隙。绒毛形成后,相同潜能的上皮祖细胞(equipotent epithelial progenitors)产生功能明确的成体干细胞(adult stem cells,ASC)。这些细胞主要存在于肠隐窝底部,并在整个生命过程中负责上皮补充。尽管从胎儿肠道到成年肠道转换过程中的组织学和形态学变化已得到表征,但这一过程在分子水平上如何协调,在很大程度上仍然未知。
2023年7月12日,丹麦哥本哈根大学Kim B. Jensen和Albin Sandelin团队在Science子刊《Science Advances》上发表题为“Transcriptional and epigenomic profiling identifies YAP signaling as a key regulator of intestinal epithelium maturation”的研究论文,该研究基于类器官培养物来研究上皮成熟细胞内在分子调控因子,通过WGBS、ATAC-seq、RNA-seq、Hi-C等转录组和表观基因组多组学分析将YAP信号传导鉴定为肠上皮成熟的关键调节因子。

标题:Transcriptional and epigenomic profiling identifies YAP signaling as a key regulator of intestinal epithelium maturation(转录和表观基因组分析将YAP信号确定为肠上皮成熟的关键调节因子)
时间:2023-07-12
期刊:Science Advances
影响因子:13.6 / 1区
技术平台:ATAC-seq、WGBS、单细胞RNA-seq、Hi-C等
研究摘要:
在肠道器官发生过程中,相同潜能的上皮祖细胞成熟为表型独特的干细胞,负责组织的终身维持。虽然与转变相关的形态学变化已得到很好的表征,但支撑成熟过程的分子机制尚未完全了解。本研究利用肠道类器官培养物来分析胎儿和成年肠道上皮细胞两种发育细胞状态的RNA表达、染色质可及性、DNA甲基化和三维(3D)染色质构象的转录组和表观基因组图谱。分析结果揭示两种细胞状态在RNA基因表达和增强子活性方面的显著差异,且两种细胞状态之间伴随着3D组织、染色质可及性、DNA甲基化的局部变化。通过综合分析鉴定出持续的Yes-Associated Protein (YAP)转录活性是未成熟胎儿状态的主要调控者。YAP相关转录网络在不同水平的染色质组织中受到调控,并可能通过细胞外基质(extracellular matrix,ECM)组成变化进行协调。总之本研究通过对类器官培养物的转录组学和表观基因学分析揭示了肠上皮成熟的关键调节因子,强调了对调控谱的无偏倚分析对于鉴定组织成熟的关键机制价值。
材料方法:
材料:
小鼠:C57BL / 6J小鼠(Taconic),转基因TetON-hYAP/H2B-mCherry
成年和胎儿上皮来源的类器官培养物:从小肠近端收获来自胎儿肠上皮碎片(每个样品来自三个胎儿肠混样)和刮擦的成年小鼠隐窝(每个样本来自一个成年小鼠)。
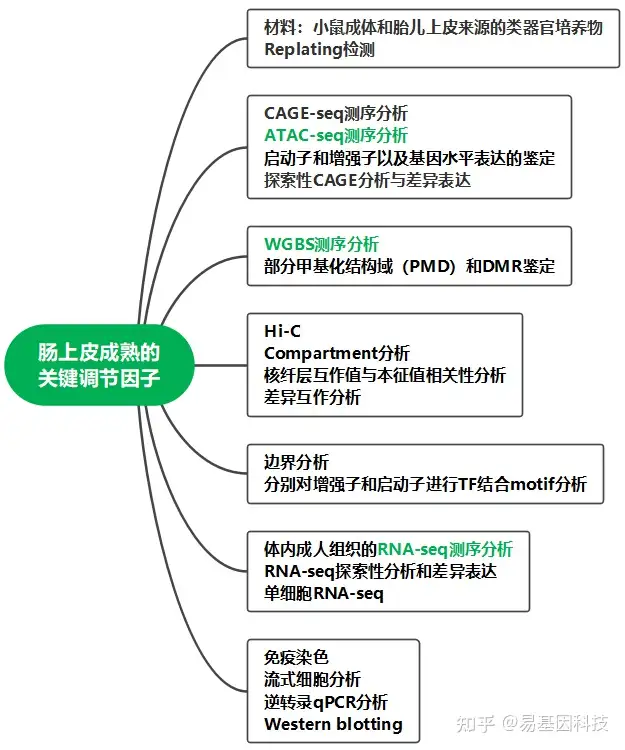
研究结果:
(1)胎儿和成年肠道类器官在转录水平上存在差异
在体外生长允许的条件下,即表皮生长因子(EGF)、Noggin和R-spondin1(ENR),成年肠上皮干细胞自体组织形成出芽类器官,而源自胎儿上皮祖细胞生长为囊状球体。这两个体外系统概括了其体内组织对应物的细胞组成,从而提供了一个易于处理的平台来研究细胞内在机制,而不受微环境或更广泛系统线索的混杂影响。为表征胎儿祖细胞和成体干细胞之间的差异,本研究从小鼠小肠的胎儿(胚胎第E16.5)和成年近端部分获得了3D培养物,分别称为胎儿肠球(胎儿类器官)(fetal enterospheres,FEnS)和成年类器官(adult organoids,aOrg)。用含有CHIR99021和烟酰胺(ENR+ChNic)的培养基瞬时处理已建立的培养物,以降低aOrg培养物中终末分化细胞类型比例。
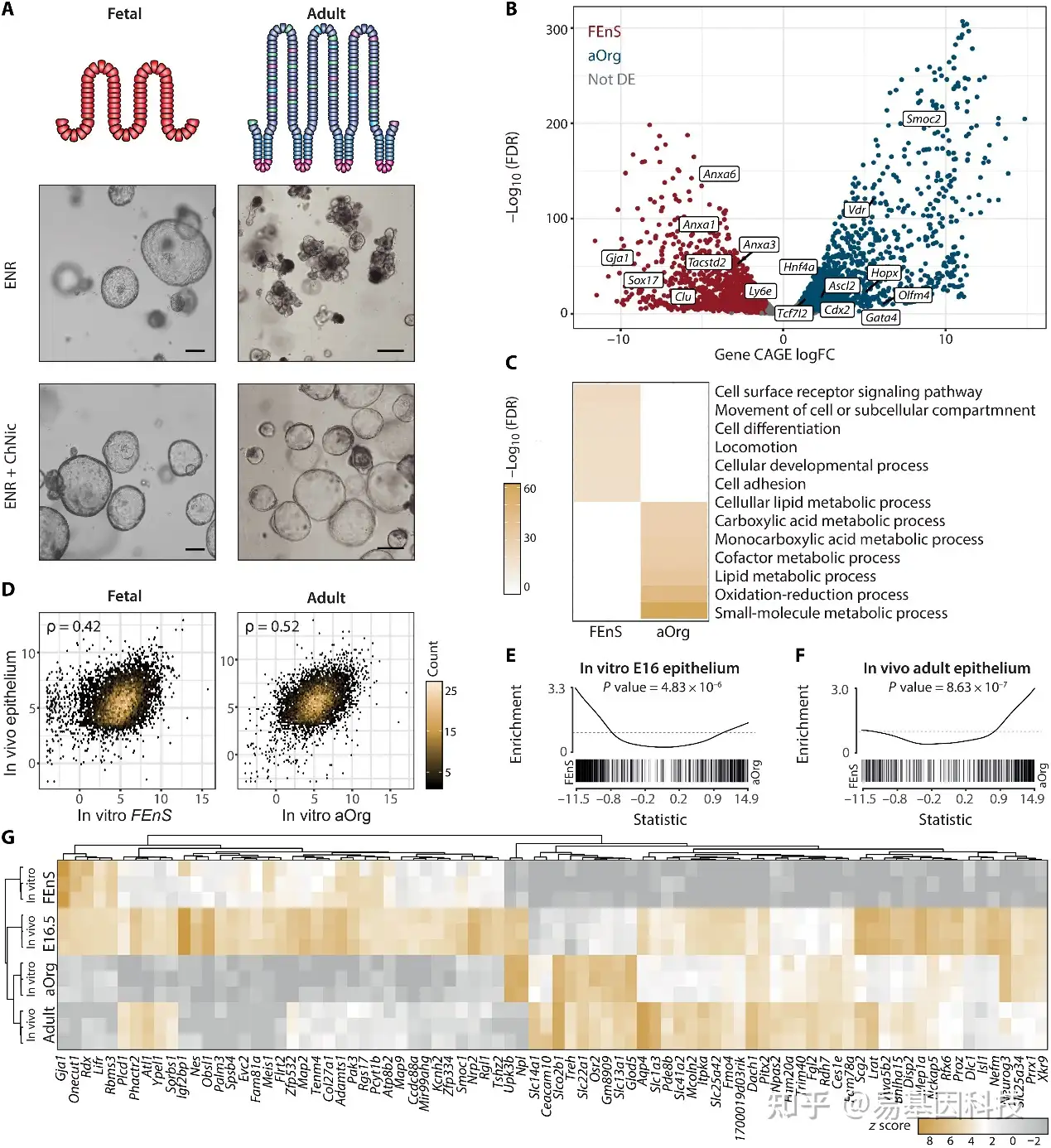
图1:胎儿和成年肠道类器官培养物在转录上存在差异,并概括了其体内对应物。
(A) 胎儿(E16.5)和成年小鼠的小肠上皮(上)及其各自体外类器官(FEnS和aOrg)在ENR和ENR+ChNic中的示意图(下)。比例尺:100μm。
(B) 聚集到ENR+ChNic中维持的培养物的基因水平的CAGE差异表达分析火山图。x轴显示aOrg与FEnS的log2倍数变化(FC),y轴显示log10转换的FDR值。颜色:红色,FEnS富集(FDR<0.05和log2FC<−1);蓝色,aOrg富集(FDR<0.05和log2FC>1);灰色,没有差异表达。
(C) FEnS和aOrg特异性基因与所有表达基因的GO富集分析。颜色表示显著的富集水平(−log10 FDR)。
(D) 体内E16.5上皮与E16.5体外类器官FEnS基因表达密度图(左)以及体内成年隐窝与体外类器官aOrg的基因表现密度图(右)。斯皮尔曼相关系数用ρ表示。
(E) CAGE数据中体内E16.5基因的基因集富集分析(GSEA),按aOrg与FEnS倍数变化排序。
(F) CAGE数据中体内成年隐窝基因的GSEA分析,按aOrg与FEnS的倍数变化排序。
(G) 基因表达热图(前50个变化最大的基因),体内E16.5和成年上皮以及体外FEnS和aOrg的分层聚类。
(2)胎儿和成年状态具有离散的增强子和启动子谱
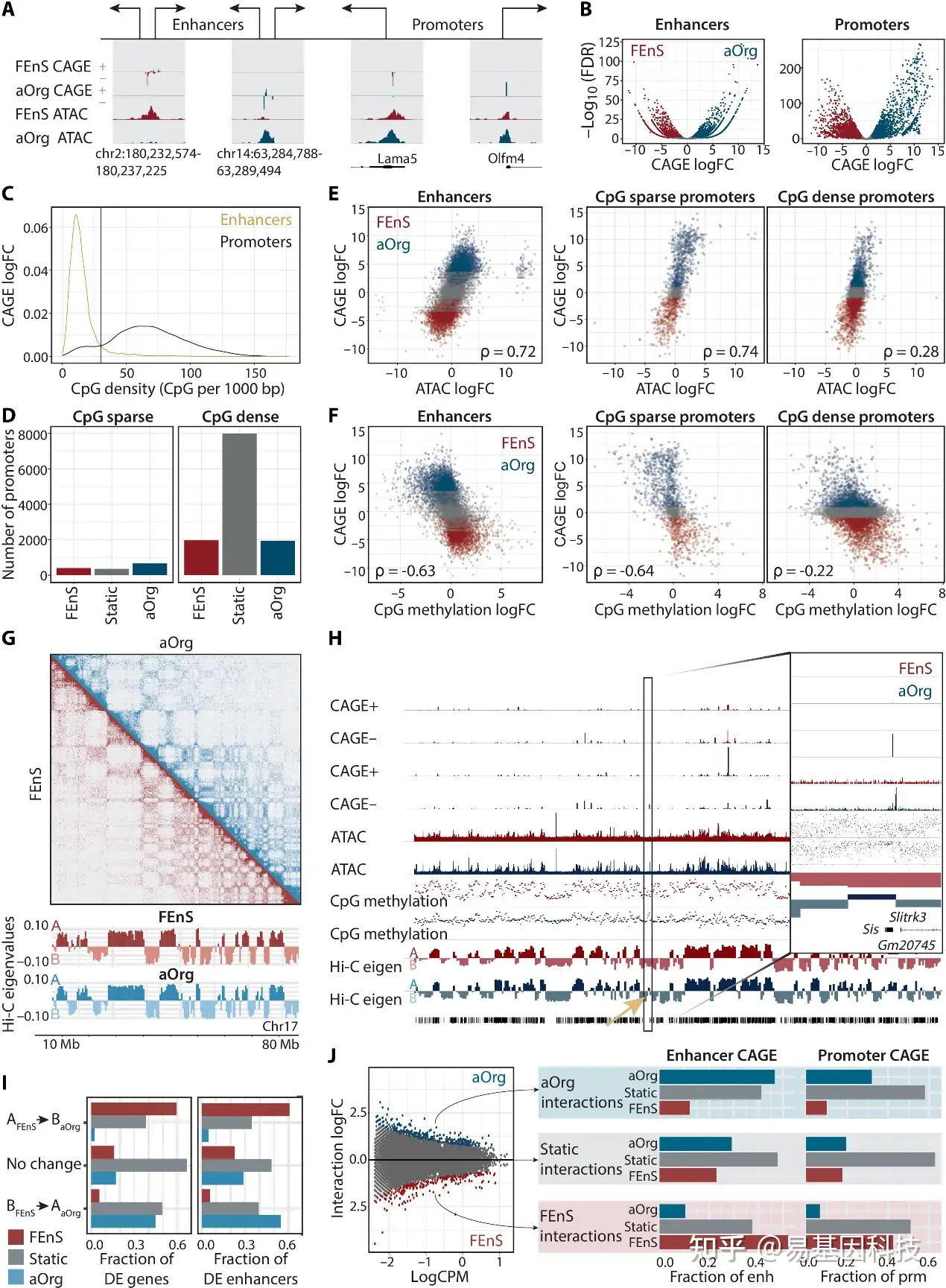
图2:转录变化反映了不同水平染色质的调控差异。
(A) 带CAGE信号的ATAC-seq peaks的典型增强子和启动子示意图。
(B) 增强子(左)和启动子(右)区域的CAGE差异表达,aOrg与FEnS在x轴上的log2倍数变化(logFC),以及在y轴上由差异表达染色的log10转换的FDR值(FEnS,红色;aOrg,蓝色;静态,灰色)。
(C) 增强子中心(黄色)或启动子顶端(黑色)1-kb窗口内CpG数量分布。
(D) 差异表达的CpG稀疏启动子的数量(每1kb<30个CpG;左)和CpG密集启动子(右)的数量。
(E) 差异表达染色的增强子和CpG稀疏和密集启动子的ATAC-logFC和CAGE-logFC之间的相关性。ρ=斯皮尔曼相关系数。
(F) CpG甲基化logFC和CAGE logFC之间的相关性,如(E)所示。
(G) 以chr17坐标为轴的代表性Hi-C互作热图(上)。沿17号染色体500kb的第一主成分(PC1)特征值(FEnS:红色;aOrg:蓝色)和分区(A:深色;B:浅色)。两个图都表示两个生物学重复的平均值。
(H) +链和−链的CAGE信号、ATAC、CpG甲基化、Hi-C PC1的特征值以及沿着chr3的RefSeq注释。放大包含由黄色箭头指示的BFEnS≥AaOrg过渡区域。
(I) 差异表达基因和增强子在组成于A或B区,AFEnS≥BaOrg或BFEnS≥AaOrg区。
(J) 右:100kb bin之间的差分互作,x轴上为每M平均互作计数(CPM),y轴上为互作logFC,用差分互作着色(aOrg,蓝色;FEnS,红色;静态,灰色)。左:差异表达的增强子和启动子重叠x轴上的差异互作bin的比例,以及y轴上增强子和启动子的差异表达比例,按差异互作方向分开。
(3)不同的转录因子与阶段特异性染色质变化相关
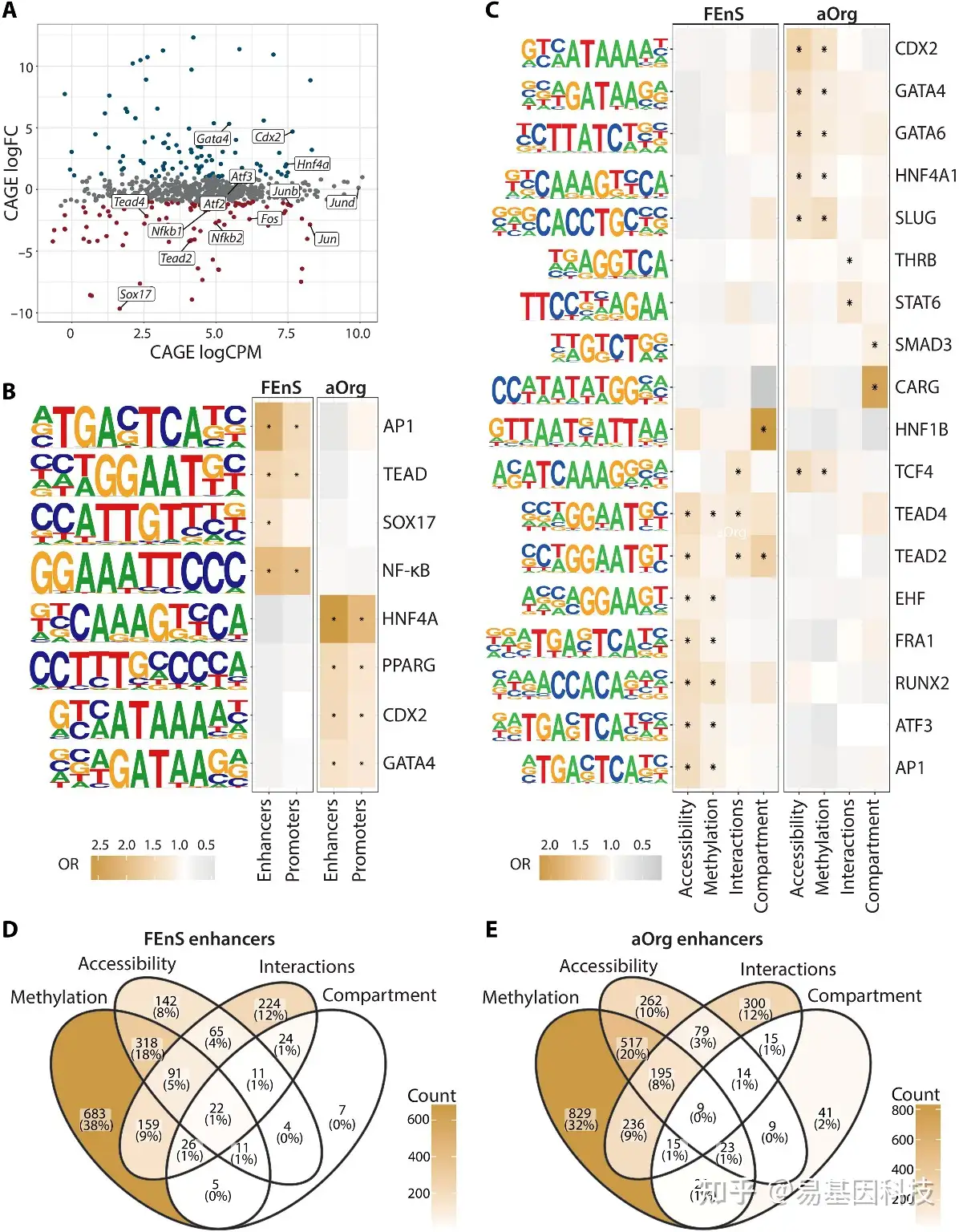
图3:不同的TF网络驱动胎儿和成年状态特异性的启动子和增强子活性。
(A) TF基因水平聚集的CAGE表达log2-CPM在log2倍变化(FC)上的MA图。FEnS或aOrg特异性启动子和增强子中motif富集(如B)的TF分别用红色和蓝色标记。
(B) 转录因子结合位点(TFBS)在增强子和启动子的富集。每一行代表一个TF或一组共有结合motif的TF。颜色分别代表所有表达的增强子和启动子的富集比值比(OR)。FDR值<0.05的富集度用星号(*)标记。
(C) 与所有状态特异性增强子相比,TFBS在差异甲基化、可及性、互作或区间转换增强子上的富集。颜色分别代表所有表达的增强子和启动子的富集比值比。FDR值<0.05的富集度用星号(*)标记。
(D和E)FEnS(D)和aOrg(E)特异性增强子之间的差异甲基化(WGBS)、可及性(ATAC)、互作(Hi-C)和区域状态(Hi-C)的重叠Venn图。
(4)胎儿祖细胞由持续的高YAP活性水平维持
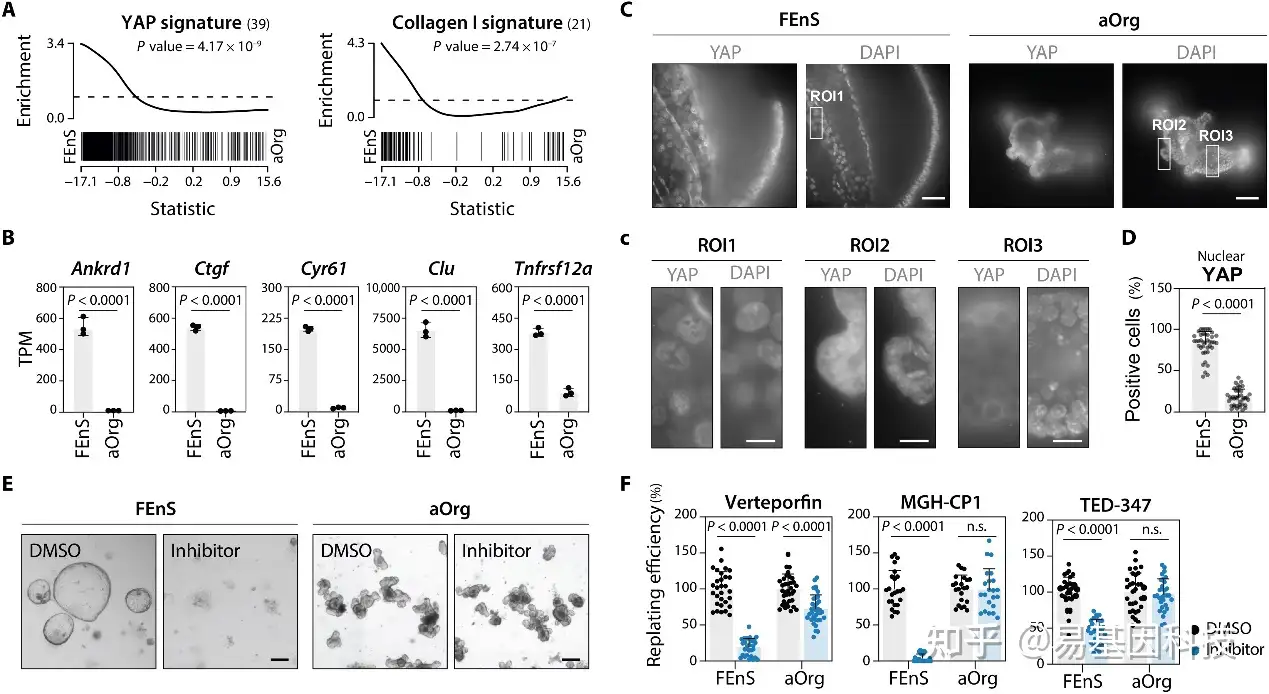
图4:胎儿细胞由持续高水平的YAP活性维持。
- GSEA显示活性YAP相关基因标记富集。
- FEnS和aOrg培养物(ENR培养基)中直接YAP靶基因的mRNA表达水平条形图。采用非配对t检验计算P值。
- 免疫荧光分析显示YAP在ENR培养基中生长的FEnS和aOrg中的亚细胞定位。在大多数胎儿细胞中观察到核信号,而成体细胞大多表现出细胞质定位。比例尺,100μm。(c)高倍放大突出显示感兴趣区域(ROI)。比例尺,25μm。
- 条形图显示具有高YAP核信号的细胞百分比。每个点表示一个单独的FEnS或aOrg结构。采用非配对t检验计算P值。
- YAP信号抑制剂处理的FEnS和aOrg培养物的代表性相位对比图像。显示了MGH-CP1处理的图像。比例尺,200μm。
- 条形图描绘了用所示抑制剂处理后的replating效率。每个点代表至少三个生物学重复。n.s.:不重要。
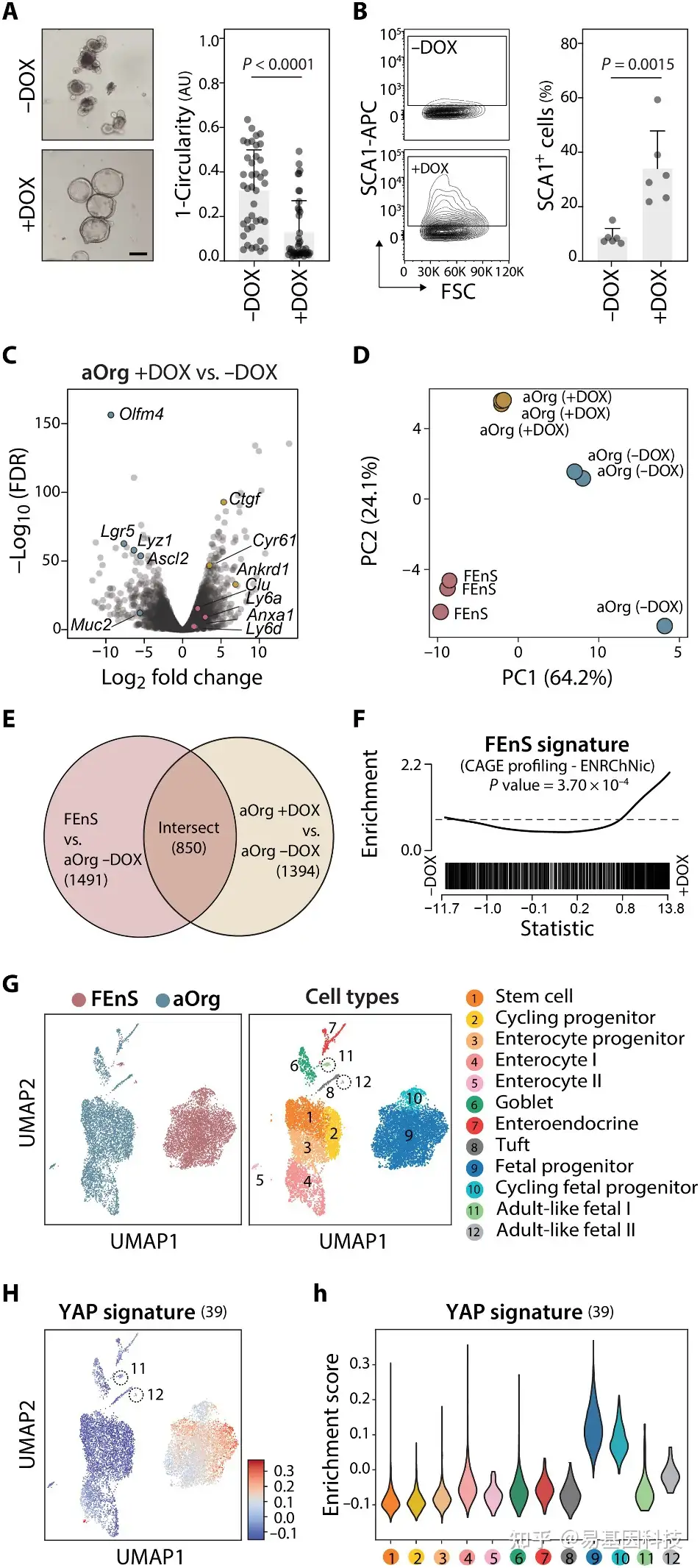
图5:YAP的激活诱导aOrg培养物中的胎儿样表征。
(A) 左图:TetON YAP aOrg培养物在含有或不含有DOX(±DOX)的ENR培养基中的代表性图像。右图,描绘类器官圆形度量化的条形图。比例尺,200μm。
(B) 流式细胞术分析SCA1蛋白在tetON YAP aOrg培养物中的表达。
(C) DOX处理后差异表达的基因的火山图。代表性的成年和胎儿特异性基因分别用蓝色和红色突出显示。代表性的已知YAP靶基因以黄色突出显示。
(D) ENR培养基中FEnS和TetON YAP aOrg(±DOX)培养物的RNA-seq图谱数据的主成分分析(PCA)。主成分分析基于所有样本中最具变异性的前1%基因(137个)进行。
(E) Venn图描绘了FEnS(相对于aOrg−DOX)和aOrg+DOX(相对于aOrg−DOX)培养物中共有上调基因比例。
(F) GSEA显示在YAP诱导(+DOX)时FEnS相关基因标记富集。
(G) 根据样品(左)或细胞类型(右)着色的FEnS和aOrg培养物的单细胞的均匀流形近似和投影(UMAP)可视化。基于已知标记基因的表达对细胞类型簇进行注释。
(H) 覆盖在UMAP细胞类型图上的YAP相关基因标记的平均表达。(h)每个细胞类型簇中YAP标记富集的小提琴图。
(5)YAP活性水平的差异与ECM基因的差异表达相关
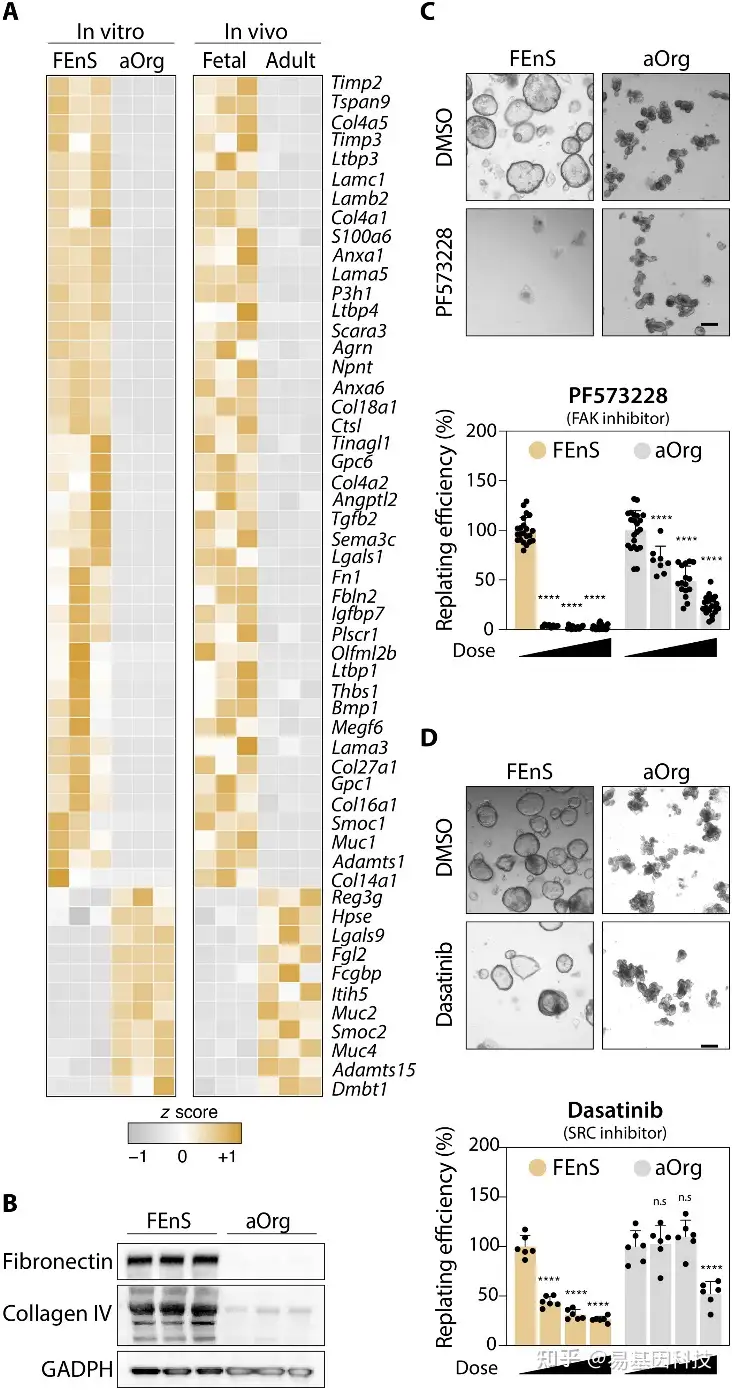
图6:FEnS和aOrg表达差异的ECM基因。
(A) 在FEnS和aOrg以及新鲜分离的胎儿和成年上皮细胞之间一致差异表达的ECM基因(GO:003102-ECM)的mRNA表达水平的热图。
(B) FEnS和aOrg中ECM蛋白纤维连接蛋白和IV型胶原的Western blot分析(各n=3个重复)。使用抗甘油醛-3-磷酸脱氢酶(GAPDH)的印迹作为负载对照。
(C和D)用FAK(C)和SRC(D)激酶的药物抑制剂处理的FEnS和aOrg培养物的Replating检测。每个点代表至少三个生物学重复。多重比较后的双向方差分析显示****P>0.0001(每次剂量与FEnS或aOrg的0μM相比)。比例尺,200μm。
研究结论:
本研究通过对类器官培养物的转录组学和表观基因组学分析揭示了肠上皮成熟的关键调节因子。
- 胎儿和成体上皮组织的类器官虽然培养条件相同,形态相似,但转录水平存在差异;
- 胎儿和成年状态在基因表达、增强子和启动子图谱有显著差异,这与两种细胞状态之间的3D组织、DNA可及性和甲基化的局部变化相关;
- 持续性高YAP转录活性可以维持未成熟的胎儿状态,而不会完全抑制在成体细胞中起作用的分化和代谢基因网络;
- YAP相关转录网络在不同水平的染色质组织中受调控,并可能通过细胞外基质组成变化进行协调。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因科技提供全面的DNA甲基化研究整体解决方案,详询易基因:0755-28317900。
参考文献:Pikkupeura LM, et al. Transcriptional and epigenomic profiling identifies YAP signaling as a key regulator of intestinal epithelium maturation. Sci Adv. 2023 Jul 14;9(28):eadf9460.
相关阅读:
课程回顾|细菌微生物基因表达调控表观研究(原核甲基化+原核转录组)
科研进展 | 全基因组DNA甲基化和转录组分析揭示绵羊羊毛类型变异的关键基因