近日,华中农大王旭彤老师团队在Briefings in Bioinformatics(BIB,IF=9.5)上发表基因组预测新模型SoyDNGP: a web-accessible deep learning framework for genomic prediction in soybean breeding。
简介
传统模型,如线性回归模型(GBLUP, rrBLUP和Bayesian方法),通常难以捕捉复杂的非加性效应。在这种情况下,深度学习方法,如DeepGS和DNNGP,可以发挥作用。它们使用多个隐藏层来捕捉数据中的复杂、非线性关系。然而,这些技术需要大型数据集才能进行准确的预测。
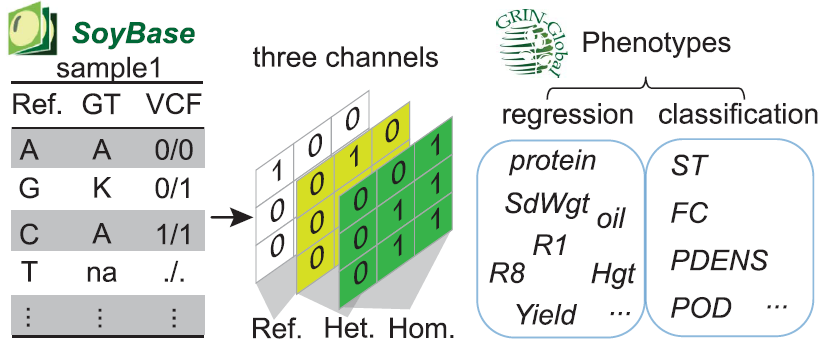
本研究是关于如何使用深度学习方法进行大豆育种中的基因组预测。使用了来自USDA大豆种质资源收藏的数千个大豆样本的基因型和来自GRIN-global web服务器的表型,建立了大豆基因组预测的神经网络框架SoyDNGP。
SoyDNGP具有独特的3D层输入和卷积神经网络(CNN)架构,与其他机器学习(ML)方法和深度学习模型(如deepGS和DNNGP)比较表明,SoyDNGP始终优于这些模型,特别是在回归任务中。测试了SoyDNGP在各种大豆群体中的适用性,包括野生大豆、地方品种和精英品种,都表现出高预测准确性。将SoyDNGP的应用扩展到其他物种,如棉花、玉米、水稻和番茄,模型保持了高预测准确性,证明了它在大豆之外的通用性和有效性。为使基因组预测更被广泛接受,推出了一个用户友好的SoyDNGP网络服务器,配有性状查询和性状预测工具。
材料方法
数据集
SoyDNGP模型训练和预测数据来自SoyBase和GRIN-Global。
来自SoyBase的大约20087个大豆资源的基因型信息,包括基于SoySNP50K 芯片的42509个高置信度SNPs。50K芯片与重测序数据(从NCBI(PRJNA608146)和GSA(CRA002269)下载得两个公共数据集,参考基因组为Williams82 v2)取交集,并填充精选32032个SNP用于模型训练。同时材料也进行了优选,共13784个,代表了来自全球各地的多样性的地方品种和精英培育品种。
表型数据都来自于GRIN-Global数据库(https://npgsweb.ars-grin.gov/gringlobal/search)。最初有23个农艺性状,关注点缩小到了10个关键性状。包括了六个数量性状和四个定性性状。
SoyDNGP的模型结构
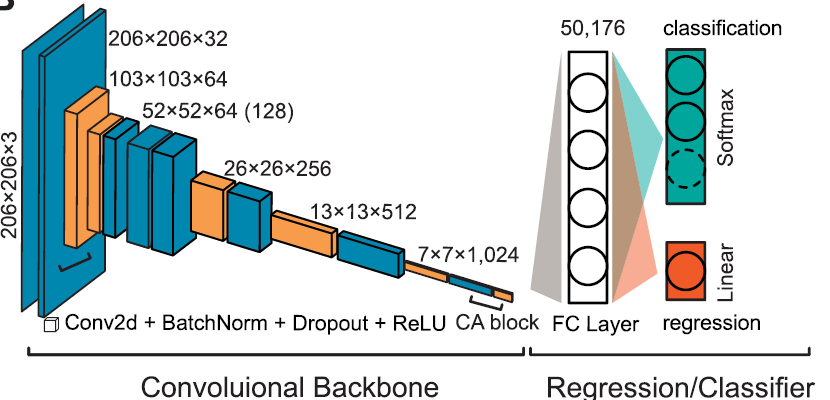
与DNNGP三层宽的卷积架构不同,受到了VGG深度学习网络中分割概念的启发,SoyDNGP采用了一种深而窄的网络结构。具体来说,SoyDNGP是围绕着'卷积块'构建的,每个卷积块包括一个卷积层、一个归一化层和一个激活层(ReLU)。模型结构如下图所示。网络中的每个特征提取单元由一个或两个这些卷积块组成,从而形成了一个有效的特征提取块结构。在卷积序列的末尾,添加了一个全连接层,以增强网络的表达能力。随着网络的深度增加,在每个卷积之后还添加了一个归一化层以增强模型的泛化能力,并添加了一个丢失层(dropout=0.3)以减轻过拟合。总体而言,网络架构集成了12个卷积层和一个单独的全连接层,设计用于处理维度为(206×206×3)的输入张量。
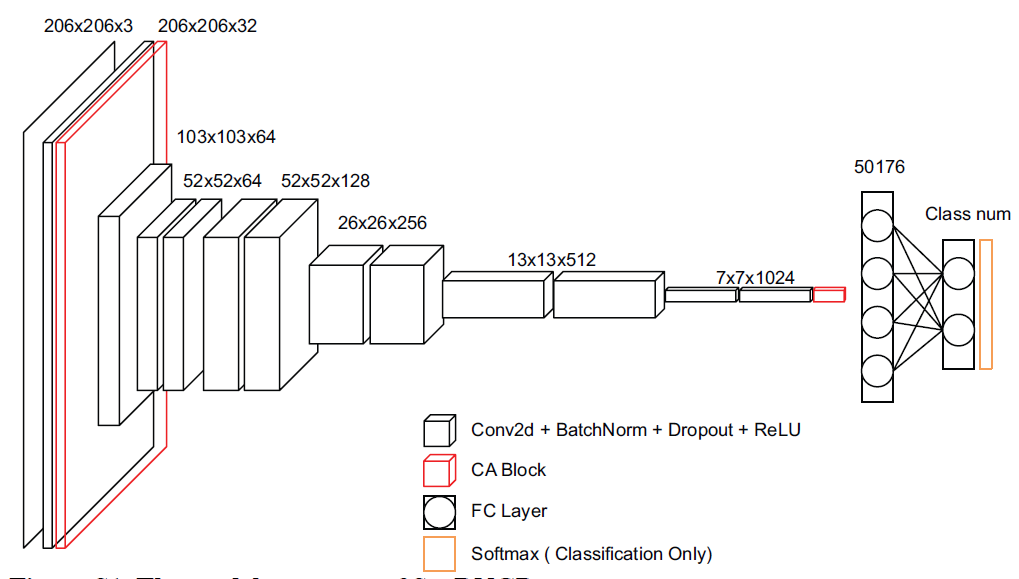
第一个卷积模块使用3×3的卷积核和步幅为1来操作,有效地增加了特征并将特征图从三个通道扩展到32个。随后的卷积块使用4×4的卷积核和步幅为2,增加了特征图的维度,同时减小了每个维度的特征图大小。在接下来的网络结构中,每个特征提取块由两个卷积层组成。
在每个特征提取块中,第一个卷积层根据特征图的维度调整卷积核的大小和采样步幅,以确保对特征图的完全遍历,并使特征图缩放和维度增加具有最小的可能卷积核。第二个卷积层使用3×3的卷积核来重新处理前一层的特征图,增强特征提取。这个过程迭代进行,直到特征图的通道数增加到1024,维度减小到7×7。随后,特征图被展平成一个1D向量,并传递给全连接层进行最终的分类和回归处理。鉴于基于SNP变异的特征矩阵具有丰富的信息密度,作者选择了在卷积填充期间移开传统的零填充方法。采用了一种利用矩阵最外层元素的非对称填充技术,以矩阵边缘作为对称轴。这显著增强了从矩阵中提取特征的能力。
为了避免由网络深度引起的模型训练过拟合的潜在问题,对Adam优化器应用了权重衰减,包括了回归任务的衰减率为1e-5,分类任务的衰减率为0.01。对于定性性状,模型使用常用的交叉熵损失函数进行训练。而对于与数量性状(如蛋白质含量和产量)相关的回归任务,SoyDNGP使用了平滑的L1损失函数(β=0.1)作为其损失函数:
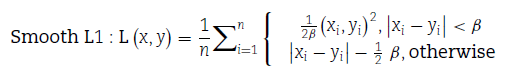
这种特定的损失函数在损失显著时提供恒定的梯度,从而减轻了由于梯度大而导致的训练参数的潜在干扰。相反,当损失最小时,梯度会动态减小,解决了常见的L1损失下收敛挑战。与传统的L1和L2损失函数相比,平滑的L1损失函数提供了更快的收敛速度,对离群值具有更好的鲁棒性,并提高了梯度的平滑度。对于每个性状,在GeForce RTX 3090或RTX A6000上进行了150个epoch的训练,并有选择地保留了在测试集上表现最佳的epoch作为最终模型的权重。
最后,值得注意的是,在第一个和最后一个卷积层之后作者都加入了一个坐标注意力(CA)机制模块。这种策略增强了对特征矩阵和通道之间的位置信息的关注,从而增强了空间信息的提取。SoyDNGP的模型结构由PyTorch(版本2.0.1)设计和实现。
比对模型的处理
为了公平比较不同模型架构,并认识到original deepGS(rDeepGS)模型在特征代表方面的有限能力,选择在保持其整体结构的同时进行了增强。这个结构包括了卷积层、ReLU激活函数、最大池化层和丢失层的组合,所有这些都连接到两个全连接层。
在rDeepGS模型中,用更紧凑的3×3卷积核替换了原来的1×18卷积核。此外,增加了模型中卷积和池化层的数量,达到了六层,总共有12层,形成了修改后的深度rDeepGS(mDeepGS)。这个修改确保了最终特征图的通道数与SoyDNGP相匹配。模型结构如图所示。
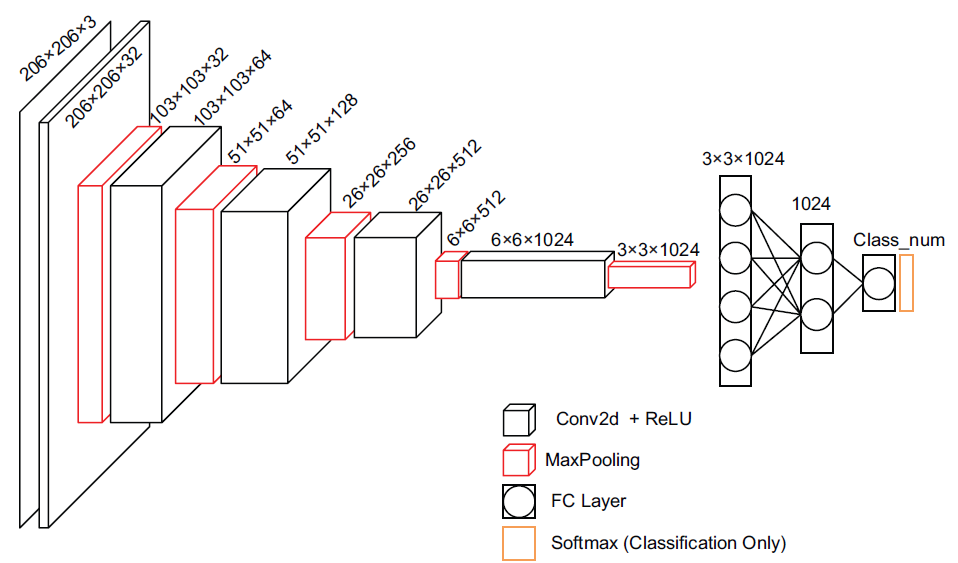
在调整了模型结构之后,保持了训练阶段中与SoyDNGP模型相同的所有其他条件。这种方法使得作者能够在相同的数据集上进行两种模型架构的公平比较。此外,它强调了在特征提取和表达能力领域,深而窄的卷积网络的优越性。
为评估SoyDNGP模型的有效性,作者在相同的数据集上使用了九种传统的机器学习算法进行了并行评估。这些传统模型包括:K-最近邻(KNN)、决策树(DT)、随机森林(RF)、多层感知器(MLP)、自适应增强(Adaboost)、高斯朴素贝叶斯(GNB)和支持向量分类(SVC),使用不同的核(线性、径向基函数和Sigmoid)。每个特征都经过这九种算法的训练,以便比较它们在相同数据集上的性能和稳健性。这些模型的超参数配置如下:对于KNN,将近邻数(n_neighbors)设定为3。对于DT和RF,将树的最大深度(max_depth)限制为5,对于RF,定义森林中树的数量(n_estimators)为10,并且用于最佳分割的特征数量(max_features)为1。对于MLP,规定了L2惩罚(正则化项)参数(alpha)为1。其余模型使用了其各自库中定义的默认参数。
最终实施了一个10折交叉验证方案(n_splits=10),以更加严格地评估模型,确保每次运行都使用不同的拆分(random_state=None)并在创建fold之前对数据进行随机洗牌(shuffle=True)。这样做是为了防止在任何给定fold中某个类别的过度表达可能会影响模型的性能。评估指标包括每个特性类别的精确度、召回率和F1分数。此外,计算了跨fold准确度的平均值和标准差,提供了对模型性能的全面视图。
主要结果
SoyDNGP在大豆基因组预测中展现了出色的能力
SoyDNGP采用从标准VCF文件转换成数据矩阵,每行矩阵都经过重新调整,形成一个大小为(M,M,3)的3D矩阵。在输入的VCF文件中,有三种类型的突变:0/0、0/1和1/1。每种突变类型在特征图中的一个不同的通道中表示,确保突变之间的相对距离。具体来说,0/1突变在第二个通道中表示。特征矩阵中的像素值p[i,j,k]只有两个可能的值:0和1。0的值表示在给定样本的特定SNP位点上存在某种类型的突变,而1的值表示该突变的缺失。关于特征矩阵的维度,根据具有最大SNP变异数的数据集的大小来确定,该数据集有42000个SNPs。这个决策是为了确保模型输入在不同的群体中都有稳健性。为了最小化遗失的SNP位点的影响,反复用样本自己的变异特征填充特征矩阵,直到所有像素都被填充。这种方法允许SoyDNGP结构考虑基因型的类型和其空间关系。两种不同的结构被用于分类(定性特征)和回归(定量特征)任务。
SoyDNGP实现了一个CNN架构,由12个卷积层和一个全连接层组成。在训练阶段,使用Adam优化器(自适应时刻估计),该优化器结合了动量和自适应学习率方法的原则,用于更新模型的权重。这种优化策略允许模型有效地从鞍点逃脱,并加速模型向最优拟合收敛。为了加入注意机制,比较了坐标关注(CA)、挤压和激励(SE)以及卷积块注意模块(CBAM)的性能。
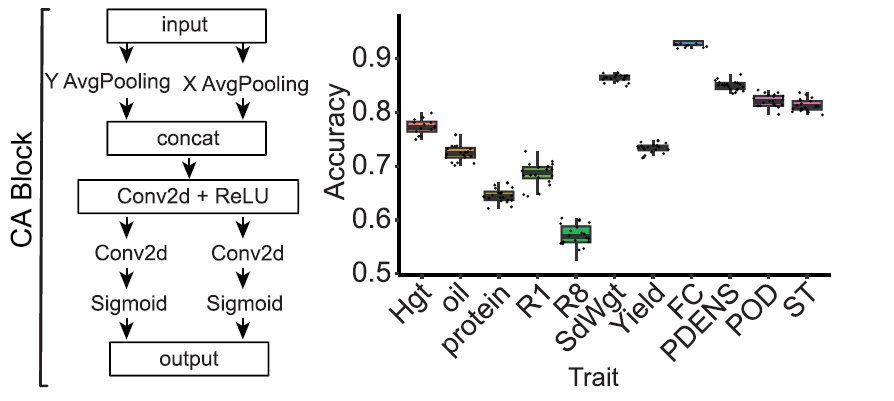
研究表明,集成注意机制显著提高了模型的稳定性和特征表示能力。SE注意机制只关注通道信息。另一方面,CABM注意机制包括通道和位置信息的提取,但没有实现这些特征的有效融合。CA注意机制纠正了这些局限性,使得从特征图中提取空间位置信息变得更为优越。此外,仅在参数数量和每秒浮点运算中存在边际差异,CA注意机制在模型训练过程中展现了更快的拟合速度。在这些选择中,CA在性能上超过了SE和CBAM,使其成为最终架构的首选。
CA模块被策略性地放置在初始和最终的卷积层之后,增强了模型对特征矩阵内部的空间细节和通道间相关性的关注能力。随后,尝试在SoyDNGP模型中添加更复杂的残差网络模块(Residual Block),但这些复杂的结构增加了参数的数量和计算负载,而没有显著提高性能。因此,作者选择了CA+基线网络结构作为最终模型。
为了确定模型训练的最佳样本大小,使用不同数量的样本训练模型,并监测预测性能。样本被分为2k、5k、8k和10k的训练组,每组都与11784、8784、5784和3784的测试集配对,经过150个epochs。研究表明,2k样本大小在准确性和其他指标方面的性能较低,而在较大的样本大小中观察到的差异并不显著。最终发现5k样本大小最适合模型构建。
单独预测的准确性结果显示,回归任务的预测准确度从R8的0.56到SdWgt的0.87,而分类任务的预测准确度从ST的0.82到FC的0.96。这个结论也得到了归一化的观察和预测表型值之间的绝对误差的支持。经过广泛的测试,该模型在回归和分类任务中都一直提供出色的预测准确性。有些特征确实展示了不平衡的类分布,导致在代表性不足的类别中模型性能较差。但是,对于具有相对均衡的类分布的表型,模型的表现特别好。例如,在“Flower color”的情况下,该模型在平衡的二分类中表现得很好。相反,在“H_CLR”的情况下,模型对于“Br”和“Bl”类别的准确度明显较低,作者将这一结果归因于这些类别在数据集中的分布偏斜。
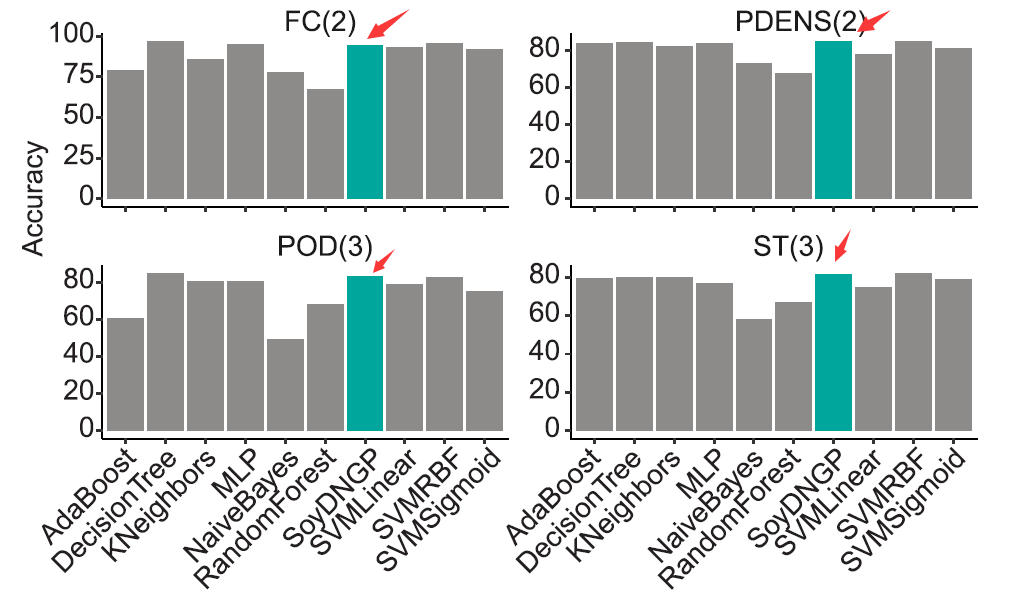
大豆基因组预测中SoyDNGP与其他算法的性能比较
使用相同的数据集来训练SoyDNGP模型和其他机器学习方法。尽管传统的机器学习没有针对回归任务进行优化,但发现其中一些能够以高准确度执行分类任务。例如,决策树(DT)模型对FC和POD的预测准确度分别达到了0.97和0.85。使用SVM RBF模型,ST和PDENS的准确度分别达到了0.82和0.84。在测试的九种机器学习方法中,SoyDNGP在所有分类特征上表现出平衡的性能,准确度范围从0.82(ST)到0.94(FC)。
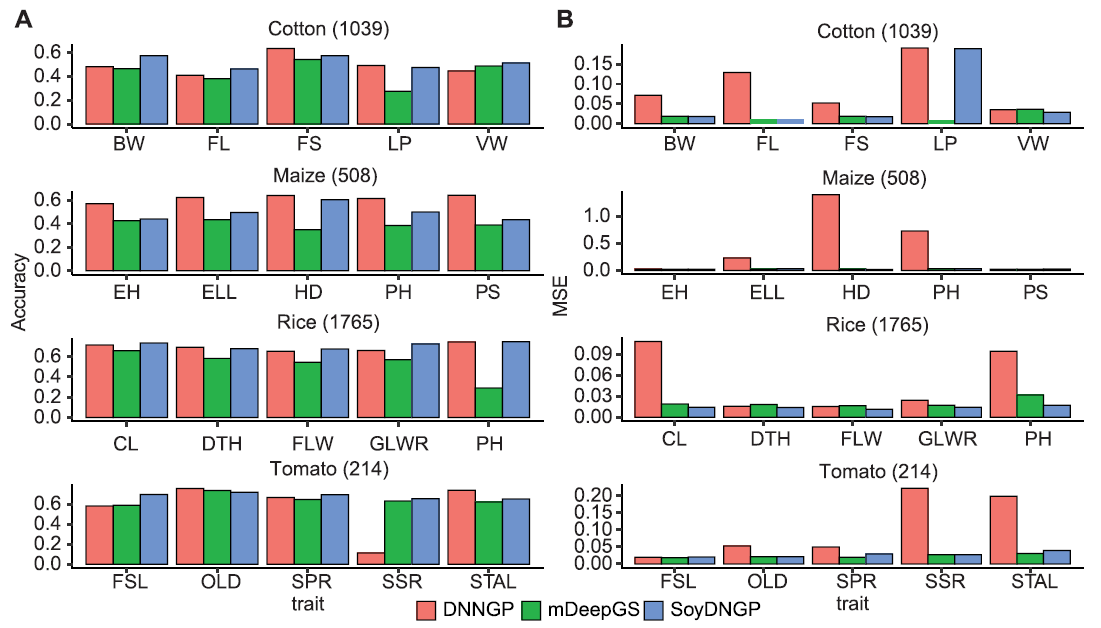
评估SoyDNGP与其他基于卷积神经网络(CNN)的深度学习模型(如deepGS和DNNGP)的性能。原始版本的deepGS(rDeepGS)在回归任务中表现不佳,尽管在分类任务中与其他方法相比表现相当。为了确认deepGS结构的效率,使用重新设计的修改版本(mDeepGS)。使用与SoyDNGP相同的数据集训练这些模型表明,无论是在回归任务的特性还是训练样本数量方面,与mDeepGS和DNNGP相比,SoyDNGP在测试中表现更好。DNNGP的相关系数(r)与SoyDNGP的相关系数相差约5%。此外,DNNGP的预测值与实际值之间的差异(通过均方误差MSE衡量)几乎比SoyDNGP大了10倍。这表明DNNGP只具有预测趋势和定性描述的能力,但在定量方面缺乏精度。由于其浅层结构,mDeepGS无法有效处理回归任务的复杂性,因此无法准确拟合。
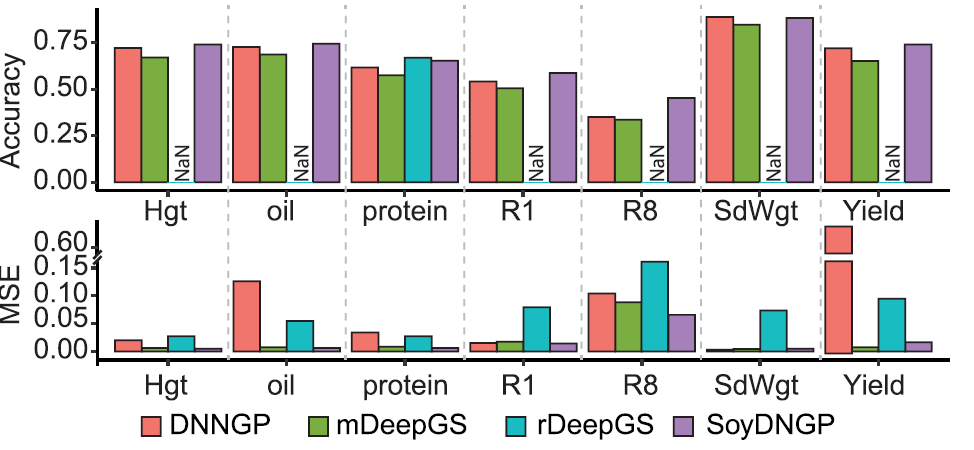
三个深度学习模型——DeepGS、DNNGP和SoyDNGP——在定性特征分类任务上表现相当。然而,在回归任务中它们的性能存在显著差异。rDeepGS,类似于传统机器学习模型,无法有效拟合回归任务。这主要归因于分类任务的复杂性较低,可以有效地使用机器学习技术解决,从而导致模型在这些任务中性能差异不大。此外,rDeepGS和mDeepGS的运行时间较短,但性能不令人满意。SoyDNGP和DNNGP几乎具有相同的运行时间,但SoyDNGP的参数量超过DNNGP的10倍。这更高的参数量使SoyDNGP能够更好地学习和拟合更复杂的特征,表现出更强的泛化能力。这些证据表明,与其他方法相比,SoyDNGP模型结构在基因组预测中具有明显优势。
SoyDNGP模型在不同大豆群体中的多功能预测能力
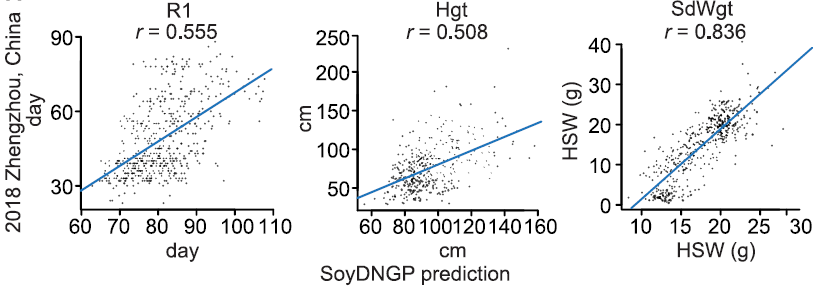
本模型是基于美国农业部大豆种质资源收集的数据开发,可能在不同国家和纬度的其他资源上的应用不确定。为评估SoyDNGP模型的预测能力,将其应用于一个包括559个大豆资源的大豆群体,包括121个野生大豆,207个地方品种和231个精英品种。对16个定性特征和12个定量特征进行了预测。为了验证对重要特征的预测准确性,将2018年在中国郑州种植的指定大豆特征的表型与我们的预测进行了对比验证。分析揭示了预测值与实际值之间的强正相关关系。例如,R1和Hgt的相关性分别为0.56和0.51。最令人印象深刻的是,SdWgt的预测准确性达到了0.84。因此,SoyDNGP预测模型在不同的大豆群体中具有广泛的适用性。对于不同群体中粒重的高预测准确性的一个可能解释是,与其他特征(如R1和Hgt)相比,环境因素在这一特征中起较小的作用。
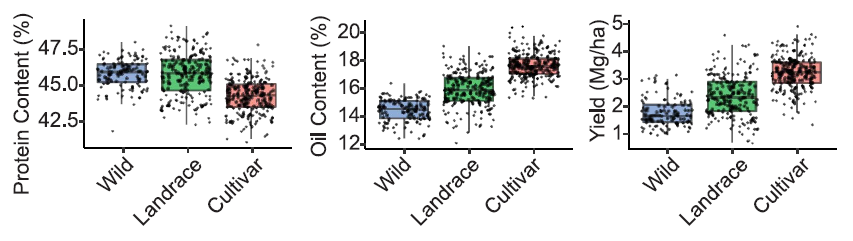
尽管野生大豆没有包含在模型训练中,但模型仍然可用于预测野生大豆的特征。例如预测显示,与地方品种和精英品种相比,野生大豆的蛋白质含量高,油分含量和产量低,这与以前的大豆研究一致。这也意味着野生大豆和栽培大豆之间的基因交换可能受到显著的基因流的促进。
SoyDNGP 在大豆之外的广泛应用
为评估SoyDNGP的多功能性和效果,对其他物种进行了测试,使用来自棉花、玉米、水稻和番茄群体的基因型数据和五个代表性特征。为方便比较,同样的数据集也被应用于DNNGP和mDeepGS。除了mDeepGS展示出最低的准确性外,SoyDNGP的预测准确度范围从玉米的平均0.50到水稻的平均0.71。在DNNGP中观察到了类似的性能范围(0.49–0.69)。值得注意的是,对于像玉米和番茄这样的较小样本大小,分别有214和508个样本,DNNGP的表现优于SoyDNGP。然而,在样本数量超过1000的大型群体,如棉花和水稻中,SoyDNGP证明了其优越性。尽管在准确性上有相似之处,DNNGP的均方误差(MSE)普遍高于SoyDNGP。基于这些发现,可以得出结论,SoyDNGP不仅能够训练和预测其他物种的性状表型,而且在性能上超越了其他方法,从而证明了其强大的多功能性和有效性。因此,SoyDNGP被视为一个有前景的基因组预测工具,其应用可能不仅限于大豆,还可能扩展到其他作物和生物,从而推进基因组学和育种研究的进步。
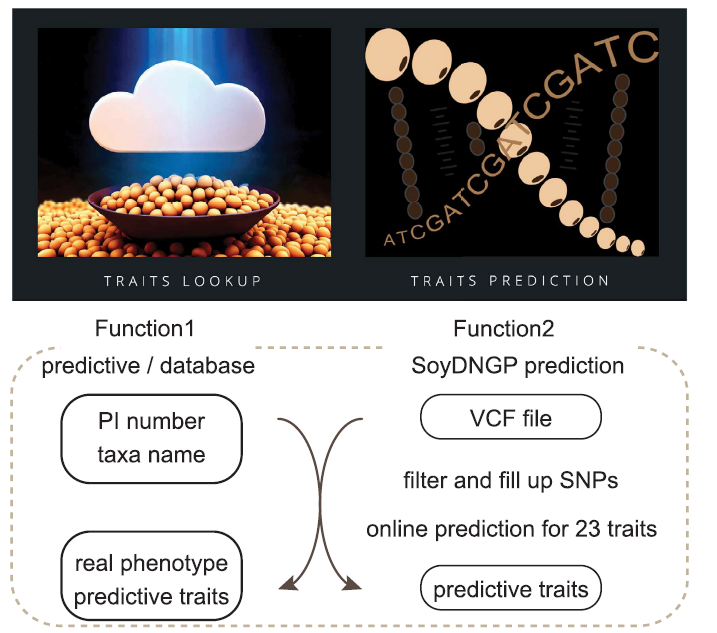
SoyDNGP是一个面向大豆基因组预测的开放友好的web服务器
为了让没有深度编程专业知识的用户能够访问SoyDNGP,作者建立了一个web服务器,可在http://xtlab.hzau.edu.cn/SoyDNGP上访问。SoyDNGP平台提供了两个便于用户浏览特性信息的界面。
第一个功能,“Trait Lookup”,允许用户输入分类标识符,例如植物引入(PI)号码或传统名称,来检查相应的记录是否已经在数据库中。此外,“Trait Lookup”部分包括了500个大豆品种的预先存在的性状预测,这些品种是除了USDA大豆种质资源收集以外的品种,并且都有可用的重测序数据。
作者每天都在增加这个数字,为用户提供一个不断扩展的数据集。这个功能对希望基于某些性状预测选择特定大豆品种的用户非常有益,从而提高了SoyDNGP的效率。
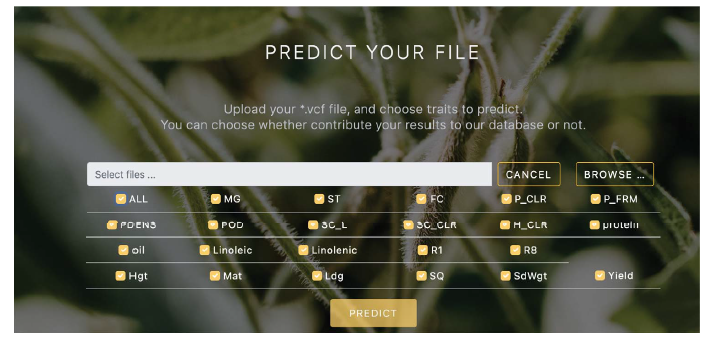
第二个功能,即“Trait Prediction”工具,允许用户上传一个VCF文件,然后预测模型使用这个文件来预测性状值。作者还为用户提供了选择性地为查询数据库提供丰富内容的选项。如果用户选择贡献,他们将不需要在将来重新访问他们的结果时再次运行预测。
讨论
已有的深度学习模型DeepGS和DNNGP都使用1D向量作为模型输入,这在表示复杂的SNP位点特征信息时可能有局限性。这种简化的方法可能无法捕捉基因型变异的全部深度,从而影响模型的预测准确性。此外,这些模型中使用的浅层、宽卷积结构可能不适合捕捉数据内部的复杂关系。
SoyDNGP可填补这些差距,这是一个使用更复杂的3D矩阵作为输入特征并采用更合理的数据处理结构的模型。SoyDNGP相对于deepGS和DNNGP模型具有几个显著优势:增强的特征密度、优化的结构、减少的特征丢失、通过正则化实现的稳定训练以及引入注意力机制。
SoyDNGP使用的3D矩阵包含位置和突变信息,更适合卷积神经网络(CNN)并提供更丰富的特征密度。DNNGP和deepGS使用浅层宽卷积层,SoyDNGP则采用更深层、更窄的架构,使用堆叠的小卷积核来实现更好的特征提取和效率。SoyDNGP使用卷积步幅为2,而不是像deepGS那样使用最大池化,有效地融合和降采样特征并最小化信息损失。SoyDNGP在卷积之间集成了Dropout和Batchnorm,并使用L2正则化,这比其前身更有效地提高了模型的稳定性并防止过拟合。SoyDNGP使用坐标注意机制来考虑空间和通道信息,从而提高了其特征提取能力。
但本研究在数据集上面临两个主要的挑战。首先是不平衡的样本分布。许多被考虑的性状有多个类别,常常带有复杂的细分,导致样本数在这些类别中的分布是偏斜的。这种不平衡在有效地训练一个稳健的模型上构成了挑战。第二个挑战是数据的可靠性。像株高、开花时间和成熟时间这样的性状常常在没有标准化协议的情况下进行测量,导致在数据收集过程中出现重大错误,影响模型的预测性能。
实验显示,随着样本大小的增加,像DNNGP和DeepGS这样的浅层神经网络开始失去其在定量表示性状方面的效果。鉴于生物技术的快速发展,越来越需要像SoyDNGP这样的更深层次的模型。作者的关注点仍然是在GS领域的模型解释性,因为它在这里比在其他计算学科,如图像识别或自然语言处理,更为关键。设计的模型使其尽可能地可解释,最小化不可逆的操作,如池化。这与识别与不同性状可能相关的关键基因位置的更广泛目标是一致的。
此外,GS领域缺乏一个像YOLO或BIOBERT在其各自领域所提供的那样通用适应性的深度学习平台。尽管Kumar等人最近推出了DeepMap,但它在灵活性和可扩展性方面有限。基于此,作者开发了SoyDNGP Next PyPI包。基于基线SoyDNGP算法,此包允许用户通过简单的Python命令轻松重构模型、训练数据和做出预测,从而增强模型对各种数据集的适应性。
总之,作者创建并验证了SoyDNGP,一个专门为预测大豆性状定制的基于CNN的模型。结果显示了SoyDNGP一贯优于deepGS和DNNGP模型,展示了较高的准确性和降低的模型复杂性。此外,作者测试了SoyDNGP在棉花、玉米、水稻和番茄等多种作物上的适用性,突显了它作为一个稳健且多功能的基因组预测工具的潜力。为了扩展SoyDNGP的应用,建立了一个用户友好的web服务器,为用户提供了简单访问特征预测和使用VCF文件计算特征的能力。
资源获取
Web服务器:http://xtlab.hzau.edu.cn/SoyDNGP
软件包安装:pip install SoyDNGPNext
模型代码:
https://github.com/IndigoFloyd/SoyDNGPNext
网站代码:
https://github.com/IndigoFloyd/SoybeanWebsite
源码:
https://doi.org/10.6084/m9.figshare.23537067.v2
若要获取原文、附件及相关数据,请关注公众号”生物信息与育种“,后台回复:SoyDNGP。