写在前面
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:(Seurat V1) 单细胞基因表达数据的空间重建
2.文献阅读:(Seurat V2) 整合跨越不同条件、技术、物种的单细胞转录组数据
3.文献阅读:(Seurat V3) 单细胞数据综合整合
4.文献阅读:(Seurat V4) 整合分析多模态单细胞数据
5.文献阅读:(Seurat V5) 用于集成、多模态和可扩展单细胞分析的字典学习
教程篇:
1.Seurat Tutorial 1:常见分析工作流程,基于 PBMC 3K 数据集
2.Seurat Tutorial 2:使用 Seurat 分析多模态数据
3.Seurat Tutorial 3:scRNA-seq 整合分析介绍
4.Seurat Tutorial 4:映射和注释查询数据集
5.Seurat Tutorial 5:使用 reciprocal PCA (RPCA) 快速整合
6.Seurat Tutorial 6:整合大型数据集的技巧
::: block-1
目录
- 1 加载数据并单独处理每种模式
- 2 识别 scRNA-seq 和 scATAC-seq 数据集之间的锚点
- 3 通过标签转移注释 scATAC-seq 细胞
- 4 共同嵌入 scRNA-seq 和 scATAC-seq 数据集
:::
官网教程:
https://satijalab.org/seurat/articles/atacseq_integration_vignette
单细胞转录组学改变了我们表征细胞状态的能力,但深入的生物学理解需要的不仅仅是 clusters 的分类列表。随着测量不同细胞模式的新方法出现,一个关键的分析挑战是整合这些数据集以更好地了解细胞身份和功能。例如,用户可以在同一生物系统上执行 scRNA-seq 和 scATAC-seq 实验,并使用同一组细胞类型标签对两个数据集进行一致注释。这种分析尤其具有挑战性,因为 scATAC-seq 数据集很难注释,因为以单细胞分辨率收集的基因组数据稀疏,而且 scRNA-seq 数据中缺乏可解释的基因标记。
在 Stuart、Butler et al, 2019 中,我们介绍了整合从同一生物系统收集的 scRNA-seq 和 scATAC-seq 数据集的方法,并在此小节中演示了这些方法。我们特别展示了以下分析:
- 如何使用带注释的 scRNA-seq 数据集来标记 scATAC-seq 实验中的细胞
- 如何从 scRNA-seq 和 scATAC-seq 中 co-visualize(co-embed)细胞
- 如何将 scATAC-seq 细胞投影到源自 scRNA-seq 实验的 UMAP 上
该小节广泛使用了 Signac 软件包,该软件包最近开发用于分析以单细胞分辨率收集的染色质数据集,包括 scATAC-seq。请访问 Signac website,了解用于分析 scATAC-seq 数据的其他说明和文档。
我们使用来自 10x Genomics 的公开可用的约 12,000 个人类 PBMC ‘multiome’ 数据集演示了这些方法。在此数据集中,scRNA-seq 和 scATAC-seq profiles 同时收集在同一细胞中。出于本小节的目的,我们将数据集视为源自两个不同的实验并将它们整合在一起。由于它们最初是在相同的细胞中测量的,因此这提供了我们可以用来评估整合准确性的 ground truth。我们强调,我们在这里使用多组学数据集是为了演示和评估目的,用户应该将这些方法应用于单独收集的 scRNA-seq 和 scATAC-seq 数据集。我们提供了一个单独的加权最近邻(WNN)小节,它描述了多组学单细胞数据的分析策略。
1 加载数据并单独处理每种模式
PBMC multiome 数据集可从 10x Genomics 获取。为了方便加载和探索,它也作为我们 SeuratData 包的一部分提供。我们分别加载 RNA 和 ATAC 数据,并假设这些数据是在单独的实验中测量的。我们在 WNN 小节中对这些细胞进行了注释,注释也包含在 SeuratData 中。
library(SeuratData)
# install the dataset and load requirements
InstallData("pbmcMultiome")
这一步可能出现报错,推测是因为国外网络的问题,需要手动下载安装
wget http://seurat.nygenome.org/src/contrib/pbmcMultiome.Seurat> Data_0.1.4.tar.gzinstall.packages('pbmcMultiome.SeuratData_0.1.4.tar.gz', repos > = NULL, type = "source")
library(Seurat)
library(Signac)
library(EnsDb.Hsapiens.v86)
library(ggplot2)
library(cowplot)
# load both modalities
pbmc.rna <- LoadData("pbmcMultiome", "pbmc.rna")
pbmc.atac <- LoadData("pbmcMultiome", "pbmc.atac")
# 重复 WNN 小节中执行的 QC 步骤
pbmc.rna <- subset(pbmc.rna, seurat_annotations != "filtered")
pbmc.atac <- subset(pbmc.atac, seurat_annotations != "filtered")
# 独立执行每种模式的标准分析 RNA 分析
pbmc.rna <- NormalizeData(pbmc.rna)
pbmc.rna <- FindVariableFeatures(pbmc.rna)
pbmc.rna <- ScaleData(pbmc.rna)
pbmc.rna <- RunPCA(pbmc.rna)
pbmc.rna <- RunUMAP(pbmc.rna, dims = 1:30)
# ATAC 分析添加基因注释信息
annotations <- GetGRangesFromEnsDb(ensdb = EnsDb.Hsapiens.v86)
seqlevelsStyle(annotations) <- "UCSC"
genome(annotations) <- "hg38"
Annotation(pbmc.atac) <- annotations
# 我们排除第一个维度,因为这通常与测序深度相关
pbmc.atac <- RunTFIDF(pbmc.atac)
pbmc.atac <- FindTopFeatures(pbmc.atac, min.cutoff = "q0")
pbmc.atac <- RunSVD(pbmc.atac)
pbmc.atac <- RunUMAP(pbmc.atac, reduction = "lsi", dims = 2:30, reduction.name = "umap.atac", reduction.key = "atacUMAP_")
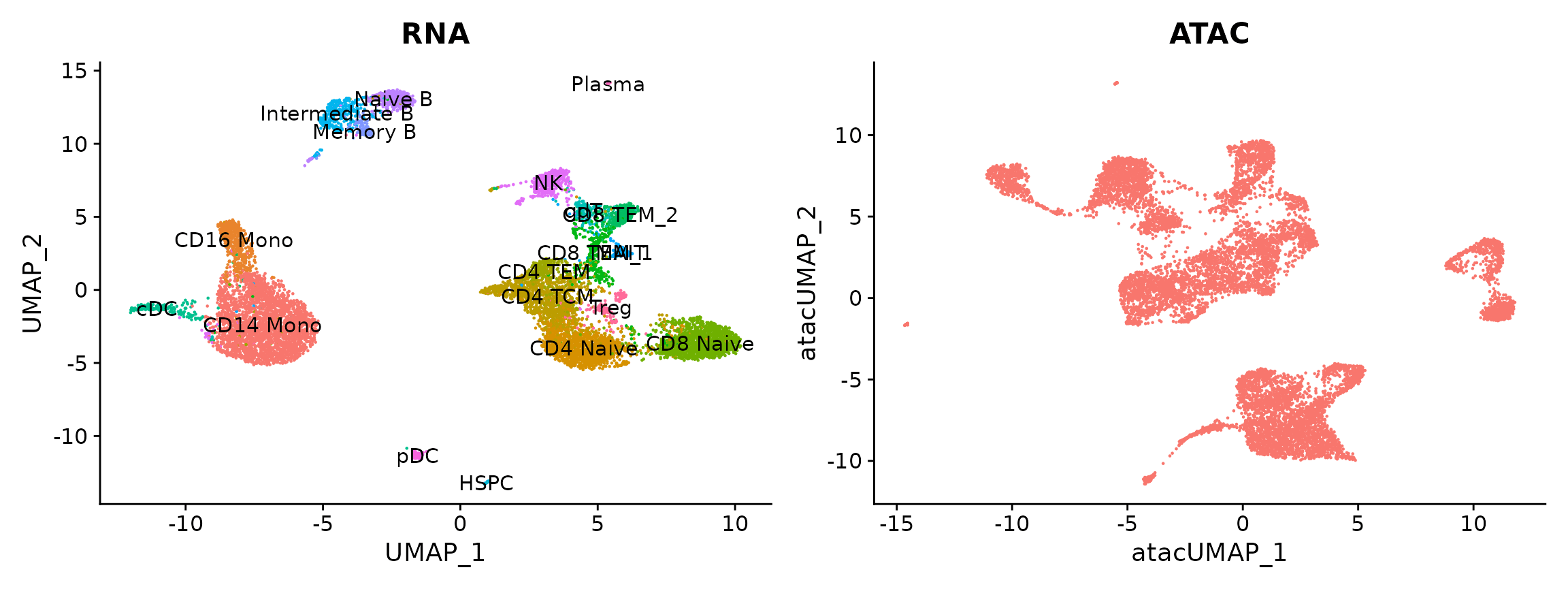
现在我们绘制两种模式的结果。先前已根据转录组状态对细胞进行了注释。我们将预测 scATAC-seq 细胞的注释。
p1 <- DimPlot(pbmc.rna, group.by = "seurat_annotations", label = TRUE) + NoLegend() + ggtitle("RNA")
p2 <- DimPlot(pbmc.atac, group.by = "orig.ident", label = FALSE) + NoLegend() + ggtitle("ATAC")
p1 + p2
2 识别 scRNA-seq 和 scATAC-seq 数据集之间的锚点
为了确定 scRNA-seq 和 scATAC-seq 实验之间的 'anchors',我们首先使用 Signac 包中的 GeneActivity() 函数对 2 kb 上游区域和 gene body 中的 ATAC-seq counts 进行量化,对每个基因的转录活性进行粗略估计。然后,将 scATAC-seq 数据中的基因活性评分以及 scRNA-seq 中的基因表达量化用作典型相关分析的输入。我们对 scRNA-seq 数据集中确定为高度可变的所有基因进行这种量化。
# 量化基因活性
gene.activities <- GeneActivity(pbmc.atac, features = VariableFeatures(pbmc.rna))
# 添加基因活性作为一个新的 assay
pbmc.atac[["ACTIVITY"]] <- CreateAssayObject(counts = gene.activities)
# 归一化基因活性
DefaultAssay(pbmc.atac) <- "ACTIVITY"
pbmc.atac <- NormalizeData(pbmc.atac)
pbmc.atac <- ScaleData(pbmc.atac, features = rownames(pbmc.atac))
# Identify anchors
transfer.anchors <- FindTransferAnchors(reference = pbmc.rna, query = pbmc.atac, features = VariableFeatures(object = pbmc.rna), reference.assay = "RNA", query.assay = "ACTIVITY", reduction = "cca")
3 通过标签转移注释 scATAC-seq 细胞
识别 anchors 后,我们可以将注释从 scRNA-seq 数据集转移到 scATAC-seq 细胞上。注释存储在 seurat_annotations 字段中,并作为 refdata 参数的输入提供。输出将包含一个矩阵,其中包含每个 ATAC-seq 细胞的预测和置信度得分。
celltype.predictions <- TransferData(anchorset = transfer.anchors, refdata = pbmc.rna$seurat_annotations, weight.reduction = pbmc.atac[["lsi"]], dims = 2:30)
pbmc.atac <- AddMetaData(pbmc.atac, metadata = celltype.predictions)
为什么你选择不同的(非默认)值来降维和加权降维?
在 FindTransferAnchors() 中,当在 scRNA-seq 数据集之间 transferring 时,我们通常将 PCA 结构从 reference 投影到 query 上。然而,当跨模态 transferring 时,我们发现 CCA 更好地捕获共享特征相关结构,因此在这里设置 reduction = 'cca'。此外,默认情况下,在 TransferData() 中,我们使用相同的投影 PCA 结构来计算影响每个细胞预测的 anchors 局部邻域的权重。在 scRNA-seq 到 scATAC-seq transfer 的情况下,我们使用通过在 ATAC-seq 数据上计算 LSI 学习到的低维空间来计算这些权重,因为这可以更好地捕获 ATAC-seq 数据的内部结构。
执行 transfer 后,ATAC-seq 细胞已将预测注释(从 scRNA-seq 数据集 transfer)存储在 Predicted.id 字段中。由于这些细胞是使用 multiome kit 测量的,因此我们还有可用于评估的 ground-truth 注释。您可以看到预测的注释和实际的注释非常相似。
pbmc.atac$annotation_correct <- pbmc.atac$predicted.id == pbmc.atac$seurat_annotations
p1 <- DimPlot(pbmc.atac, group.by = "predicted.id", label = TRUE) + NoLegend() + ggtitle("Predicted annotation")
p2 <- DimPlot(pbmc.atac, group.by = "seurat_annotations", label = TRUE) + NoLegend() + ggtitle("Ground-truth annotation")
p1 | p2
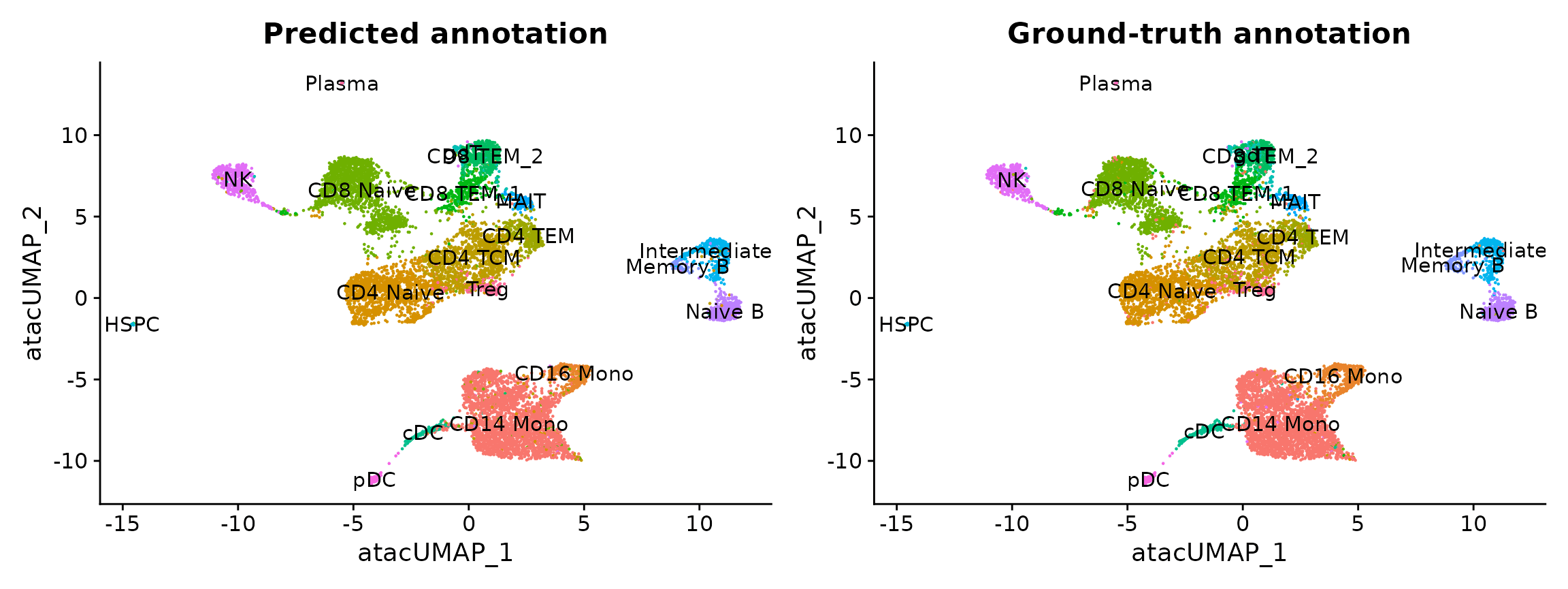
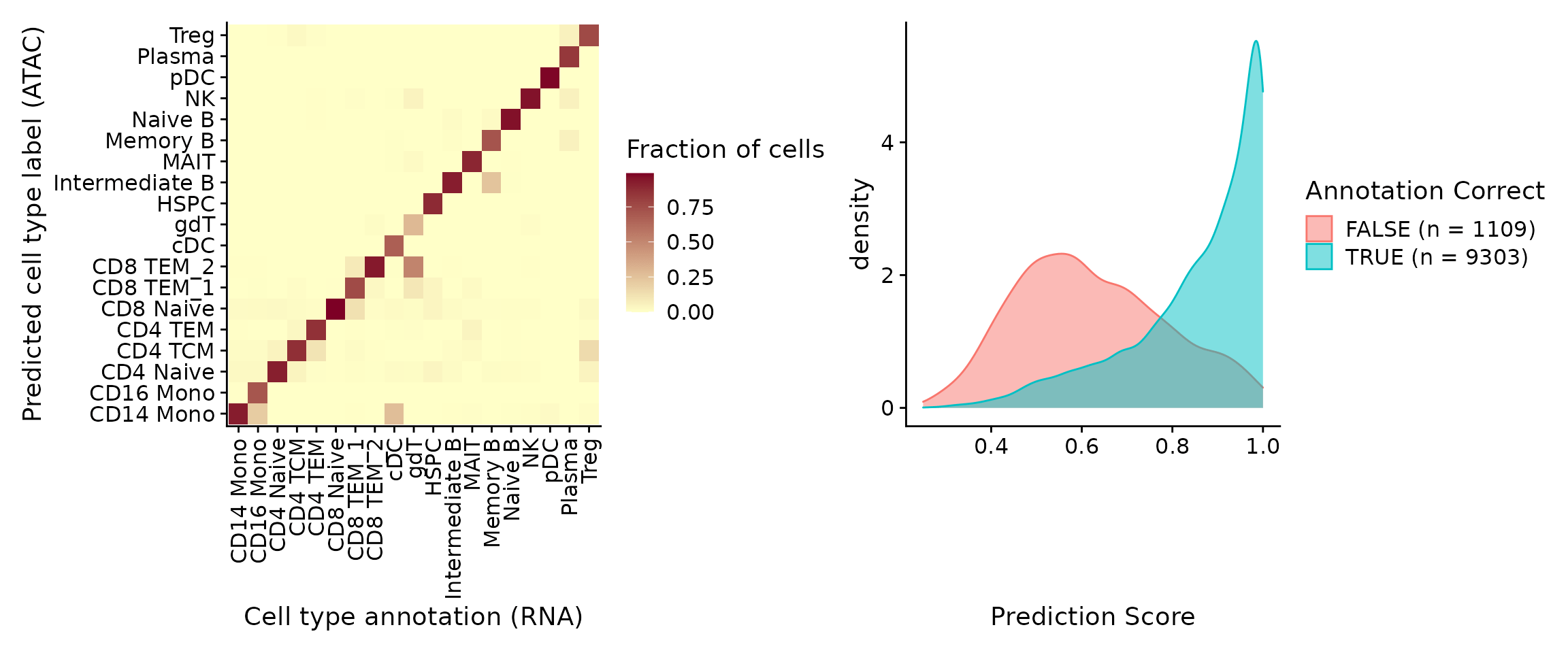
在此示例中,scATAC-seq profile 的注释通过 scRNA-seq 整合正确预测约 90%。此外,prediction.score.max 字段量化了与我们预测注释相关的不确定性。我们可以看到,正确注释的细胞通常与高预测分数相关(>90%),而注释错误的细胞与极低的预测分数相关(<50%)。错误的分配也往往反映密切相关的细胞类型(i.e. Intermediate vs. Naive B cells)。
predictions <- table(pbmc.atac$seurat_annotations, pbmc.atac$predicted.id)
# 对每种细胞类型中的细胞数量进行归一化
predictions <- predictions/rowSums(predictions)
predictions <- as.data.frame(predictions)
p1 <- ggplot(predictions, aes(Var1, Var2, fill = Freq)) +
geom_tile() +
scale_fill_gradient(name = "Fraction of cells",
low = "#ffffc8", high = "#7d0025") +
xlab("Cell type annotation (RNA)") +
ylab("Predicted cell type label (ATAC)") +
theme(axis.text.x = element_text(angle = 90, vjust = 0.5, hjust = 1))
correct <- length(which(pbmc.atac$seurat_annotations == pbmc.atac$predicted.id))
incorrect <- length(which(pbmc.atac$seurat_annotations != pbmc.atac$predicted.id))
data <- FetchData(pbmc.atac, vars = c("prediction.score.max", "annotation_correct"))
p2 <- ggplot(data, aes(prediction.score.max, fill = annotation_correct,
colour = annotation_correct)) +
geom_density(alpha = 0.5) +
theme_cowplot() +
scale_fill_discrete(name = "Annotation Correct",
labels = c(paste0("FALSE (n = ", incorrect, ")"),
paste0("TRUE (n = ", correct, ")"))) +
scale_color_discrete(name = "Annotation Correct",
labels = c(paste0("FALSE (n = ", incorrect, ")"),
paste0("TRUE (n = ", correct, ")"))) +
xlab("Prediction Score")
p1 + p2
4 共同嵌入 scRNA-seq 和 scATAC-seq 数据集
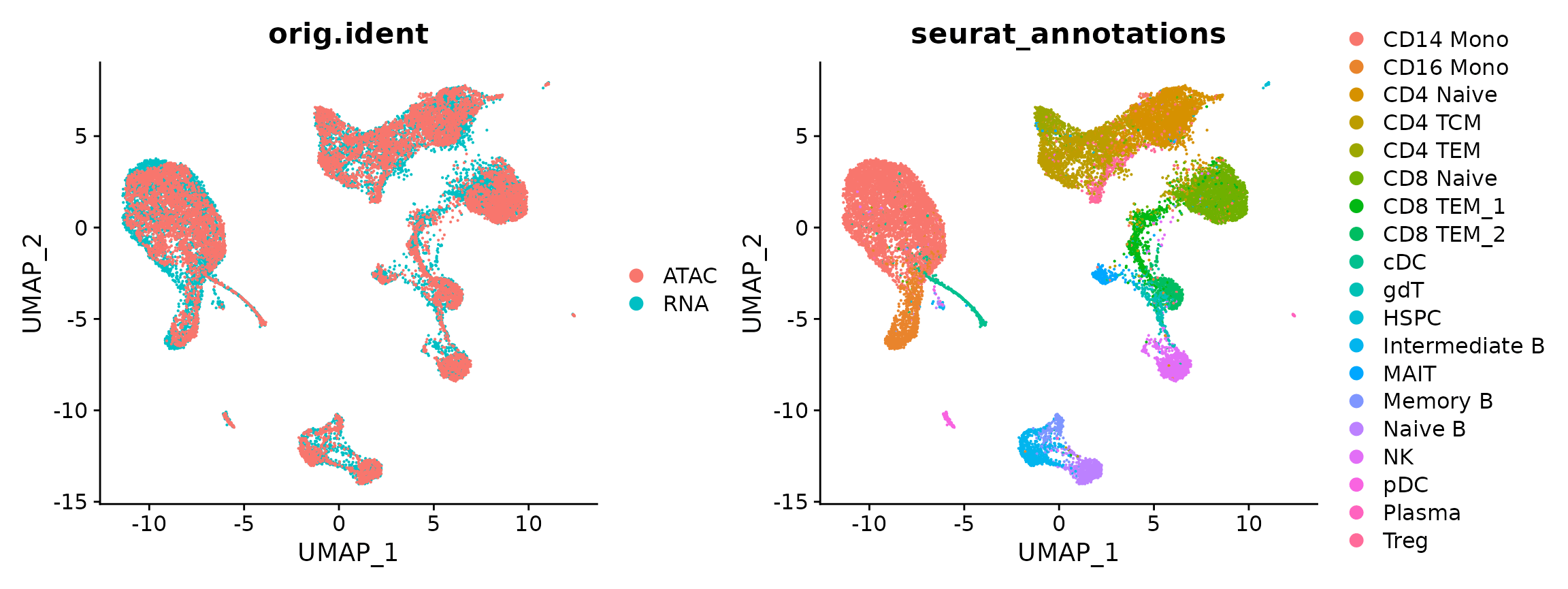
除了跨模态 transferring 标签之外,还可以在同一图上可视化 scRNA-seq 和 scATAC-seq 细胞。我们强调,这一步主要是为了可视化,是一个可选步骤。通常,当我们在 scRNA-seq 和 scATAC-seq 数据集之间进行整合分析时,我们主要关注如上所述的标签 transfer。我们在下面展示了我们的共嵌入工作流程,并再次强调这是出于演示目的,特别是在这种特殊情况下,scRNA-seq profiles 和 scATAC-seq profiles 实际上是在同一细胞中测量的。
为了执行 co-embedding,我们首先根据之前计算的 anchors 将 RNA expression 'impute' 到 scATAC-seq 细胞中,然后合并数据集。
# 请注意,我们将 imputation 限制为 scRNA-seq 中的可变基因,但可以 impute 完整的转录组(如果我们愿意的话)
genes.use <- VariableFeatures(pbmc.rna)
refdata <- GetAssayData(pbmc.rna, assay = "RNA", slot = "data")[genes.use, ]
# refdata(输入)包含 scRNA-seq 细胞的 scRNA-seq 表达矩阵。
# imputation(输出)将包含每个 ATAC 细胞的估算 scRNA-seq 矩阵。
imputation <- TransferData(anchorset = transfer.anchors, refdata = refdata,
weight.reduction = pbmc.atac[["lsi"]], dims = 2:30)
pbmc.atac[["RNA"]] <- imputation
coembed <- merge(x = pbmc.rna, y = pbmc.atac)
# 最后,我们在这个组合对象上运行 PCA 和 UMAP,以可视化两者的 co-embedding 数据集
coembed <- ScaleData(coembed, features = genes.use, do.scale = FALSE)
coembed <- RunPCA(coembed, features = genes.use, verbose = FALSE)
coembed <- RunUMAP(coembed, dims = 1:30)
DimPlot(coembed, group.by = c("orig.ident", "seurat_annotations"))
本文由mdnice多平台发布