论文研读_协方差矩阵自适应演化
根据代码,可以看出主要包含以下几个模块:
- 初始化模块:定义优化函数、问题维度、初始点、步长等参数的初始化。
- 生成模块:随机生成λ个后代样本。
- 选择模块:根据适应度对后代进行排序,选择较好的μ个后代进行重组,得到新的均值。
- 更新模块:更新协方差矩阵、进化路径、步长等自适应策略参数。
- 判断模块:判断是否达到停止条件,否则返回生成模块。
- 输出模块:输出结果。
其中核心是生成-选择-更新的循环过程,实现自适应进化策略,通过协方差矩阵自适应和进化路径累积来自适应样本分布,逐步向优化目标逼近。各模块协同工作,使算法可以有效地搜索高维空间,实现对复杂多模函数的优化。
决策变量是如何分组的?
不需要对决策变量进行特定的分组操作
在每一代中,CMA-ES生成一组新的候选解,这一组解是通过当前的多元正态分布进行采样得到的。每一组解中的每一个个体(解)都是对全部决策变量的一个可能取值。然后根据这一组候选解在目标函数上的性能,对多元正态分布的均值和协方差矩阵进行更新。这样,CMA-ES实际上是在整个搜索空间中同时考虑所有的决策变量,而非将决策变量分组处理。
步长

- 单个步骤相互抵消,因此演化路径较短 σ减小
- 单个步骤不相关
- 单个步骤指向相同的方向,因此进化路径很长 σ增加
为了评估当前的步长是否合适,CMA-ES 通过将连续的移动步长序列相加构建了一个演化路径
通过比较该路径与随机选择(意味着每一步之间是不相关的)状态下期望会生成的路径长度,我们可以相应地调整σ
代码中的步长控制代码如下所示:
sigma = sigma * exp((cs/damps)*(norm(ps)/chiN - 1));
该代码用于更新步长(标准差)的值。sigma表示当前的步长,ps是进化路径,chiN是期望值的常数。通过计算进化路径的范数与期望值的比例,来调整步长的大小。
协方差矩阵
在CMA-ES算法中,Rank-one-Update和Rank-μ-Update是两种不同的协方差矩阵更新方法。
- Rank-one-Update
这是最简单的协方差矩阵更新方法。它只利用种群中最好的一个个体来更新协方差矩阵。
具体做法是利用最好个体与均值的差向量来调整协方差矩阵,使协方差矩阵朝该差向量的方向变化。这可以加速收敛速度。
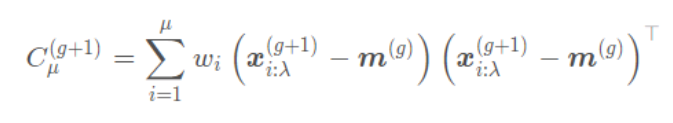
- Rank-μ-Update
它利用种群中多个最好个体来更新协方差矩阵。
具体是使用种群中μ个最好个体的加权平均与均值的差向量来调整协方差矩阵。因为利用了更多信息,它可以使协方差矩阵更稳定可靠。
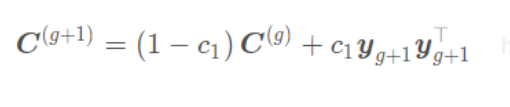
两者的区别在于使用的信息量不同。Rank-one-Update仅用一个个体,收敛速度快但不稳定。Rank-μ-Update利用多个个体,收敛稳定但速度较慢。CMA-ES会结合使用它们,先采用Rank-one-Update加速收敛,当接近最优点时再切换到Rank-μ-Update增加稳定性。这结合了两者的优点。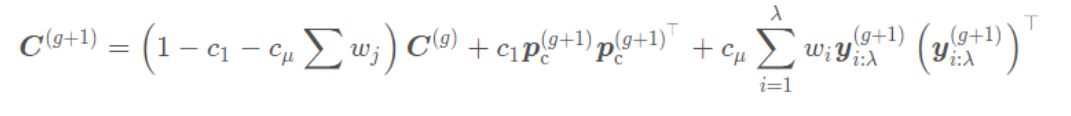

协方差矩阵相关的代码如下所示:
% Cumulation: Update evolution paths
ps = (1-cs) * ps ...
+ sqrt(cs*(2-cs)*mueff) * invsqrtC * (xmean-xold) / sigma;
hsig = sum(ps.^2)/(1-(1-cs)^(2*counteval/lambda))/N < 2 + 4/(N+1);
pc = (1-cc) * pc ...
+ hsig * sqrt(cc*(2-cc)*mueff) * (xmean-xold) / sigma;
% Adapt covariance matrix C
artmp = (1/sigma) * (arx(:,arindex(1:mu)) - repmat(xold,1,mu)); % mu difference vectors
C = (1-c1-cmu) * C ... % regard old matrix
+ c1 * (pc * pc' ... % plus rank one update
+ (1-hsig) * cc*(2-cc) * C) ... % minor correction if hsig==0
+ cmu * artmp * diag(weights) * artmp'; % plus rank mu update
ps和pc分别表示进化路径的两个部分,通过它们的线性组合来更新协方差矩阵。artmp是计算后代样本与当前均值的差异,用于进行协方差矩阵的更新。整个过程包括了对协方差矩阵的rank-one和rank-mu更新。
均值与迭代方向
均值表示种群的分布中心,它反映了种群中所有个体的平均位置,均值受两个因素影响:
- 初始均值
- 种群分布的演化
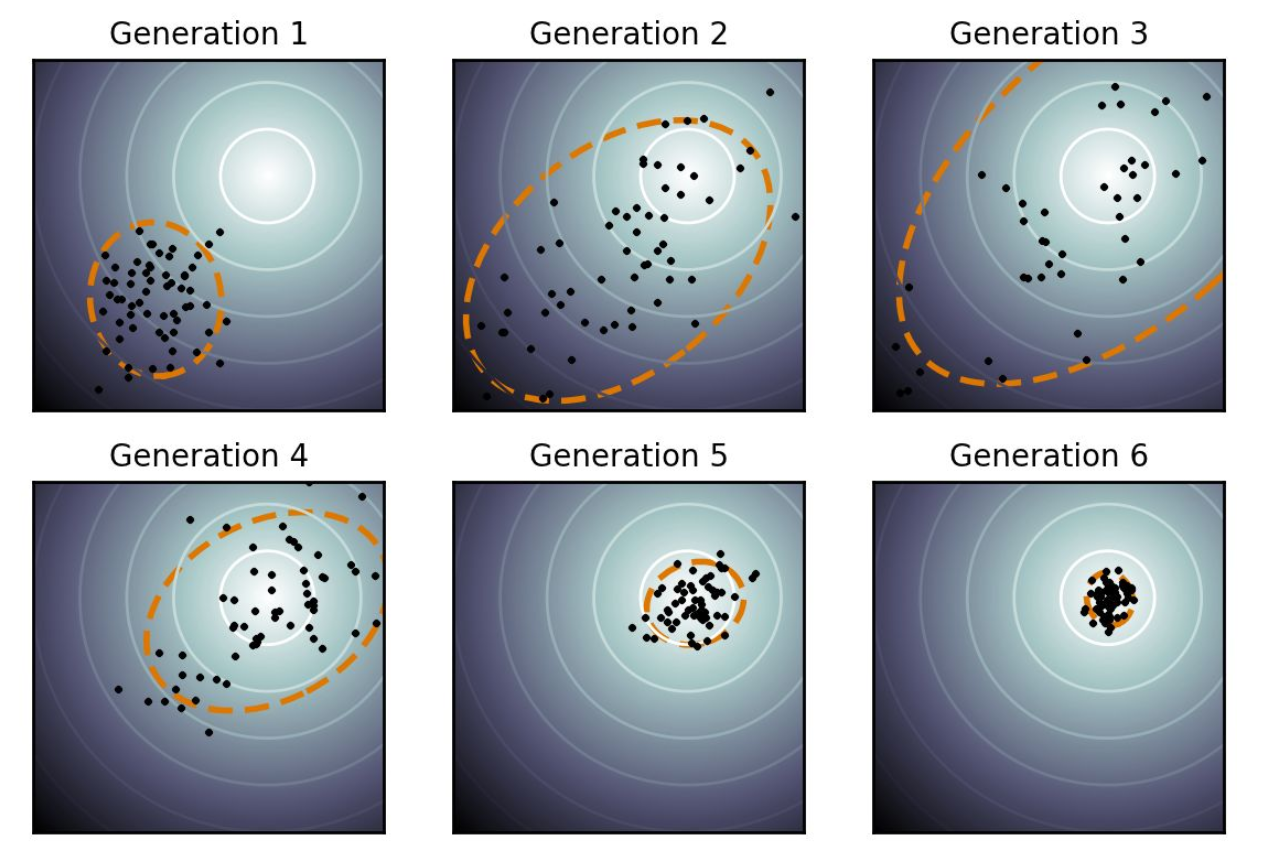
CMA-ES估计协方差时用第2代均值,基于g代均值得到协方差矩阵,然后采样得g+1代个体及g+1代个体的分布(未使用g+1代均值,分布已发生变化)
先根据一个原始的均值得到一个分布,再根据这个分布采样个体,生成新的子代(这算是一次迭代),然后下个阶段计算均值
这里可以看到整个种群往上偏移,所以均值在上面,然后在红点位置继续采样
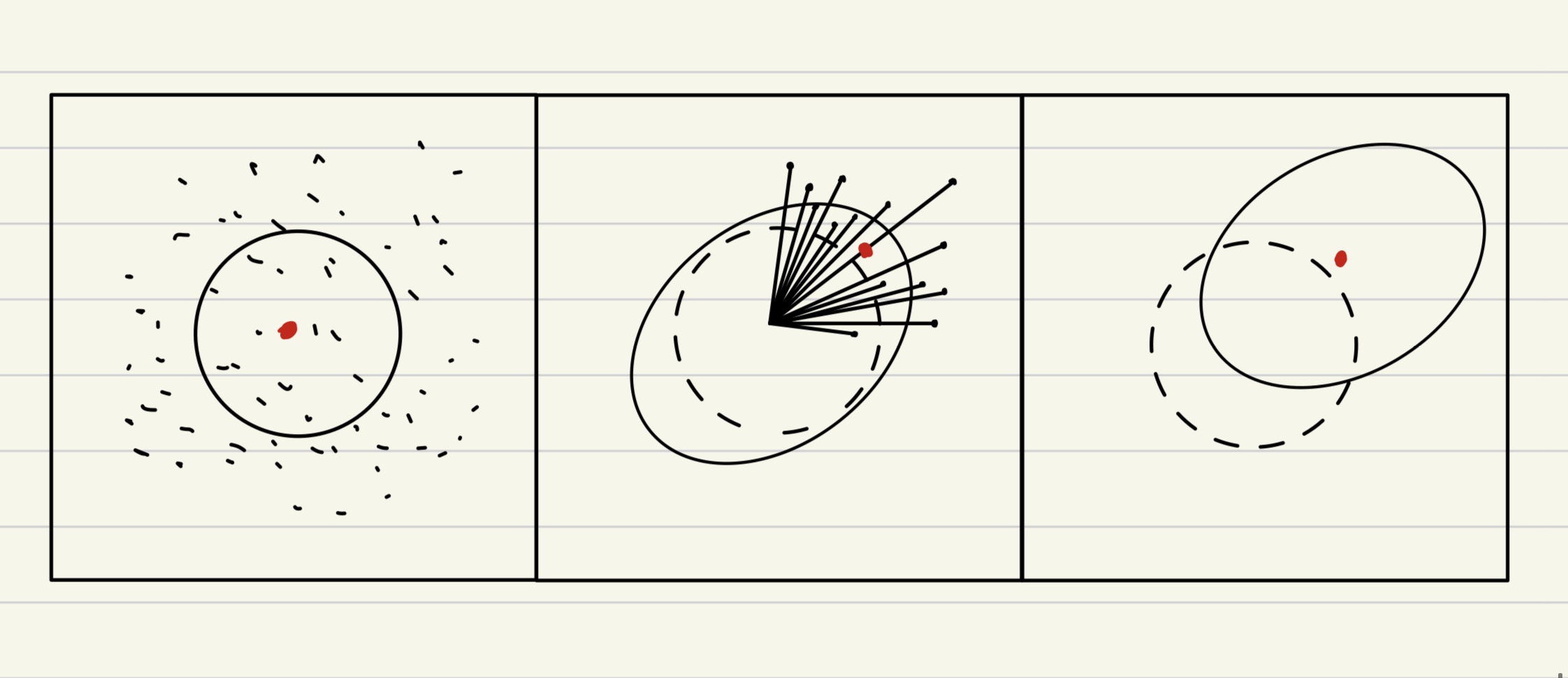
利用旧均值生成新个体
最近几代的协方差矩阵信息重要程度比前面多代信息高,比重在65%左右
蓝圈是种群的分布,可能几次迭代后,分布没有什么变化

下图是上面两张图的结合
终止条件
1.判断适应度是否小于设定值1e-10
2.判断是否达到最大迭代次数1e3*N^2
初始化
function xmin=purecmaes
% -------------------- Initialization --------------------------------
% User defined input parameters (need to be edited)
strfitnessfct = 'fsphere'; %优化的函数三维球体name of objective/fitness function
N = 3; % 三维球体number of objective variables/problem dimension
xmean = rand(N,1); % 初始化均值 objective variables initial point
sigma = 0.5; % 初始化标准差 coordinate wise standard deviation (step size)
stopfitness = 1e-10; % 停止优化指标stop if fitness < stopfitness (minimization)
stopeval = 1e3*N^2; % 迭代次数最大值stop after stopeval number of function evaluations
% Strategy parameter setting: Selection
lambda = 4+floor(3*log(N)); %后代数量 population size, offspring number
mu = lambda/2; % number of parents/points for recombination
weights = log(mu+1/2)-log(1:mu)'; % muXone array for weighted recombination
mu = floor(mu);
weights = weights/sum(weights); % normalize recombination weights array
mueff=sum(weights)^2/sum(weights.^2); %后代方差有效数量 variance-effectiveness of sum w_i x_i
% Strategy parameter setting: Adaptation
cc = (4 + mueff/N) / (N+4 + 2*mueff/N); % time constant for cumulation for C
cs = (mueff+2) / (N+mueff+5); % t-const for cumulation for sigma control
c1 = 2 / ((N+1.3)^2+mueff); %rank-one的学习率 learning rate for rank-one update of C
cmu = min(1-c1, 2 * (mueff-2+1/mueff) / ((N+2)^2+mueff)); % rank-mu的学习率 for rank-mu update
damps = 1 + 2*max(0, sqrt((mueff-1)/(N+1))-1) + cs; % damping for sigma
% usually close to 1
% Initialize dynamic (internal) strategy parameters and constants
pc = zeros(N,1); ps = zeros(N,1); % evolution paths for C and sigma
B = eye(N,N); % B defines the coordinate system
D = ones(N,1); % diagonal D defines the scaling
C = B * diag(D.^2) * B'; % covariance matrix C
invsqrtC = B * diag(D.^-1) * B'; % C^-1/2
eigeneval = 0; % track update of B and D
chiN=N^0.5*(1-1/(4*N)+1/(21*N^2)); % expectation of
% ||N(0,I)|| == norm(randn(N,1))
out.dat = []; out.datx = []; % for plotting output
i=0;
生成种群
% -------------------- Generation Loop --------------------------------
counteval = 0; % the next 40 lines contain the 20 lines of interesting code
while counteval < stopeval
i=i+1;
% Generate and evaluate lambda offspring(随机产生后代)
for k=1:lambda,
arx(:,k) = xmean + sigma * B * (D .* randn(N,1)); % m + sig * Normal(0,C)
arfitness(k) = feval(strfitnessfct, arx(:,k)); % objective function call
counteval = counteval+1;
end
% Sort by fitness and compute weighted mean into xmean(排序,选择值较小的采样点)
[arfitness, arindex] = sort(arfitness); % minimization
xold = xmean;
xmean = arx(:,arindex(1:mu)) * weights; % recombination, new mean value(新的均值)
% Cumulation: Update evolution paths(更新进化路径,在协方差矩阵更新的时候利用,协方差更新的方法:rank one 和rank U 融合的方式)
ps = (1-cs) * ps ...
+ sqrt(cs*(2-cs)*mueff) * invsqrtC * (xmean-xold) / sigma;
hsig = sum(ps.^2)/(1-(1-cs)^(2*counteval/lambda))/N < 2 + 4/(N+1);
pc = (1-cc) * pc ...
+ hsig * sqrt(cc*(2-cc)*mueff) * (xmean-xold) / sigma;
% Adapt covariance matrix C(更新协方差矩阵)
artmp = (1/sigma) * (arx(:,arindex(1:mu)) - repmat(xold,1,mu)); % mu difference vectors
C = (1-c1-cmu) * C ... % regard old matrix
+ c1 * (pc * pc' ... % plus rank one update
+ (1-hsig) * cc*(2-cc) * C) ... % minor correction if hsig==0
+ cmu * artmp * diag(weights) * artmp'; % plus rank mu update
% Adapt step size sigma更新步长
sigma = sigma * exp((cs/damps)*(norm(ps)/chiN - 1));
% Update B and D from C
if counteval - eigeneval > lambda/(c1+cmu)/N/10 % to achieve O(N^2)
eigeneval = counteval;
C = triu(C) + triu(C,1)'; % enforce symmetry
[B,D] = eig(C); % eigen decomposition, B==normalized eigenvectors
D = sqrt(diag(D)); % D contains standard deviations now
invsqrtC = B * diag(D.^-1) * B';
end
% Break, if fitness is good enough or condition exceeds 1e14, better termination methods are advisable
if arfitness(1) <= stopfitness || max(D) > 1e7 * min(D)
break;
end
% Output
more off; % turn pagination off in Octave
disp([num2str(counteval) ': ' num2str(arfitness(1)) ' ' ...
num2str(sigma*sqrt(max(diag(C)))) ' ' ...
num2str(max(D) / min(D))]);
% with long runs, the next line becomes time consuming
out.dat = [out.dat; arfitness(1) sigma 1e5*D' ];
out.datx = [out.datx; xmean'];
end % while, end generation loop
% ------------- Final Message and Plotting Figures --------------------
disp([num2str(counteval) ': ' num2str(arfitness(1))]);
xmin = arx(:, arindex(1)); % Return best point of last iteration.
% Notice that xmean is expected to be even
% better.
figure(1); hold off; semilogy(abs(out.dat)); % abs for negative fitness
figure(2);
semilogy(out.dat(:,1) - min(out.dat(:,1)), 'k-'); % difference to best ever fitness, zero is not displayed
title('fitness, sigma, sqrt(eigenvalues)'); grid on; xlabel('iteration');
figure(3); hold off; plot(out.datx);
title('Distribution Mean'); grid on; xlabel('iteration')
figure(4)
plot3(out.datx(1,1),out.datx(1,2),out.datx(1,3),'*')
hold on
plot3(out.datx(:,1),out.datx(:,2),out.datx(:,3))
% ---------------------------------------------------------------
function f=frosenbrock(x)
if size(x,1) < 2 error('dimension must be greater one'); end
f = 100*sum((x(1:end-1).^2 - x(2:end)).^2) + sum((x(1:end-1)-1).^2);
function f=fsphere(x)
f=sum(x.^2);
function f=fssphere(x)
f=sqrt(sum(x.^2));
function f=fschwefel(x)
f = 0;
for i = 1:size(x,1),
f = f+sum(x(1:i))^2;
end
function f=fcigar(x)
f = x(1)^2 + 1e6*sum(x(2:end).^2);
function f=fcigtab(x)
f = x(1)^2 + 1e8*x(end)^2 + 1e4*sum(x(2:(end-1)).^2);
function f=ftablet(x)
f = 1e6*x(1)^2 + sum(x(2:end).^2);
function f=felli(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=1e6.^((0:N-1)/(N-1)) * x.^2;
function f=felli100(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=1e4.^((0:N-1)/(N-1)) * x.^2;
function f=fplane(x)
f=x(1);
function f=ftwoaxes(x)
f = sum(x(1:floor(end/2)).^2) + 1e6*sum(x(floor(1+end/2):end).^2);
function f=fparabR(x)
f = -x(1) + 100*sum(x(2:end).^2);
function f=fsharpR(x)
f = -x(1) + 100*norm(x(2:end));
function f=fdiffpow(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=sum(abs(x).^(2+10*(0:N-1)'/(N-1)));
function f=frastrigin10(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
scale=10.^((0:N-1)'/(N-1));
f = 10*size(x,1) + sum((scale.*x).^2 - 10*cos(2*pi*(scale.*x)));
function f=frand(x)
f=rand;% ---------------------------------------------------------------
function f=frosenbrock(x)
if size(x,1) < 2 error('dimension must be greater one'); end
f = 100*sum((x(1:end-1).^2 - x(2:end)).^2) + sum((x(1:end-1)-1).^2);
function f=fsphere(x)
f=sum(x.^2);
function f=fssphere(x)
f=sqrt(sum(x.^2));
function f=fschwefel(x)
f = 0;
for i = 1:size(x,1),
f = f+sum(x(1:i))^2;
end
function f=fcigar(x)
f = x(1)^2 + 1e6*sum(x(2:end).^2);
function f=fcigtab(x)
f = x(1)^2 + 1e8*x(end)^2 + 1e4*sum(x(2:(end-1)).^2);
function f=ftablet(x)
f = 1e6*x(1)^2 + sum(x(2:end).^2);
function f=felli(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=1e6.^((0:N-1)/(N-1)) * x.^2;
function f=felli100(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=1e4.^((0:N-1)/(N-1)) * x.^2;
function f=fplane(x)
f=x(1);
function f=ftwoaxes(x)
f = sum(x(1:floor(end/2)).^2) + 1e6*sum(x(floor(1+end/2):end).^2);
function f=fparabR(x)
f = -x(1) + 100*sum(x(2:end).^2);
function f=fsharpR(x)
f = -x(1) + 100*norm(x(2:end));
function f=fdiffpow(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
f=sum(abs(x).^(2+10*(0:N-1)'/(N-1)));
function f=frastrigin10(x)
N = size(x,1); if N < 2 error('dimension must be greater one'); end
scale=10.^((0:N-1)'/(N-1));
f = 10*size(x,1) + sum((scale.*x).^2 - 10*cos(2*pi*(scale.*x)));
function f=frand(x)
f=rand;
参考文献:
https://blog.csdn.net/qq_40206371/article/details/117091441
https://blog.csdn.net/hba646333407/article/details/108836648