写在前面
学习一个软件最好的方法就是啃它的官方文档。本着自己学习、分享他人的态度,分享官方文档的中文教程。软件可能随时更新,建议配合官方文档一起阅读。推荐先按顺序阅读往期内容:
文献篇:
1.文献阅读:(Seurat V1) 单细胞基因表达数据的空间重建
2.文献阅读:(Seurat V2) 整合跨越不同条件、技术、物种的单细胞转录组数据
3.文献阅读:(Seurat V3) 单细胞数据综合整合
4.文献阅读:(Seurat V4) 整合分析多模态单细胞数据
5.文献阅读:(Seurat V5) 用于集成、多模态和可扩展单细胞分析的字典学习
教程篇:
1.Seurat Tutorial 1:常见分析工作流程,基于 PBMC 3K 数据集
2.Seurat Tutorial 2:使用 Seurat 分析多模态数据
3.Seurat Tutorial 3:scRNA-seq 整合分析介绍
::: block-1
目录
- 1 单细胞参考映射介绍
- 2 数据集预处理
- 3 三个胰岛细胞数据集的整合
- 4 使用整合参考进行细胞类型分类
- 5 单峰 UMAP 投影
:::
官网教程:
https://satijalab.org/seurat/articles/integration_mapping
1 单细胞参考映射介绍
在本节,我们首先构建一个 integrated reference,然后演示如何利用这个 reference 来注释新的 query datasets。生成 integrated reference 的详细步骤见 Introduction to scRNA-seq integration。生成后,此 reference 可用于通过细胞类型标签传输和将 query cells 投影到 reference UMAP 等任务来分析其他 query datasets。值得注意的是,这不需要矫正原始的 query data,因此如果有高质量的 reference,这可能是一种有效的策略。
2 数据集预处理
出于本示例的目的,我们选择了通过四种技术产生的人类胰岛细胞数据集,即 CelSeq (GSE81076)、CelSeq2 (GSE85241)、Fluidigm C1 (GSE86469), and SMART-Seq2 (E-MTAB-5061)。为方便起见,我们通过 SeuratData 包分发此数据集。metadata 包含四个数据集中每个细胞的技术(tech column)和细胞类型注释(celltype column)。
library(Seurat)
library(SeuratData)
InstallData("panc8")
为了构建 reference,我们将识别各个数据集之间的 'anchors'。首先,我们将组合对象拆分为一个 list,每个数据集作为一个元素(这只是必要的,因为数据被捆绑在一起以便于分发)。
data("panc8")
pancreas.list <- SplitObject(panc8, split.by = "tech")
pancreas.list <- pancreas.list[c("celseq", "celseq2", "fluidigmc1", "smartseq2")]
pancreas.list
# $celseq
# An object of class Seurat
# 34363 features across 1004 samples within 1 assay
# Active assay: RNA (34363 features, 0 variable features)
# $celseq2
# An object of class Seurat
# 34363 features across 2285 samples within 1 assay
# Active assay: RNA (34363 features, 0 variable features)
# $fluidigmc1
# An object of class Seurat
# 34363 features across 638 samples within 1 assay
# Active assay: RNA (34363 features, 0 variable features)
# $smartseq2
# An object of class Seurat
# 34363 features across 2394 samples within 1 assay
# Active assay: RNA (34363 features, 0 variable features)
在找到 anchors 之前,我们执行标准预处理(log-normalization),并为每个 anchors 单独识别可变特征。请注意,Seurat 实现了一种改进的基于方差稳定变换(vst)的变量特征选择方法。
for (i in 1:length(pancreas.list)) {
pancreas.list[[i]] <- NormalizeData(pancreas.list[[i]], verbose = FALSE)
pancreas.list[[i]] <- FindVariableFeatures(pancreas.list[[i]], selection.method = "vst", nfeatures = 2000, verbose = FALSE)
}
3 三个胰岛细胞数据集的整合
接下来,我们使用 FindIntegrationAnchors() 函数识别 anchors,该函数将 Seurat 对象列表作为输入。在这里,我们将其中三个对象整合到一个 reference 中(稍后我们将在本小节中使用第四个对象作为查询数据集来演示映射)。
- 我们在这里使用所有默认参数来识别 anchors,包括数据集的“维度”(30;随意尝试在很宽的范围内更改此参数,例如在 10 到 50 之间)。
reference.list <- pancreas.list[c("celseq", "celseq2", "smartseq2")]
pancreas.anchors <- FindIntegrationAnchors(object.list = reference.list, dims = 1:30)
然后我们将这些 anchors 传递给 IntegrateData() 函数,该函数返回一个 Seurat 对象。
- 返回的对象将包含一个新的
Assay,其中包含所有细胞的 integrated(or ‘batch-corrected’)表达矩阵,使它们能够被联合分析。
pancreas.integrated <- IntegrateData(anchorset = pancreas.anchors, dims = 1:30)
运行 IntegrateData() 后,Seurat 对象将包含一个带有 integrated 表达矩阵的新 Assay。请注意,原始(未校正的值)仍存储在对象的“RNA”assay 中,因此您可以来回切换。
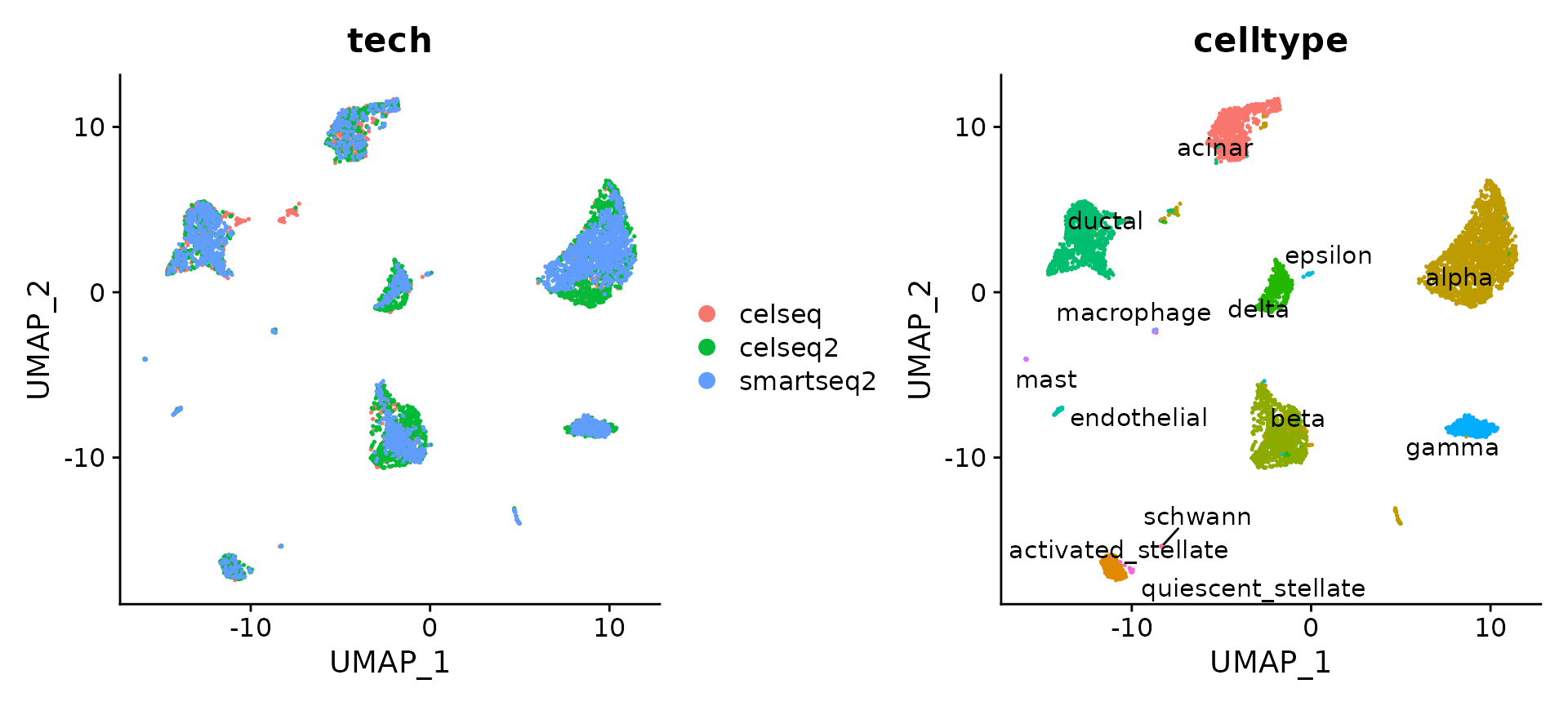
然后我们可以使用这个新的 integrated 矩阵进行下游分析和可视化。在这里,我们 scale integrated 数据,运行 PCA,并使用 UMAP 可视化结果。integrated 的数据集按细胞类型而不是技术进行聚类。
library(ggplot2)
library(cowplot)
library(patchwork)
# 切换到 integrated assay。该 assay 的可变特征在整合数据过程中自动设置
DefaultAssay(pancreas.integrated) <- "integrated"
# 运行可视化和聚类的标准工作流程
pancreas.integrated <- ScaleData(pancreas.integrated, verbose = FALSE)
pancreas.integrated <- RunPCA(pancreas.integrated, npcs = 30, verbose = FALSE)
pancreas.integrated <- RunUMAP(pancreas.integrated, reduction = "pca", dims = 1:30, verbose = FALSE)
p1 <- DimPlot(pancreas.integrated, reduction = "umap", group.by = "tech")
p2 <- DimPlot(pancreas.integrated, reduction = "umap", group.by = "celltype", label = TRUE, repel = TRUE) +
NoLegend()
p1 + p2
4 使用整合参考进行细胞类型分类
Seurat 还支持将 reference data(or meta data)投射到 query object。虽然许多方法是保守的(两个过程都从识别 anchors 开始),但数据 transfer 和 integration 之间有两个重要区别:
- In data transfer, Seurat 不会更正或修改 query expression data。
- In data transfer, Seurat 有一个选项(set by default)将 reference 的 PCA 结构投射到 query 上,而不是使用 CCA 学习联合结构。我们通常建议在 scRNA-seq 数据集之间投射数据时使用此选项。
找到 anchors 后,我们使用 TransferData() 函数根据 reference data(参考细胞类型标签的向量)对 query cells 进行分类。TransferData() 返回一个包含预测 ID 和预测分数的矩阵,我们可以将其添加到 query metadata 中。
pancreas.query <- pancreas.list[["fluidigmc1"]]
pancreas.anchors <- FindTransferAnchors(reference = pancreas.integrated, query = pancreas.query, dims = 1:30, reference.reduction = "pca")
predictions <- TransferData(anchorset = pancreas.anchors, refdata = pancreas.integrated$celltype, dims = 1:30)
pancreas.query <- AddMetaData(pancreas.query, metadata = predictions)
因为我们有来自完整 integrated 分析的原始标签注释,所以我们可以评估我们预测的细胞类型注释与完整参考的匹配程度。在这个例子中,我们发现细胞类型分类的一致性很高,超过 96% 的细胞被正确标记。
pancreas.query$prediction.match <- pancreas.query$predicted.id == pancreas.query$celltype
table(pancreas.query$prediction.match)
##
## FALSE TRUE
## 21 617
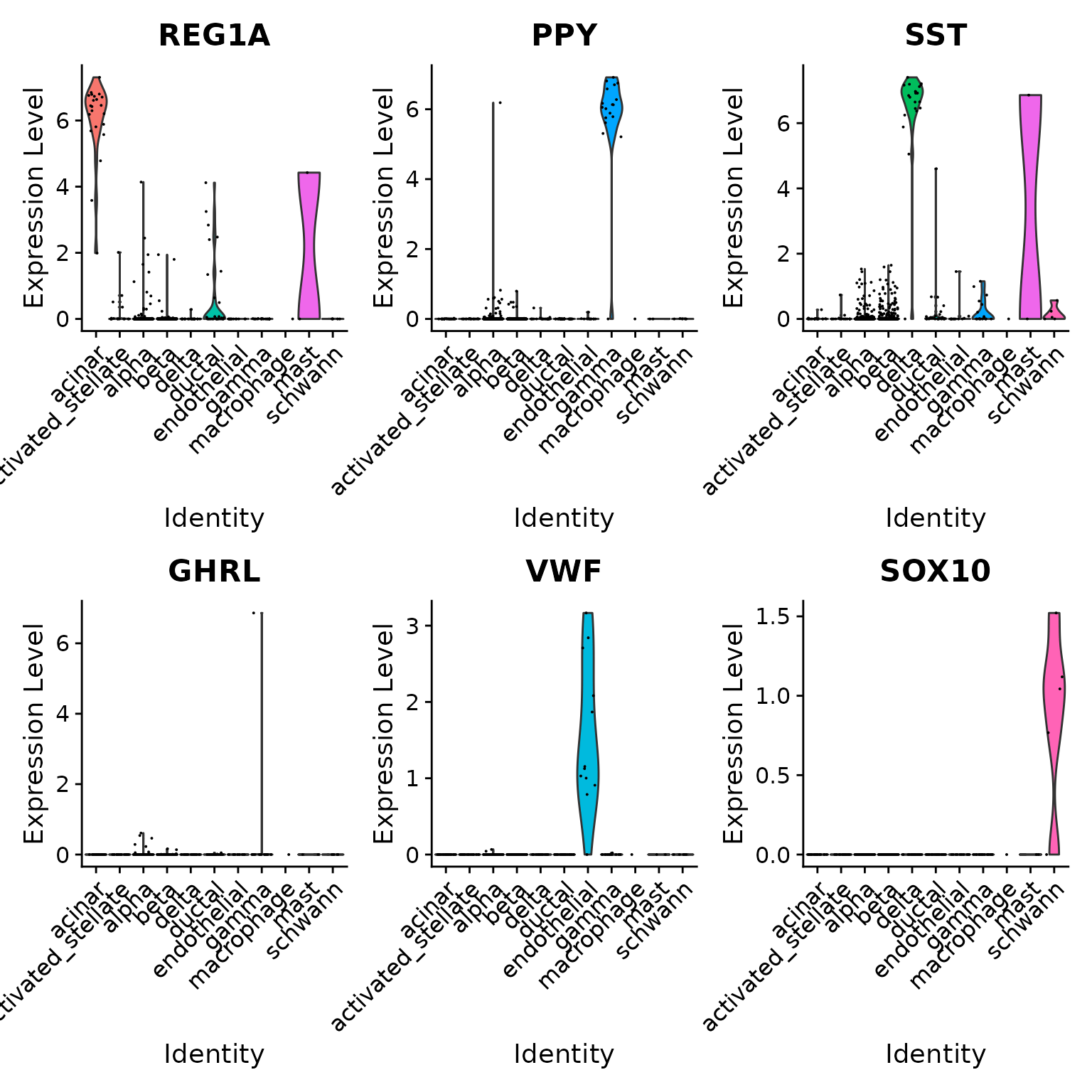
为了进一步验证这一点,我们可以检查特定胰岛细胞群的一些典型细胞类型标记。请注意,即使其中一些细胞类型仅由一两个细胞表示(e.g. epsilon cells),我们仍然能够正确分类它们。
table(pancreas.query$predicted.id)
##
## acinar activated_stellate alpha beta
## 22 17 253 256
## delta ductal endothelial gamma
## 22 30 12 18
## macrophage mast schwann
## 1 2 5
VlnPlot(pancreas.query, c("REG1A", "PPY", "SST", "GHRL", "VWF", "SOX10"), group.by = "predicted.id")
5 单峰 UMAP 投影
在 Seurat v4 中,我们还启用了将 query 投影到 reference UMAP 结构上。这可以通过计算 reference UMAP 模型然后调用 MapQuery() 而不是 TransferData() 来实现。
pancreas.integrated <- RunUMAP(pancreas.integrated, dims = 1:30, reduction = "pca", return.model = TRUE)
pancreas.query <- MapQuery(anchorset = pancreas.anchors, reference = pancreas.integrated, query = pancreas.query,
refdata = list(celltype = "celltype"), reference.reduction = "pca", reduction.model = "umap")
MapQuery 在做什么?
MapQuery() 是三个函数的包装:TransferData()、IntegrateEmbeddings() 和 ProjectUMAP()。TransferData() 用于传输细胞类型标签并估算 ADT 值;IntegrateEmbeddings() 用于通过纠正查询的投影低维嵌入来整合 reference 与 query;最后使用 ProjectUMAP() 将查询数据投影到 reference 的 UMAP 结构上。使用中间函数执行此操作的等效代码如下:
pancreas.query <- TransferData(anchorset = pancreas.anchors, reference = pancreas.integrated, query = pancreas.query,
refdata = list(celltype = "celltype"))
pancreas.query <- IntegrateEmbeddings(anchorset = pancreas.anchors, reference = pancreas.integrated,
query = pancreas.query, new.reduction.name = "ref.pca")
pancreas.query <- ProjectUMAP(query = pancreas.query, query.reduction = "ref.pca", reference = pancreas.integrated,
reference.reduction = "pca", reduction.model = "umap")
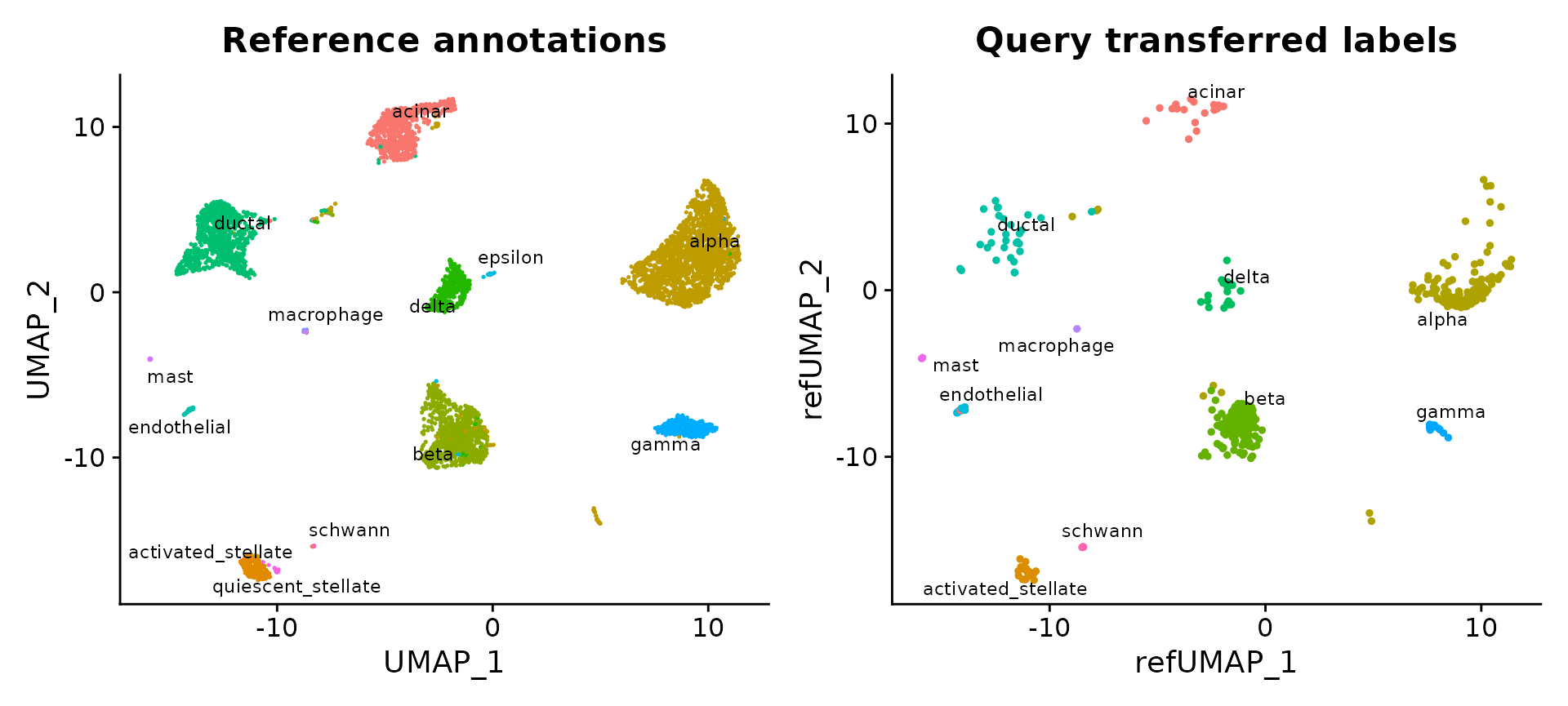
我们现在可以在 reference 旁边可视化 query cells。
p1 <- DimPlot(pancreas.integrated, reduction = "umap", group.by = "celltype", label = TRUE, label.size = 3,repel = TRUE) + NoLegend() + ggtitle("Reference annotations")
p2 <- DimPlot(pancreas.query, reduction = "ref.umap", group.by = "predicted.celltype", label = TRUE, label.size = 3, repel = TRUE) + NoLegend() + ggtitle("Query transferred labels")
p1 + p2

本文由mdnice多平台发布