大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2020年12月31日,美国明尼苏达大学Natalia Y. Tretyakova教授团队在《Int J Mol Sci》杂志发表题为“Multi-Omics Characterization of Inflammatory Bowel Disease-Induced Hyperplasia/Dysplasia in the Rag2-/-/Il10-/- Mouse Model”的研究文章,该研究通过简化基因组甲基化测序(RRBS)+氧化简化基因组甲基化测序(oxRRBS)及对应的转录组测序(RNA-seq)揭示炎症性肠病 (IBD)导致基因组中甲基化和羟甲基化模式变化,表明了IBD导致发育异常的表观遗传机制。

标题:Multi-Omics Characterization of Inflammatory Bowel Disease-Induced Hyperplasia/Dysplasia in the Rag2-/-/Il10-/- Mouse Model (Rag2-/-/Il10-/-小鼠模型中炎症性肠病诱导增生/发育异常的多组学表征)
时间:2020-12-31
期刊:International Journal Of Molecular Sciences
影响因子:IF 6.208
技术平台:RRBS、oxRRBS、RNA-seq、RT-qPCR等
研究摘要:
假设表观遗传失调在炎症性肠病(inflammatory bowel disease,IBD)和结肠癌发展之间的关联中发挥作用。本研究对从肝螺杆菌(H.hepaticus)感染的IBD小鼠模型中收获近端结肠组织,并进行DNA甲基化组、DNA羟甲基化组和转录组分析。RRBS和oxRRBS分析共鉴定出1606个差异甲基化区域(DMR)和3011个差异羟甲基化区域(DhMR),与胃肠疾病、炎性疾病和癌症相关的基因和这些DMR/DhMR重叠。RNA-seq揭示了许多与炎症和癌症相关的基因显著表达变化,包括Duox2、Tgm2、Cdhr5和Hk2在内的几个基因在DNA甲基化/羟甲基化和基因表达水平上均表现出变化。总体而言,本研究结果表明,慢性炎症会引发基因组中DNA甲基化和羟甲基化模式的变化,从而改变关键肿瘤发生基因的表达,并可能导致结直肠癌的发生。
项目设计:
(1)样本选取:
IBD与正常小鼠近侧肠道组织的5mC、5hmC、表达图谱分析
(2)项目设计流程图:
实验结果
(1)肝螺杆菌(H. hepaticus)感染的Rag2-/-/Il10-/-小鼠结肠组织的组织病理学分析
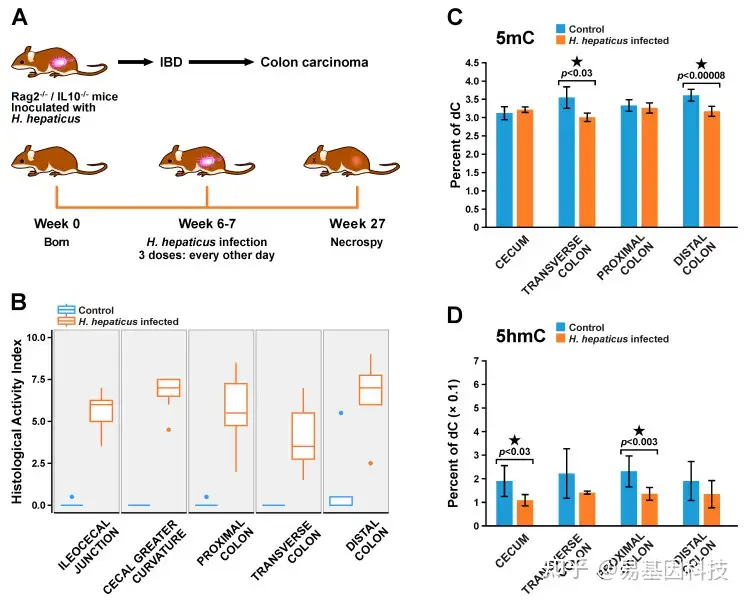
图1:炎症性肠病(IBD)与正常组织的组织病理学检验
- 动物研究设计:Rag2-/-/Il10-/-小鼠在6-7周龄时H.hepaticus感染或无菌培养基处理(对照),并在感染后20周处死
- 感染H.hepaticus(橙色)的Rag2-/-/Il10-/-雄性小鼠和仅用盐水处理的对照小鼠(蓝色)的结肠组织的组织病理学结果:通过对胃肠道不同部位的炎症、水肿、上皮缺陷、隐窝萎缩、增生和发育异常(Dysplasia)的个体评分求和来计算组织学活性指数(n=7)
- 感染或未感染H.hepaticus的Rag2-/-/Il10-/-雄性小鼠胃肠道不同部位的整体5-甲基胞嘧啶(5mC)水平,(盲肠每组n=4,横结肠每组n=3,近端和远端结肠每组n=7)
- 感染或未感染H.hepaticus的Rag2-/-/Il10-/-雄性小鼠胃肠道不同部位的整体5-羟甲基胞嘧啶(5hmC)水平
(2)炎症性肠病(IBD)诱导的增生/发育异常中DNA甲基化和DNA羟甲基化表征
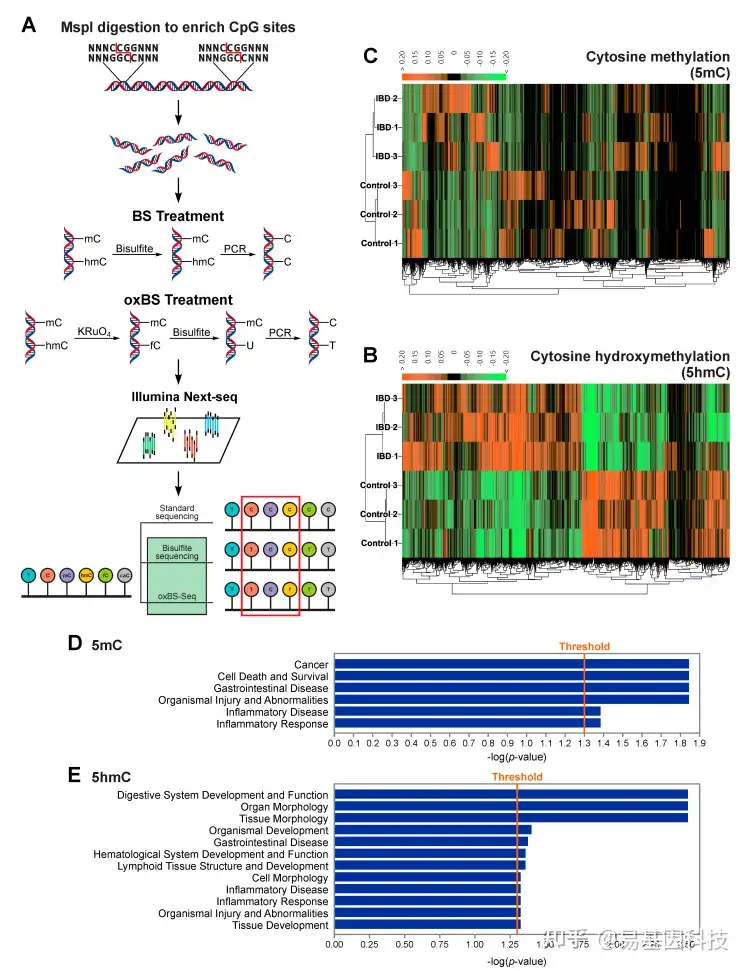
图2:差异甲基化区域(DMR)和差异羟甲基化区域(DhMR)富集的基因在与癌症发生和胃肠疾病相关的基因富集。
- RRBS和oxRRBS技术示意图:通过比较RRBS和oxRRBS数据集分析5mC和5hmC水平。
B-C. 通过RRBS与oxRRBS结合鉴定1606个DMR(B)和3011个DhMR(C)的热图。
D-E. 通过Ingenuity Pathway Analysis(IPA),使用显著性阈值p=0.05的Fisher精确检验,预测受DMR(D)和DhMR(E)基因影响的功能和疾病。
(3)差异甲基化分析揭示IBD诱导的增生/发育异常中存在广泛的DMR和DhMR
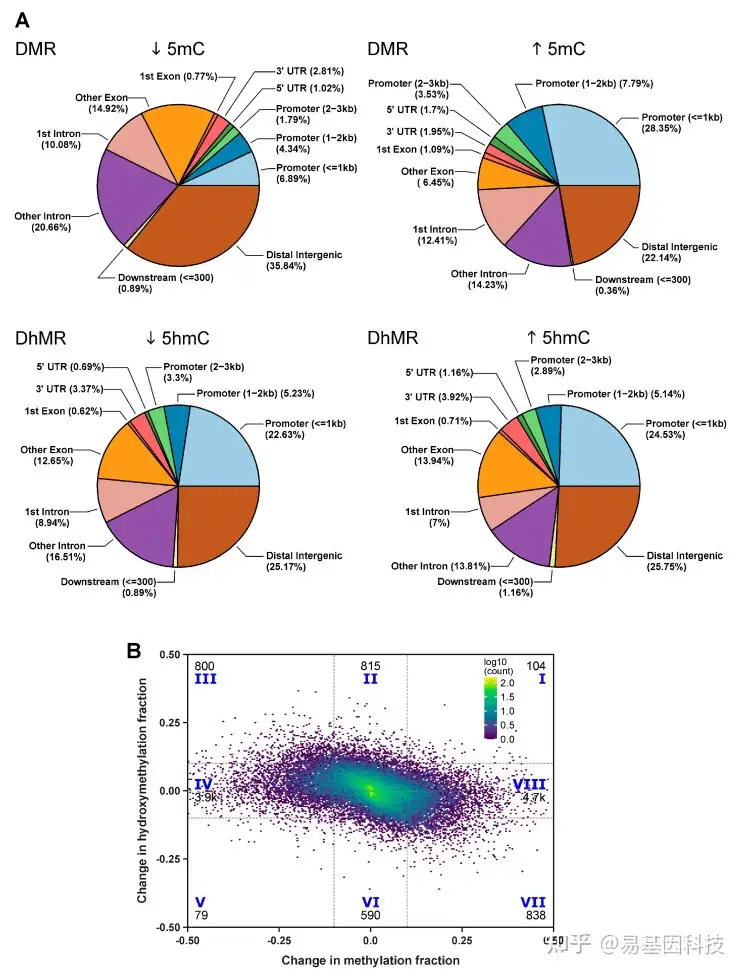
图3:Rag2-/-/Il10-/-小鼠在H.hepaticus诱导炎症在单核苷酸分辨率下的甲基化和羟甲基化动态变化
- 784个低DMR,822个高DMR,1454个低DhMR和1557个高DhMR的基因组分布饼图
- DMR/DhMR内每个CpG位点5mC和5hmC的动态变化。0.1是5mC/5hmC水平显著性变化的截止值。数字代表点数量。
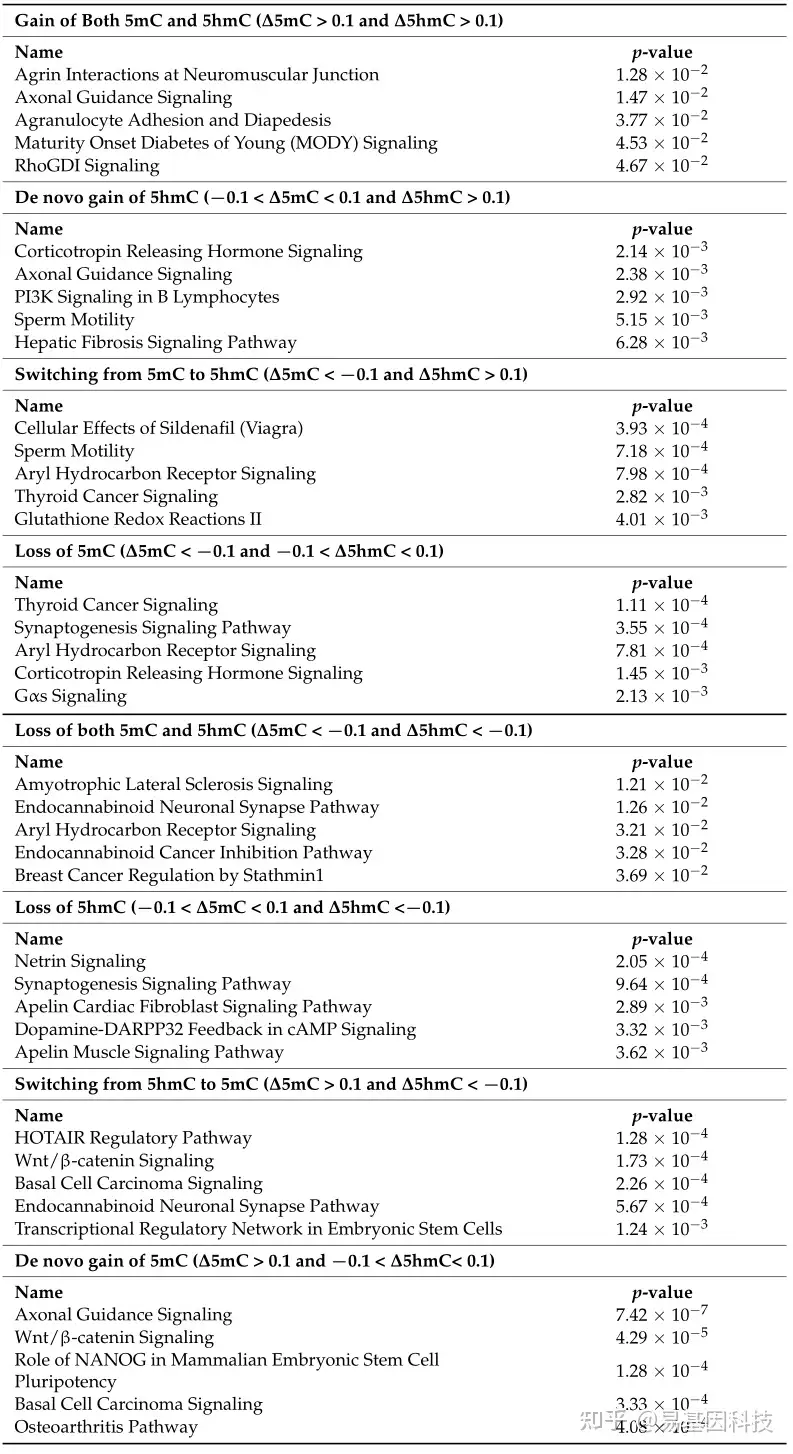
表1:H.hepaticus诱导的Rag2−/−/Il10−/−小鼠炎症CpG位点IPA差异甲基化和羟基甲基化的经典通路
(4)IBD诱导增生/发育异常中的基因表达变化

图4:炎症性肠病(IBD)和对照小鼠中近端结肠的转录组分析
- 基于最高方差的500个基因的无监督分层聚类,显示对照组和IBD组之间明显分离
- IPA鉴定上游调控因子Il10ra周围的网络
- 使用显著性阈值p=0.05的Fisher精确检验,预测受差异表达基因影响的疾病和功能
(5)DNA甲基化异常调控差异表达基因的综合分析

图5:IBD诱导的转录组、DNA甲基化组和羟甲基化组变化之间的相关性
A-B. 差异表达且DMR(A)或DhMR(B)富集的基因散点图。颜色表示不同基因组表征,点大小表示每个区域的CpG数量。
- DNA表观遗传标记在IBD诱导的结直肠癌(CRC)发生中的作用的模型。
总结:
本研究采用RRBS+oxRRBS在H.hepaticus感染的IBD Rag2-/-/Il10-/-小鼠模型中,分别绘制了IBD诱导的DNA羟甲基化和DNA甲基化在全基因组中的变化。同时结合RNA-seq基因表达分析,研究结果首次全面揭示了结肠炎症诱导的表观遗传学变化,为进一步研究IBD生物标志物的发现提供了有用的信息,并有可能促进IBD新疗法的未来发展。
关于易基因精准DNA甲基化/羟甲基化测序(oxBS-seq)
羟甲基化5hmC是哺乳动物基因组上的第六碱基,在发育、衰老、神经退行性疾病、复杂疾病及肿瘤发生过程中起重要作用。DNA羟甲基化是近年发现的一种新的DNA修饰并迅速成为研究热点。随着研究的深入,发现之前被认为是检测DNA甲基化标准的重亚硫酸盐测序并不能区分DNA甲基化(5mC)和DNA羟甲基化(5hmC)。
易基因联合剑桥大学建立了化学氧化法结合重亚硫酸盐转化的测序技术(oxidative bisulfite sequencing, oxBS-Seq),该技术不仅可以精确检测DNA甲基化,排除DNA羟甲基化的影响,还可以双文库结合同时单碱基分辨率精确检测DNA羟甲基化。
传统BS转化无法区分5mC和5hmC
传统的Bisulfite测序中,5hmC经过Bisulfite处理后变为CMS,CMS在测序中仍然被读作C碱基,因此不能区分5mC和5hmC。
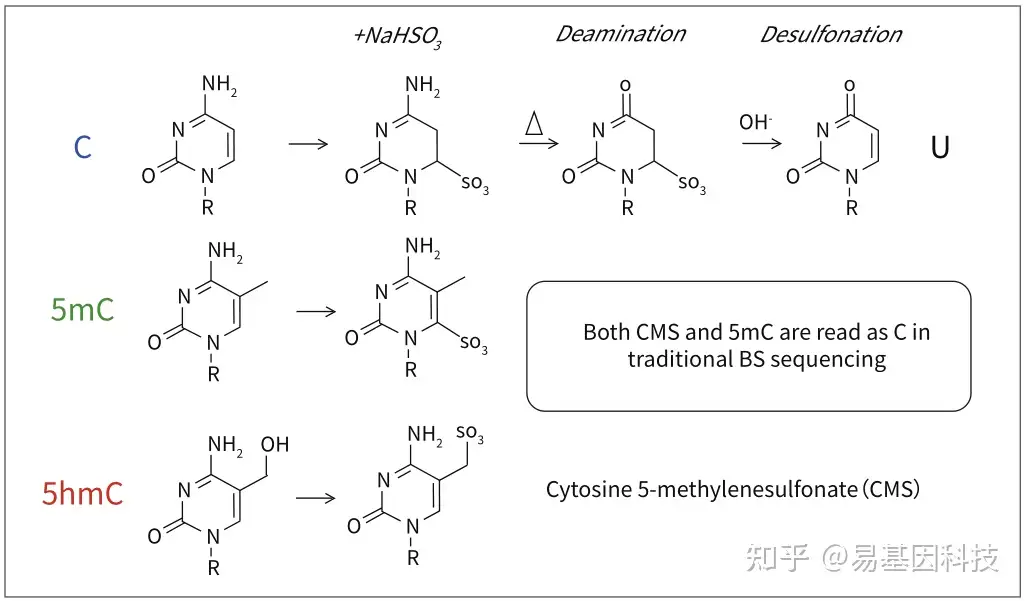
oxBS技术原理
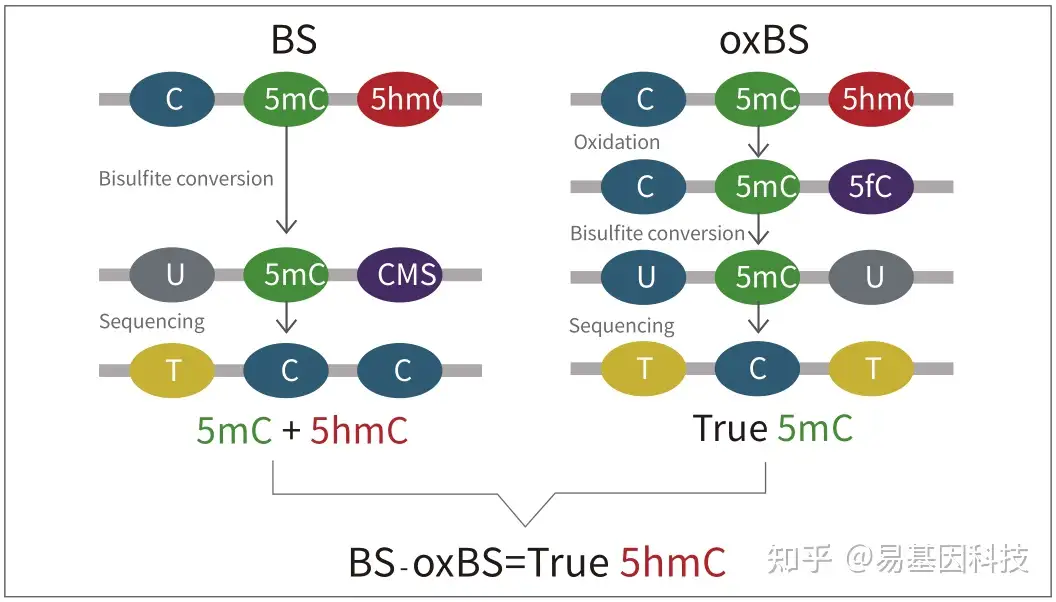
技术优势:
- DNA甲基化检测全新的“标准”;
- 单碱基检测DNA羟甲基化修饰;
- 多重质控标准检测氧化效率和Bisulfite转换率;
- 实验偏好性低,重复性高(R2>0.98);
- 易基因自主研发的甲基化特异性多重PCR引物设计软件;
- 可满足多种测序应用需求:
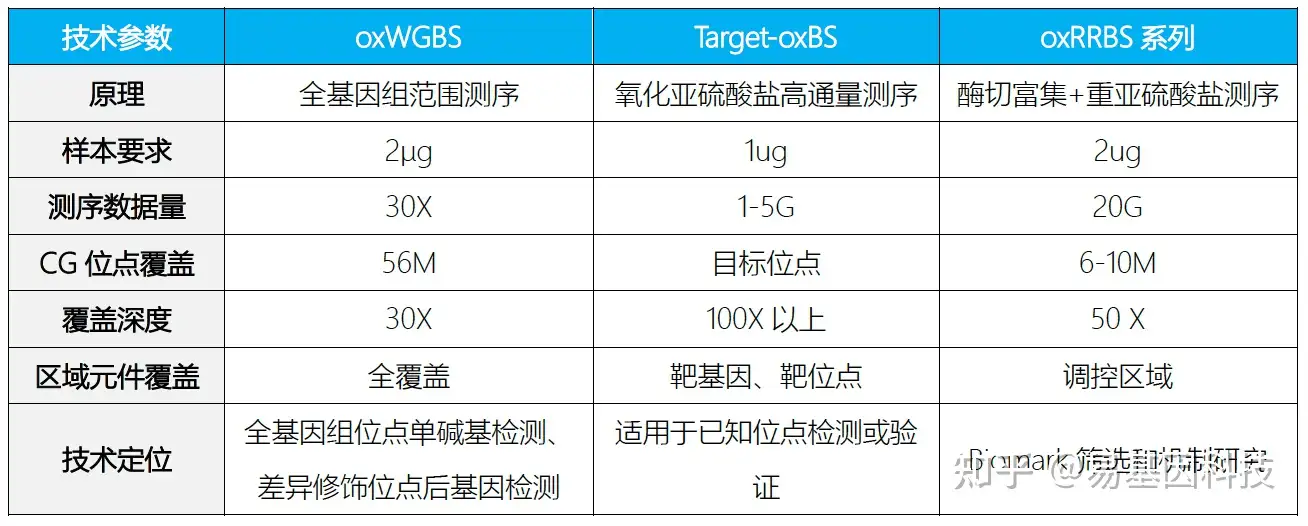
- 全基因组氧化甲基化测序(oxWGBS)
- 简化基因组氧化甲基化测序(oxRRBS)
- 目标区域靶基因氧化甲基化测序(Target-oxBS)。
技术路线:
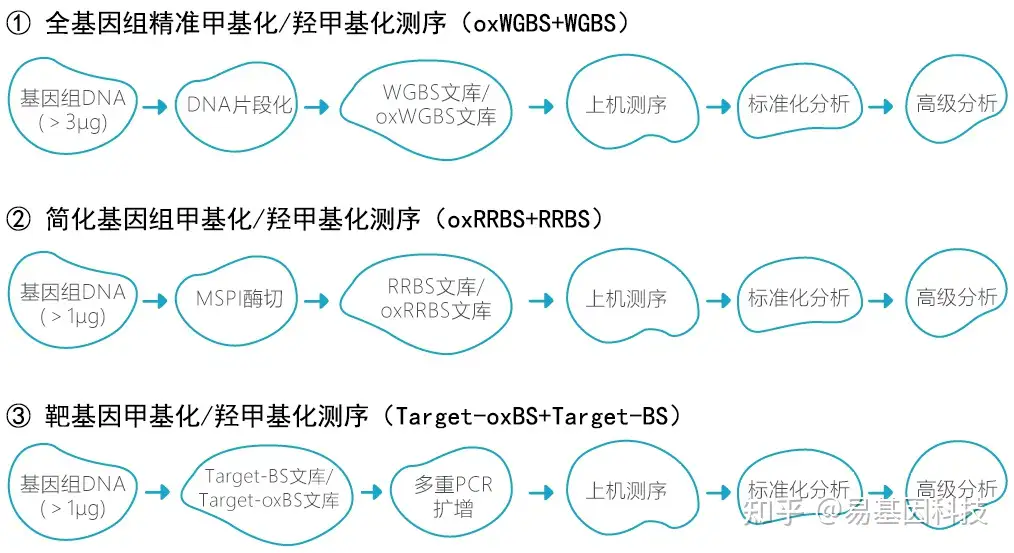
技术指标:
易基因科技提供全面的DNA甲基化研究整体解决方案,技术详情请致电易基因。
参考文献:
Han Q, Kono TJY, Knutson CG, Parry NM, Seiler CL, Fox JG, Tannenbaum SR, Tretyakova NY. Multi-Omics Characterization of Inflammatory Bowel Disease-Induced Hyperplasia/Dysplasia in the Rag2-/-/Il10-/- Mouse Model. Int J Mol Sci. 2020 Dec 31;22(1) pii: ijms22010364.
相关阅读:
一文读懂|精准简化基因组甲基化测序(RRBS+oxRRBS)分析怎么做