DNA甲基化等表观遗传标记在调控不同成熟阶段果实成熟中起着关键作用。m6A甲基化已被证明可以调控番茄成熟,但目前尚不清楚 mRNA m6A甲基化是否对不同类型水果的成熟调控具有功能保守性。
2021年6月,中国科学院植物研究所秦国政研究组在《Genome Biology》杂志发表题为“N6-methyladenosine RNA modification regulates strawberry fruit ripening in an ABA-dependent manner”的研究论文。 该研究以草莓为研究对象,通过MeRIP-seq和对应的RNA-seq等方法揭示了RNA甲基化修饰(m6A)是调控ABA通路的重要成员,进而影响草莓果实成熟。

标题:N6-methyladenosine RNA modification regulates strawberry fruit ripening in an ABA-dependent manner(N6-甲基腺苷RNA修饰以ABA依赖性方式调节草莓果实成熟)
时间:2021-06-03
期刊:Genome Biology
影响因子:IF 12.3/ 1区
技术平台:MeRIP-seq(m6A-seq)、RNA-seq、定量RT-PCR、m6A-IP-qPCR、RIP等
研究摘要:
本研究通过MeRIP-seq等分析表明了m6A甲基化在草莓果实成熟过程中是显著变化。编码序列(CDS)区域中的m6A修饰表现出成熟特异性,并靶向稳定mRNA,而终止密码子周围和3′UTR区域内的m6A修饰通常与相关mRNA丰度呈负相关。研究人员CDS区域鉴定了数千个m6A高甲基化转录本,包括脱落酸(abscisic acid,ABA)生物合成和信号通路中的NCED5、ABAR和AREB1转录本。本研究证明了甲基转移酶MTA和MTB对于草莓果实的正常成熟必不可少,且MTA介导的m6A修饰促进NCED5和AREB1的mRNA稳定性,同时促进ABAR翻译。
通过比较不同成熟阶段草莓果实的m6A甲基组(m6A methylome),研究人员发现m6A修饰在草莓果实成熟过程中呈现动态变化。与番茄果实类似,草莓果实中m6A修饰可富集在终止密码子和3’ UTR区域,且与基因表达呈负相关。而与番茄果实不同的是,草莓中m6A修饰还可大量富集于CDS区域,尤其在成熟启动以后;该区域的m6A修饰整体上与基因表达呈正相关。差异m6A甲基组分析表明,ABA合成基因NCED5及信号转导基因ABAR和AREB1的m6A水平在草莓果实成熟过程中显著升高;m6A修饰可提高NCED5和AREB1的mRNA稳定性,并促进ABAR的翻译效率。进一步研究显示,m6A甲基转移酶MTA和MTB正调控草莓果实成熟,且MTA在NCED5,ABAR和AREB1的m6A修饰中发挥重要作用。值得一提的是,在番茄果实中,m6A修饰可调节DNA去甲基酶基因的稳定性,而在草莓果实中,DNA去甲基酶基因却不受m6A调控,说明m6A修饰通过不同的方式影响番茄和草莓果实成熟。
研究结果
(1)m6A甲基化是草莓果实中mRNA的共有表征
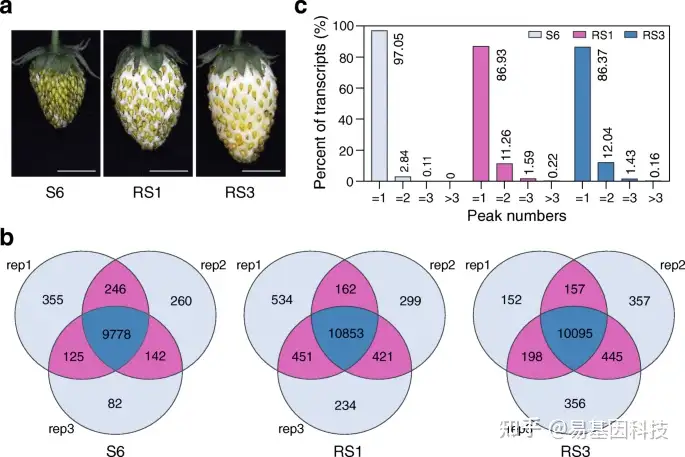
图1:草莓果实中的表观转录组m6A甲基化。
- 不同发育阶段果实的代表性照片。S6,生长期6;RS1,成熟期1;RS3,成熟期3。比例尺=1cm。
- 维恩图描绘了在果实三个发育阶段的m6A-seq三个生物学重复的m6A peaks重叠分析。Rep:重复。
- 样品中不同m6A peaks值的m6A转录本百分比
(2)m6A分布在草莓果实成熟起始阶段表现出显著变化
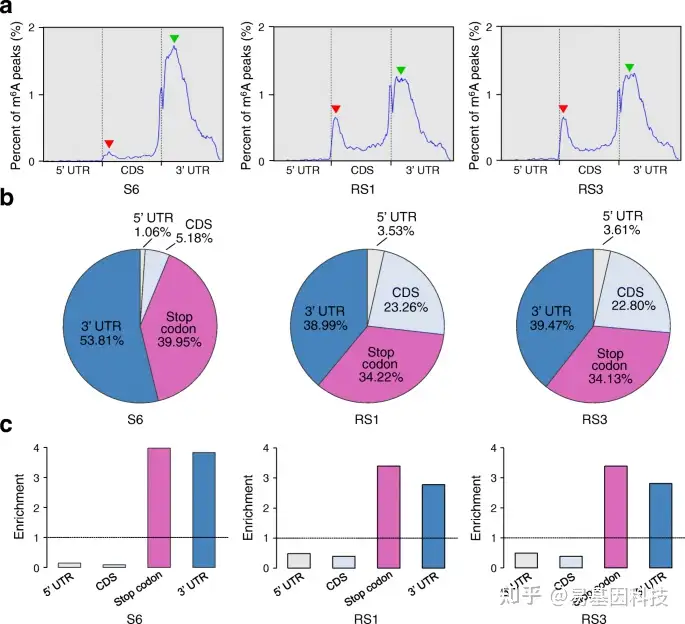
图2:草莓果实成熟过程中的m6A动态分布。
- 沿转录本分布的m6A peaks宏基因组图谱。UTR,非翻译区;CDS,编码序列。红色箭头表示成熟过程中与起始密码子相邻的CDS区域中m6A分布变化,绿色箭头表示终止密码子周围或3′UTR内m6A分布变化。S6,生长阶段6;RS1,成熟阶段1;成熟阶段3。
b-c. 在四个不重叠的转录片段中m6A peaks百分比(b)和相对富集度(c)
(3)m6A甲基化总体上影响草莓果实成熟过程中的mRNA丰度
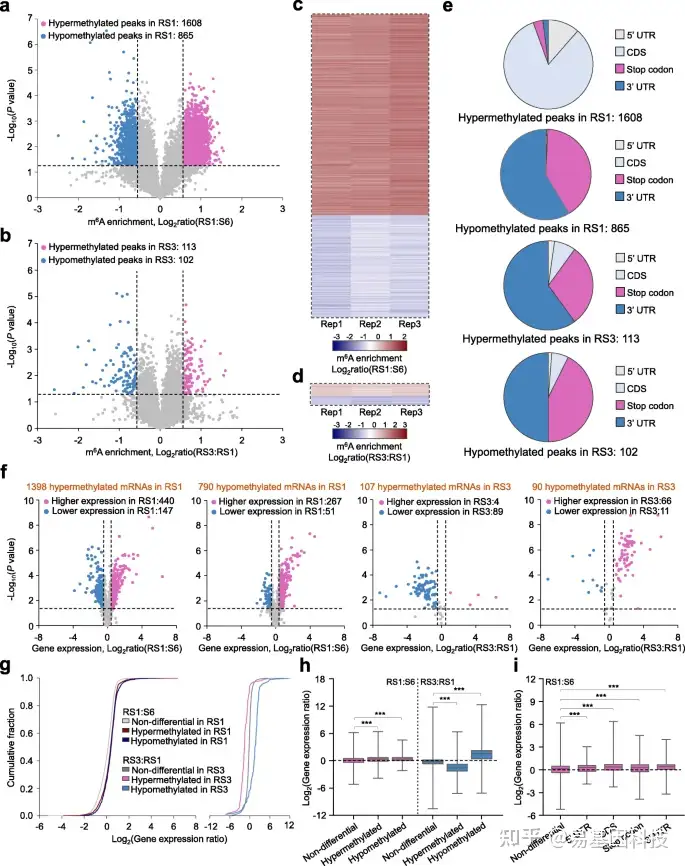
图3:草莓果实中m6A修饰与mRNA丰度的相关性分析。
- 火山图显示与S6期相比,RS1期果实中的高甲基化(红色)和低甲基化(蓝色)m6A peaks。S6,生长阶段6;RS1,成熟阶段1。
- 火山图描绘了与RS1期相比,RS3期果实中的高甲基化(红色)和低甲基化(蓝色)m6A peaks。RS3,成熟阶段3。
c-d. a(c)和b(d)中所示的差异m6A peaks的m6A富集比热图。
e. 差异m6A peaks值的分布特征(如a和b所示)。UTR,未翻译区;CDS,编码序列。
f. 火山图显示a和b中所示的不同m6A peaks转录本的表达比率。RS3期与RS1期比较,mRNA水平显著升高和降低的转录本分别用红色和蓝色突出显示(倍数变化≥1.5;P值< 0.05)。
g-h. RS1期与S6期果实、RS3与RS1果实的基因表达变化累积分布(g)和基因表达比箱形图(h)。m6A修饰转录本分为三类:高甲基化、低甲基化和非差异转录本。
- 基于RS1和S6阶段果实之间的差异m6A peaks分布的基因表达比箱形图。星号表示显著差异(***P< 0.001;Wilcoxon试验)
(4)ABA生物合成和信号通路中的基因在成熟开始时表现出m6A甲基化差异变化
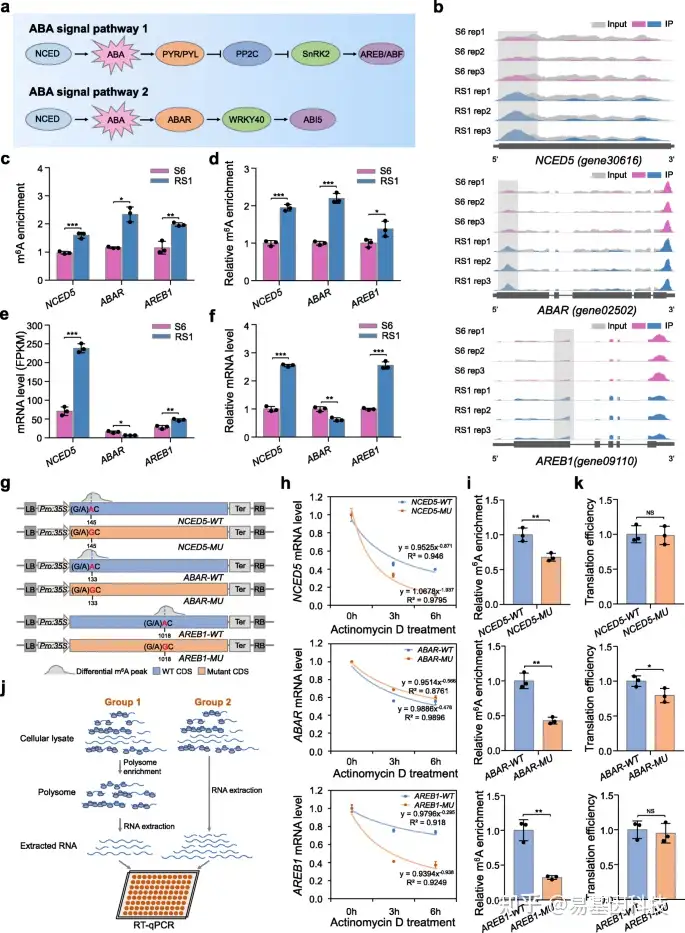
图4:m6A修饰促进ABA通路中基因的mRNA稳定性或翻译。
- 植物中两个关键ABA信号通路的简要模型。
- 综合基因组浏览器(IGB)轨迹显示m6A reads在9-顺式-环氧类胡萝卜素双加氧酶5(NCED5)、假定ABA受体(ABAR)和ABA响应元件结合蛋白1(AREB1)转录本中的分布。高甲基化m6A peaks(倍数变化≥1.5;P值< 0.05)通过阴影框表示。S6,生长阶段6;RS1,成熟阶段1。Rep,重复。
- m6A-seq数据中NCED5、ABAR和AREB1的m6A富集分析。
- m6A免疫沉淀(IP)-qPCR验证m6A富集。
e-f. 通过RNA-seq(e)和定量RT-PCR(f)检测NCED5、ABAR和AREB1的转录水平。肌动蛋白基因在f中用作内部对照。
g. 用于mRNA稳定性分析表达盒的示意图。将NCED5、ABAR和AREB1基因的野生(WT)或突变(MU)CDS分别克隆到由CaMV 35S启动子驱动的pCambia2300载体中。使用定点突变试剂盒将m6A-seq中鉴定并通过SELECT验证的特异性m6A位点(以红色突出显示)从腺苷(A)突变为鸟嘌呤(G)。
h. NCED5、ABAR和AREB1的mRNA稳定性分析。野生(WT)或突变(MU)CDS在本氏烟草叶片中表达。在指定的时间点放线菌素D处理后,提取总RNA,并将本氏N.benthamiana ACTIN基因作为内部对照进行定量RT-PCR分析。
- m6A-IP-qPCR分析显示野生或突变转录本中的相对m6A富集。
j. 翻译效率分析的简要工作流程。
k. NCED5、ABAR和AREB1的翻译效率。翻译效率表示为多体RNA中mRNA相对于总RNA的丰度比。数据表示为平均值±标准差(n=3)。星号表示显著差异(*P< 0.05,**P< 0.01,***P< 0.001;学生t检验)
(5)草莓果实中m6A甲基转移酶表征
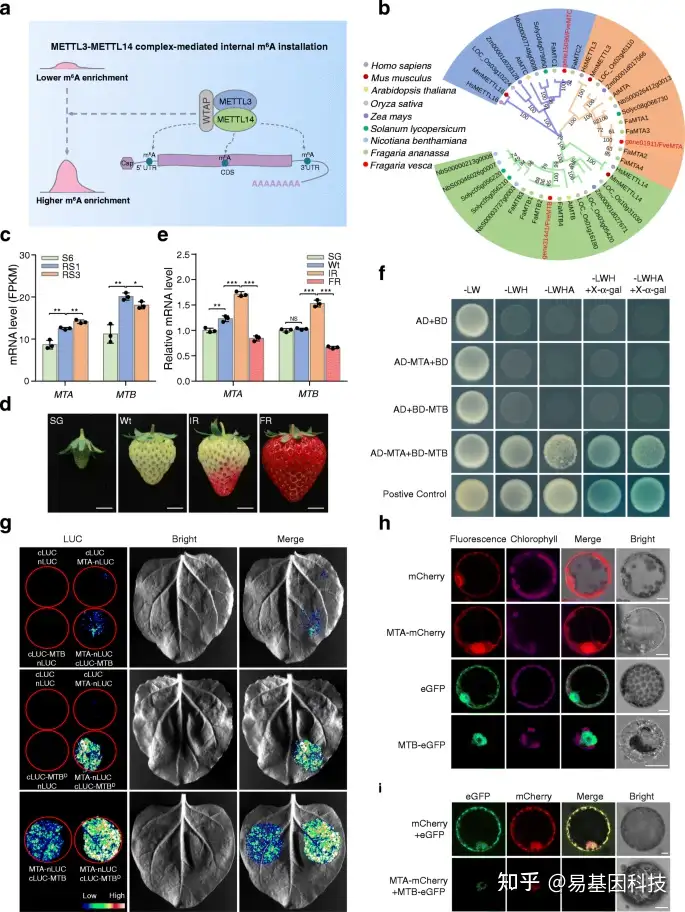
图5:m6A甲基转移酶MTA与草莓中的MTB互作。
- 哺乳动物中甲基转移酶复合体介导的m6A装置的工作模型。m6A甲基转移酶METTL3和METTL14以异二聚体的形式互作并作为内部m6A装置的稳定催化核心。
- 真核生物m6A甲基转移酶的系统发育分析。系统发育树由MEGA(5.2版)生成。给出了每个分支1000次重复的Bootstrap值。物种名称缩写如下:Hs, Homo sapiens; Ms, Mus musculus; At, Arabidopsis thaliana; Os, Oryza sativa; Zm, Zea mays; Sl, Solanum lycopersicum; Nb, Nicotiana benthamiana; Fa, Fragaria × ananassa; Fve, Fragaria vesca。
- RNA-seq揭示了二倍体林地草莓不同发育阶段m6A甲基转移酶基因MTA和MTB的转录水平。S6,生长阶段6;RS1,成熟阶段1;成熟阶段3。
- 不同发育阶段的八倍体栽培草莓果实的代表性图像。SG,小绿色;Wt,白色;IR,初始红色;FR,全红色。比例尺=1cm。
- 通过定量RT-PCR分析八倍体草莓果实中MTA和MTB的转录水平。ACTIN基因被用作内部对照。数据表示为平均值±标准差(n=3)。星号表示显著差异(*P< 0.05,**P< 0.01,***P< 0.001;学生t检验)。
- Y2H分析揭示了MTA和MTB之间的互作。与GAL4激活结构域(AD)融合的MTA(AD-MTA)和与GAL4结合结构域(BD)融合的MTB(BD-MTB)在酵母中共表达。转化子在SD/-Leu/-Trp(-LW)上生长,并在含有或不含有X-α-gal的SD/-Leu/-Trp/-His(-LWH)和SD/-Leu-Trp/-His/-Ade(-LWHA)上进一步筛选。
- LCI分析揭示了MTA和MTB之间的互作。与LUC的N末端融合的MTA(MTA nLUC)与MTB或其与LUC C的C末端融合的MT-A70结构域(cLUC MTB或cLUC MTBD)在benthamiana叶片中共表达。
h-i. MTA和MTB的亚细胞定位(h)和共定位(i)。MTA-mCherry或/和MTB-eGFP融合蛋白在N.benthamiana叶片中瞬时表达。使用表达eGFP或/和mCherry的N.benthamiana叶片作为阴性对照。比例尺=10 μm
(6)m6A甲基转移酶正调控草莓果实成熟
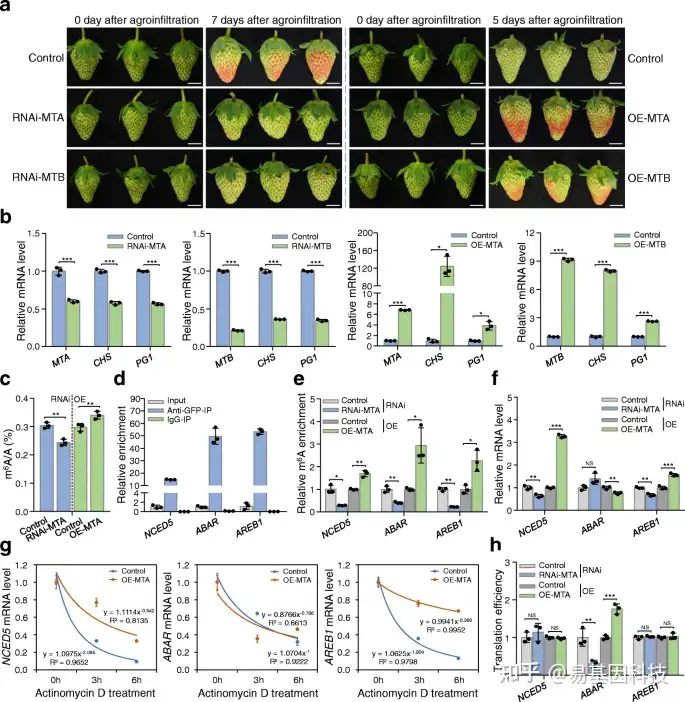
图6:m6A甲基转移酶正调控草莓果实成熟。
- MTA/MTB RNA干扰(RNAi MTA/RNAi MTB)和过表达(OE-MTA/OE-MTB)果实的成熟表型。用空质粒农业渗透的草莓果实作为对照。实验进行三个以上生物学重复的,并给出了具有代表性的结果。比例尺=1 cm。
- 通过定量RT-PCR分析RNAi和过表达果实中MTA和MTB以及两个重要成熟基因CHS和PG1的转录水平。ACTIN基因起内部对照作用。
- LC-MS/MS分析揭示了MTA沉默(RNAi-MTA)或MTA过表达(OE-MTA)果实中整体m6A甲基化水平变化。
- RNA免疫沉淀(RIP)分析揭示了MTA蛋白与NCED5、ABAR和AREB1的转录本结合。从表达MTA-eGFP融合蛋白的草莓果实中提取蛋白质-RNA复合体,并用抗GFP单克隆抗体或小鼠IgG(阴性对照)进行免疫沉淀。
e–h. 在MTA沉默(RNAi MTA)或MTA过表达(OE-MTA)果实中NCED5、ABAR和AREB1的相对m6A富集(e)、基因表达(f)、mRNA稳定性(g)和翻译效率(h)变化。通过m6A-IP-qPCR和定量RT-PCR分别分析m6A的相对富集度和基因表达。在放线菌素D处理后的指定时间点提取总RNA,并进行定量RT-PCR分析mRNA稳定性。翻译效率表示为多体RNA中mRNA相对于总RNA的丰度比。数据表示为平均值±标准差(n=3)。星号表示显著差异(*P< 0.05,**P< 0.01,***P< 0.001;学生t检验)。NS,不显著
(7)MTA介导的m6A甲基化调节编码翻译起始因子和延伸因子的基因
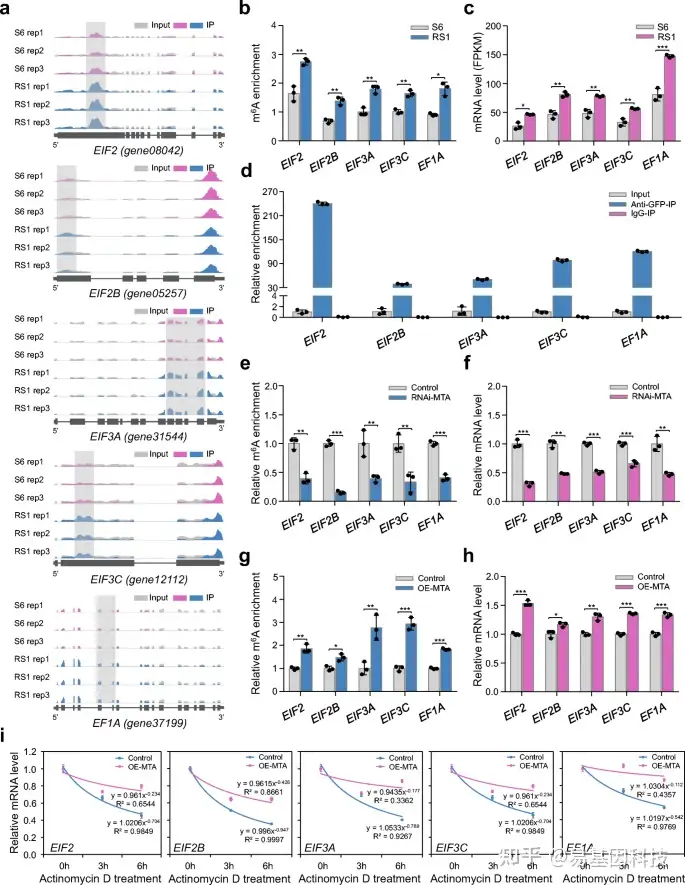
图7:m6A对草莓翻译起始因子或延伸因子的影响。
- IGB轨迹显示编码翻译起始因子(EIF2、EIF2B、EIF3A和EIF3C)和延伸因子(EF1A)编码基因转录本中的m6A reads分布。高甲基化m6A peaks(倍数变化≥1.5;P值< 0.05)与S6阶段的果实的比较分析具有显著差异。S6,生长阶段6;RS1,成熟阶段1。Rep,重复。
- m6A-seq数据中EIF2、EIF2B、EIF3A、EIF3C和EF1A的m6A富集。
- 通过RNA-seq分析EIF2、EIF2B、EIF3A、EIF3C和EF1A的转录水平。
- RNA免疫沉淀(RIP)分析揭示了MTA蛋白与EIF2、EIF2B、EIF3A、EIF3C和EF1A的转录本结合。
e–i . MTA沉默(RNAi MTA)或MTA过表达(OE-MTA)果实中EIF2、EIF2B、EIF3A、EIF3C和EF1A的相对m6A富集度(e,g)、基因表达(f,h)和mRNA稳定性(i)变化。通过m6A-IP-qPCR和定量RT-PCR分别分析m6A的相对富集度和基因表达。ACTIN基因为内部对照。
(8)MTA在m6A mRNA甲基化过程中发挥重要作用
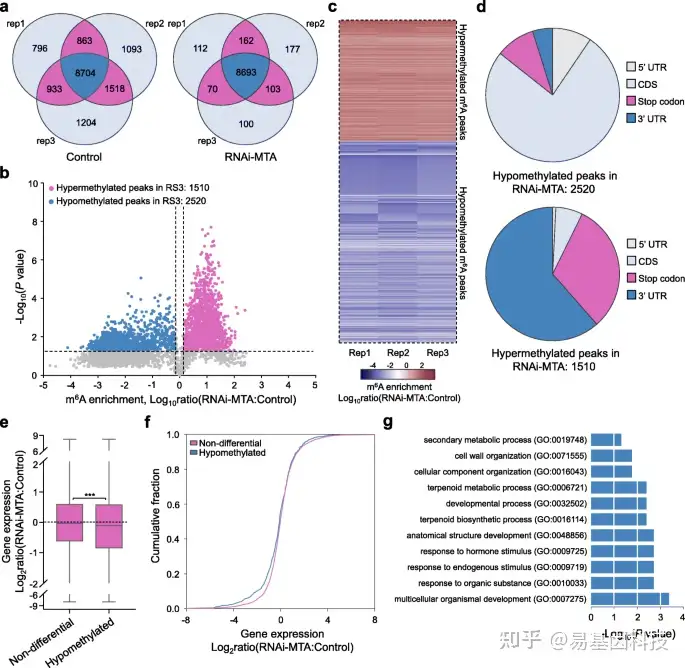
图8:MTA沉默草莓果实中m6A mRNA甲基化组变化。
- Venn图描绘了MTA RNAi(RNAi-MTA)和对照的果实的三个生物学重复m6A-seq实验的m6A peaks重叠分析。
- 火山图显示与对照组相比,MTA沉默果实中的高甲基化(红色)和低甲基化(蓝色)m6A peaks。
- b中所示的差异m6A peaks的m6A富集比热图。
- b中所显示的差异m6A peaks的分布特征。UTR,未翻译区;CDS,编码序列。
e-f. MTA沉默的果实和对照之间的基因表达比率(e)和基因表达变化的累积分布(f)的箱形图。基于RNA-seq数据,分析了与对照相比,MTA沉默果实中低甲基化和非差异性m6A peaks的转录本表达。星号表示显著差异(***P< 0.001;Wilcoxon试验)。
g. 与对照相比,MTA沉默果实中低甲基化m6A peaks的转录本GO富集分析。在agriGO数据库中分析GO富集情况,以及Yekutieli校正P值<0.05
参考文献:
Zhou L, Tang R, Li X, Tian S, Li B, Qin G. N6-methyladenosine RNA modification regulates strawberry fruit ripening in an ABA-dependent manner. Genome Biol. 2021 Jun 3;22(1):168. pii: 10.1186/s13059-021-02385-0. doi: 10.1186/s13059-021-02385-0. PubMed PMID: 34078442.
相关阅读:
研究速递 | MeRIP-seq等从m6A RNA甲基化角度揭示NFATc1对破骨细胞的调控机制
MeRIP-seq+ChIP-seq等揭示m6A甲基化在休眠期转录休眠调控中的潜在功能 | 发育研究