大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
子宫肌瘤(uterine fibroids,UF)是生殖系统最常见的良性肿瘤,也是子宫切除手术最常见的指征。尽管患病率很高,但子宫肌瘤的确切发病机制在很大程度上仍未知。有证据表明,发育期间暴露于激素可能与子宫肌层易感UF发育有关,当发育暴露于内分泌干扰化学物质(endocrine-disrupting chemical,EDC)如己烯雌酚(diethylstilbestrol,DES)、邻苯二甲酸盐(Phthalates)和大豆植物雌激素染料染料木素(genistein),会增加动物模型中UF的发生率、多样性和总体大小。通过充分了解环境EDC影响人类表观基因组的机制,可以更容易地提供明确的临床指导,以解决普通人群中常见的低水平暴露对健康的潜在影响。尽管子宫肌层干细胞(myometrial stem cells,MMSC)已被鉴定为UF的来源细胞,但由于发育暴露于EDC而导致MMSC编程的表观遗传机制尚未表征。
2023年8月31日,芝加哥大学妇产科Qiwei Yang为第一作者和通讯作者、Ayman Al-Hendy为共同通讯作者在《Cell Mol Life Sci》杂志发表题为“Developmental reprogramming of myometrial stem cells by endocrine disruptor linking to risk of uterine fibroids”的研究论文, 研究通过RNA-seq、ChIP-seq、RRBS、功能获得/丧失分析和荧光素酶活性实验,在体外和Eker大鼠模型中研究了子宫肌层干细胞(UFs的假定来源)的发育重编程,揭示了混合谱系白血病蛋白-1(mixed lineage leukemia protein-1,MLL1) / DNA低甲基化介导MMSC发育重编程的表观遗传机制。

标题:Developmental reprogramming of myometrial stem cells by endocrine disruptor linking to risk of uterine fibroids(内分泌干扰素对子宫肌层干细胞的发育重编程与子宫肌瘤风险相关)
时间:2023-08-31
期刊:Cellular and Molecular Life Sciences(细胞与分子生命科学)
影响因子:IF 8 / 1区
技术平台:ChIP-seq、RNA-seq、RRBS、Target-BS、功能获得/丧失分析和荧光素酶活性实验等
研究摘要:
组织和器官生长期间很容易受环境影响,但尚不清楚在此期间暴露是如何导致表观基因组变化和增加子宫肌瘤(UF)等激素相关疾病风险。
研究通过RNA-seq、ChIP-seq、RRBS、功能获得/丧失分析和荧光素酶活性实验,在体外和Eker大鼠模型中研究了子宫肌层干细胞(MMSC)的发育重编程。
研究结果表明,在Eker大鼠发育过程中,当暴露于内分泌干扰化学物质(endocrine-disrupting chemical,EDC)己烯雌酚时,MMSC的雌激素应答转录组发生重编程。MMSC中的重编程基因被称为雌激素应答基因(estrogen-responsive genes,ERGs),由混合谱系白血病蛋白-1(MLL1)和DNA低甲基化机制激活。暴露于天然类固醇的Eker大鼠在发育性暴露于EDC后,MMSC中ERG表达显著上调,从而增强雌激素活性。
本研究揭示了MLL1/DNA低甲基化介导MMSC重编程的表观遗传学机制。EDC暴露表观靶向MMSC,并导致ERG亚群表达的持续变化,在ERG上产生激素印记,导致“超雌激素”表型,并增加UFs的激素依赖性风险。
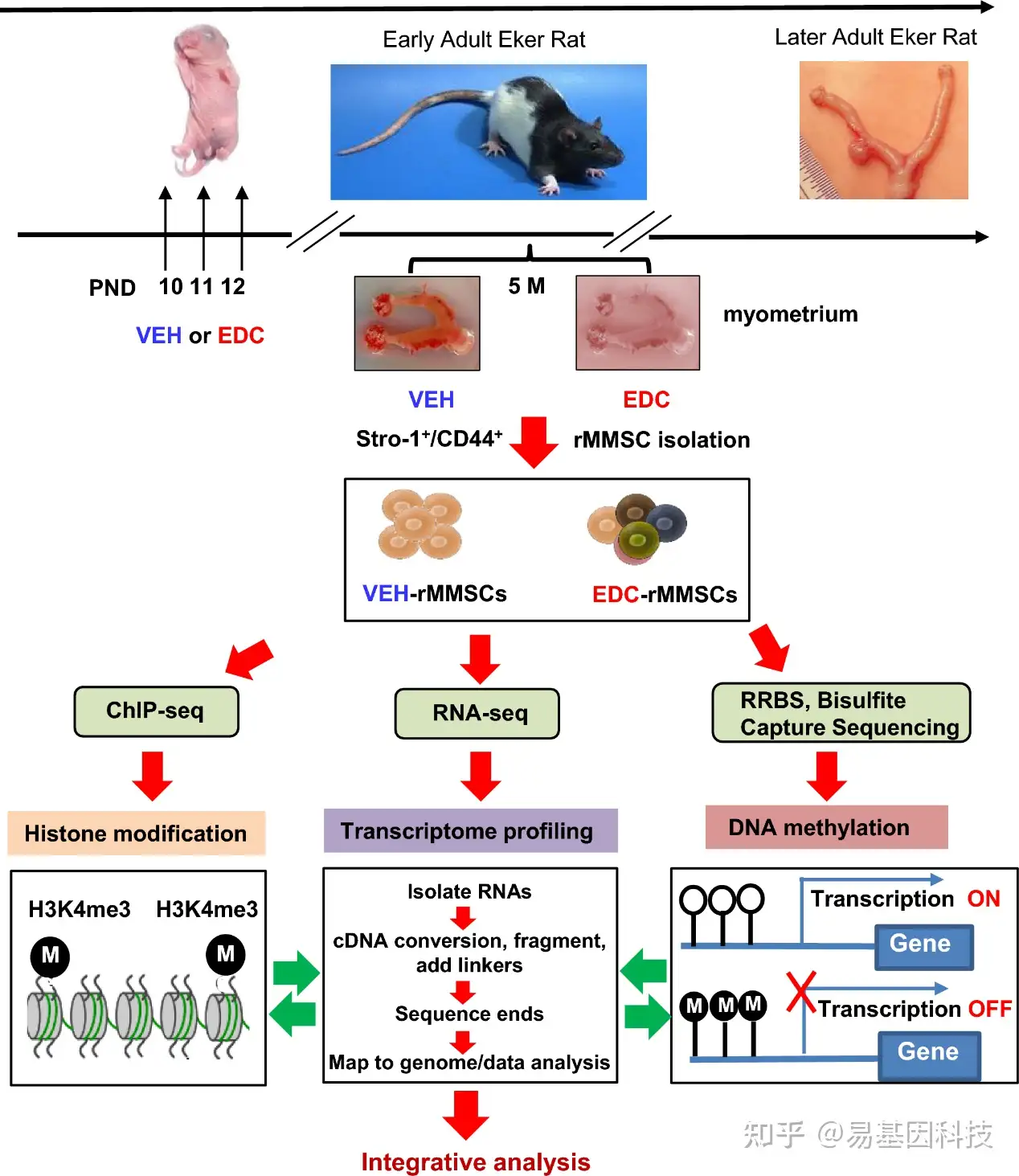
图1:实验图。
Eker大鼠幼崽在出生后10-12天暴露于VEH(每天皮下注射50μl芝麻籽油,vehicle,n=5)和EDC(每天皮下注射10μg己烯雌酚,n=5)。幼崽在5月龄时被处死,代表早期成年期。从大鼠中分离出子宫肌层组织,并使用Stro-1/CD44表面标记进行MMSC分离。利用多组学分析,包括RNA-seq、ChIP-seq和RRBS,分别鉴定转录组、组蛋白修饰和DNA甲基化的整体变化。同时还进行目标基因的亚硫酸盐NGS测序(Target-BS),以检测基因CpG岛的DNA甲基化。
研究结果
(1)Eker大鼠EDC暴露与VEH暴露MMSC中差异转录模式的鉴定
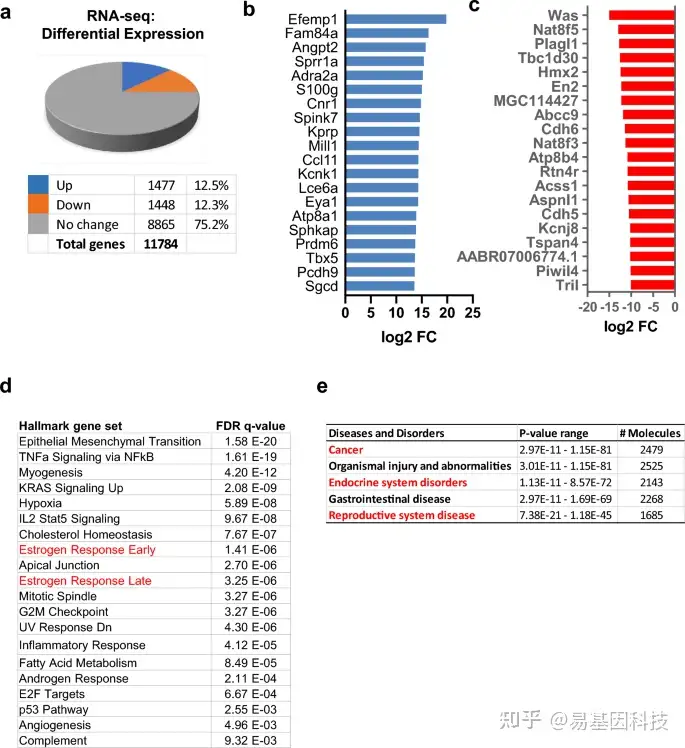
图2:发育性EDC诱导大鼠MMSC基因重编程。
- EDC己烯雌酚组和VEH组的各5只动物混样,采用FACS策略进行MMSC分离。饼图显示通过RNA-seq检测的EDC -MMSC和VEH-MMSC之间的RNA表达变化基因百分比;FDR<0.05时临界值为两倍。
- EDC-MMSC与VEH-MMSC中排名前20位的up-DEG列表。
- EDC-MMSC与VEH-MMSC中排名前20位的down-DEG列表。
- 过表达分析表明,包括雌激素反应通路(红色)在内的多种UF相关通路受早期EDC暴露影响。
- 通过Ingenuity Pathway Analysis(IPA)分析发育性EDC己烯雌酚暴露对相关疾病的影响,用红色突出显示
(2)发育性EDC暴露重编程MMSC中的雌激素应答基因
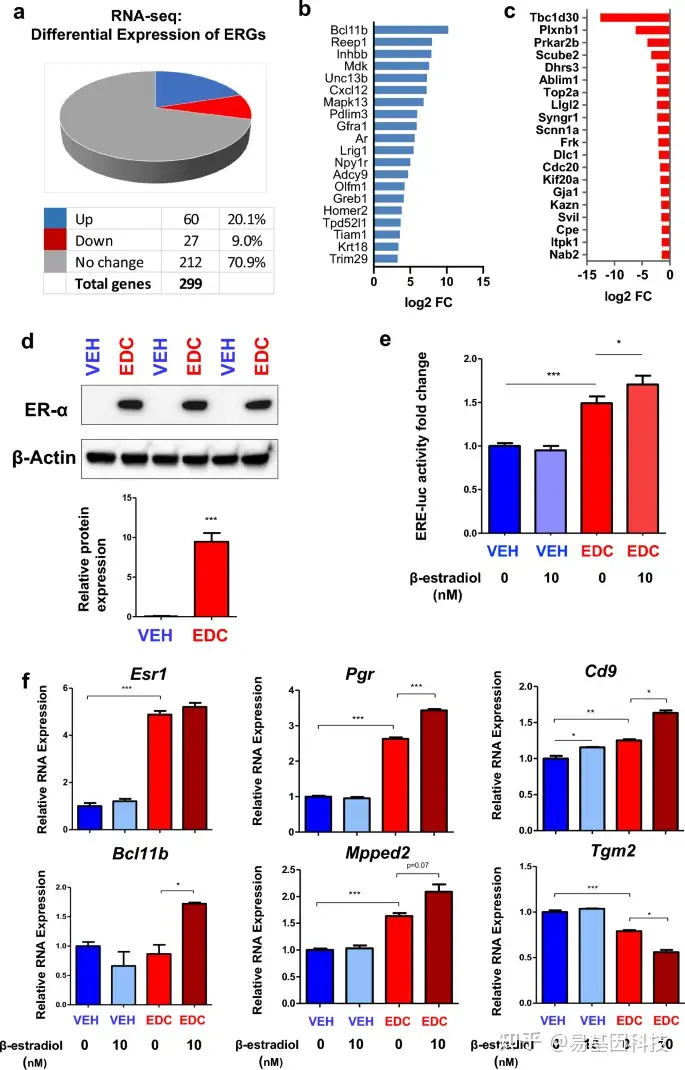
图3:转录谱分析显示雌激素应答基因的重编程。
- RNA-seq分析,与VEH-MMSCs相比,EDC-MMSC中RNA表达变化的ERG百分比饼图。
- 在EDC-MMSC与VEH-MMSC中RNA-seq分析显示差异表达的前20个up-ERG列表。
- 在EDC-MMSC与VEH-MMSC中RNA-seq分析显示差异表达的前20个down-ERG列表。
- 与VEH-MMSC相比,EDC-MMSC中的ER-a表达上调。从EDC-MMSC和VEH-MMSC制备裂解液,并使用ER-a抗体进行Western blot分析。
- 比较EDC-MMSC和VEH-MMSC在有或无雌激素(10nM)时的萤光素酶活性。
- DES(己烯雌酚,diethylstilbestrol)-MMSC和VEH-MMSC中有或无雌激素时ERG表达变化。学生t检验,*p < 0.05,**p < 0.01,***p < 0.001
(3)EDC暴露激活MLL1表观遗传通路并通过H3K4me3破坏MMSC的表观基因组
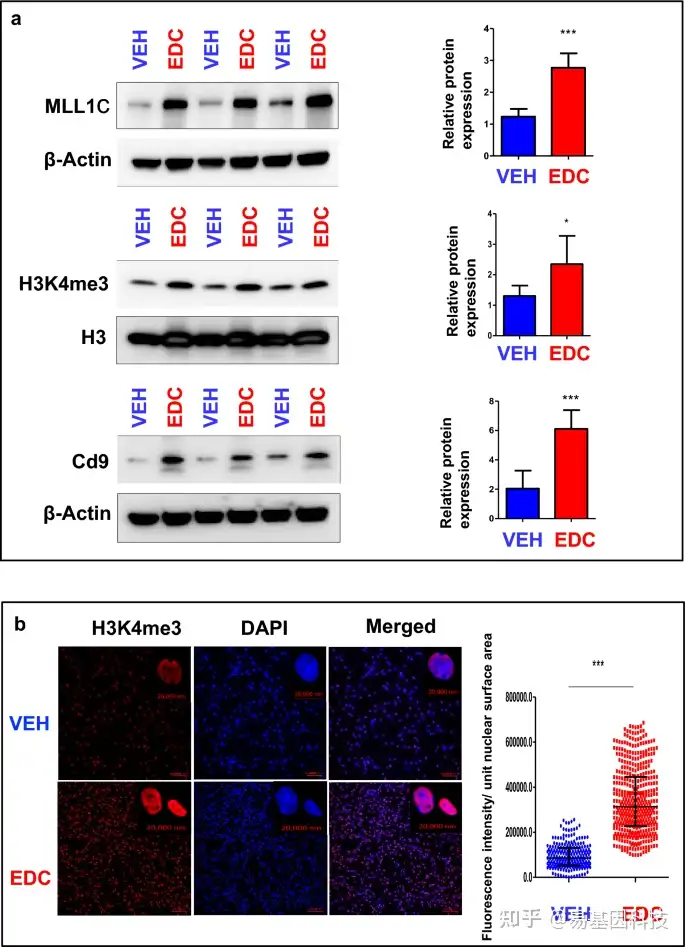
图4:EDC发育性暴露激活MLL1。
- 用抗MLL1、H3K4me3和Cd9抗体进行Western blotting以分析MLL1C(MLL1活化形式)、H3K4me3和Cd9水平。总H3和β-肌动蛋白用作负载对照。生物学重复(n=3)进行定量。
- EDC-MMSC与VEH-MMSC中H3K4me3染色和定量分析的共聚焦成像图(Confocal imaging)。使用三个单独的细胞盖片进行定量。学生t检验,*p<0.05,**p<0.01,***p<0.001
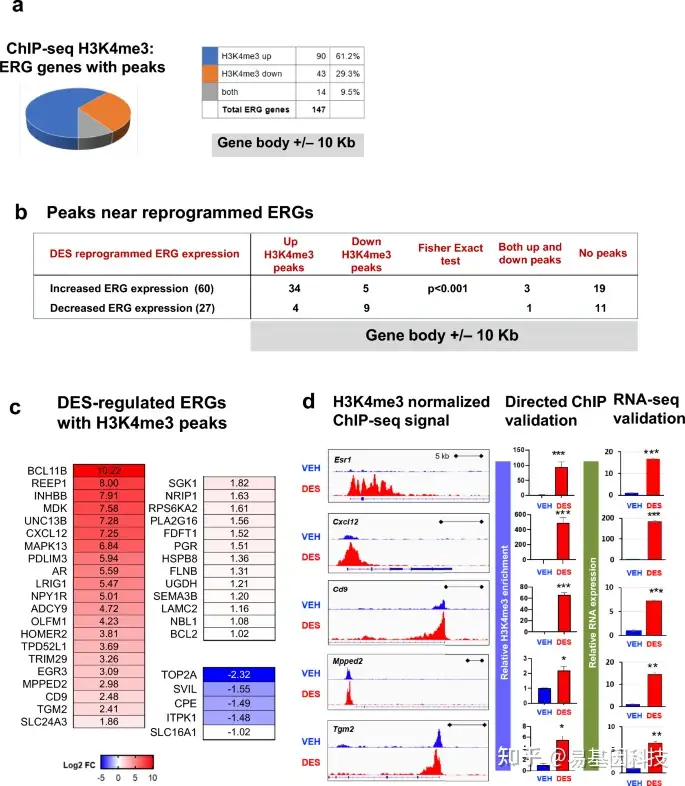
图5:EDC暴露破坏MMSC的表观基因组。
- ERG peaks数量饼图。
- EDC暴露将H3K4me3与ERG RNA表达整合。
- EDC调控H3K4me3 peaks的ERG。
- Integrative Genomics Viewer直方图显示Esr-1、Cxcl12、Cd9、Mpped2和Tgm2中的H3K4me3占比(左侧panel)。对于每个基因,上方和下方的浏览器图像显示VEH-MMSC(蓝色轨迹)和EDC-MMSC(红色轨迹)中H3K4me3 peaks分布的选定区域的扩展视图。中间panel:通过ChIP-qPCR对H3K4me3靶基因进行定向ChIP测序验证。右侧panel;通过q-PCR进行RNA测序验证。学生t检验,*p<0.05,**p<0.01,***p < 0.001
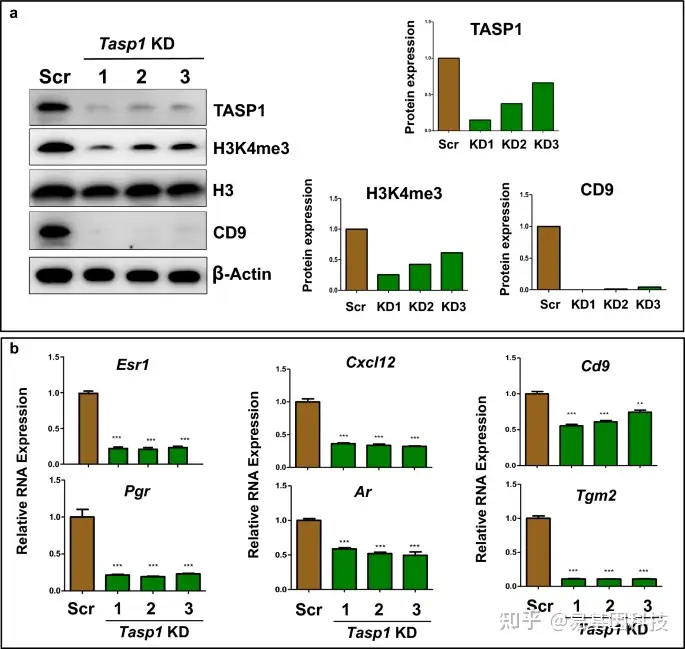
图6:Tasp1敲除逆转了EDC暴露诱导的重编程ERG。
- 在EDC-MMSC中使用shRNA慢病毒(pLKD-TASP1)质粒敲低TASP1表达后,进行Western blot分析以确定TASP1在MLL1介导的表观遗传学通路中的作用。分别使用抗TASP1、H3K4me3和CD9抗体进行Western blot分析TASP1、H3K4me3、和CD9蛋白的表达水平(左图)。生物学重复(n=3)用于定量。使用Image J(右图)进行定量分析。
- EDC暴露诱导的6个重编程ERG(Esr-1、Pgr、Cxcl12、Ar、Cd9和Tgm2)表达上调被EDC-MMSC中Tasp1敲除所逆转。通过qPCR检测EDC-MMSC炒慢病毒(scrambled lentivirus)感染或三种不同个体Tasp1敲低慢病毒的ERG RNA表达。*p<0.05,**p<0.01,***p < 0.001;学生t检验
(4)EDC暴露重编程甲基化并导致ERG表达变化。
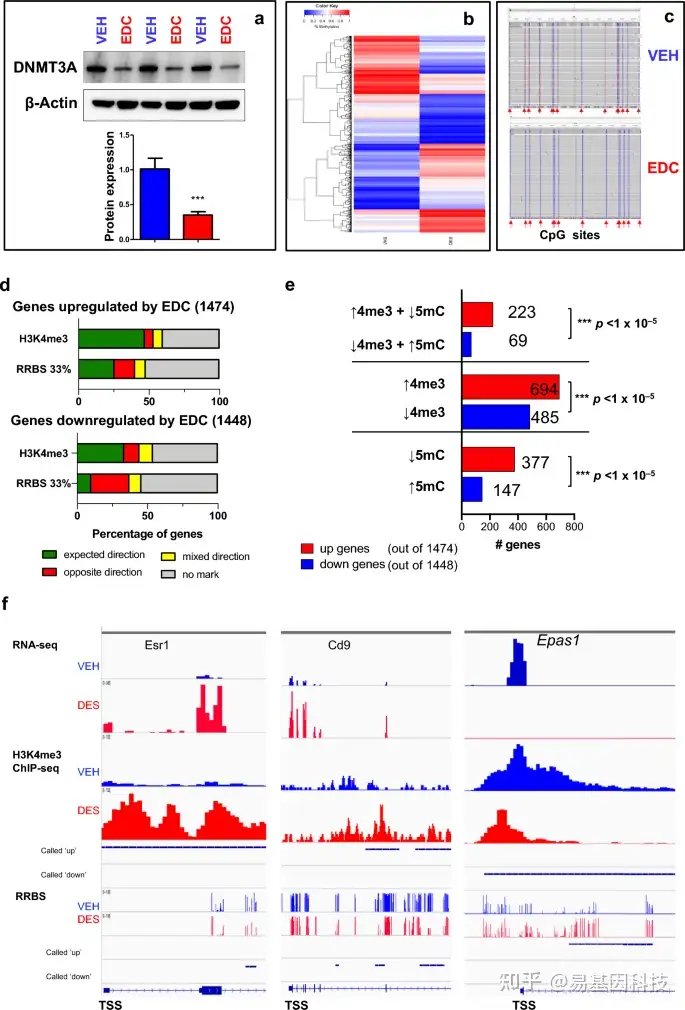
图7:发育性EDC暴露诱导甲基化组变化并通过DNA启动子甲基化导致ERG重编程。
- Western blot分析检测VEH-MMSC和EDC-MMSC中DNMT3A的蛋白水平。
- VEH-MMSC和EDC-MMSC中DNA甲基化基因热图。
- 通过靶向亚硫酸盐NGS检测Esr1的甲基化分布,覆盖其CpG岛上的14个CpG位点。
- 具有H3K4me3和DNA甲基化状态的EDC调控基因百分比。
- 具有H3K4me3和DNA甲基化状态的基因数量。
- 综合基因组查看器(IGV)图和整合多组学分析。EDC-MMSC中H3K4me3富集、DNA甲基化和RNA表达peaks用红色表示,而VEH-MMSC中的H3K4me3富集、DNA甲基化和RNA表达peaks用蓝色表示。学生t检验,***p<0.001
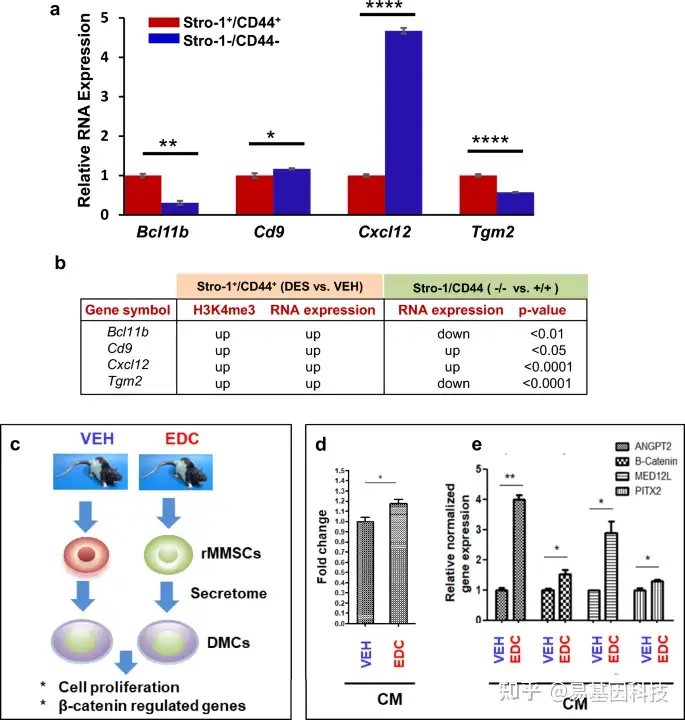
图8:DES-MMSC中ERG的特异性重编程影响差异子宫肌层细胞(differential myometrial cells,DMC)。
- DES-MMSC和DES-DMC之间的ERG(包括Bcl11b、Cd9、Cxcl12和Tgm2)差异表达条形图。
- H3K4me3状态与MMSC和DMC之间重编程ERG的比较表达相关性,以VEH-MMSC为参考。p值显示Stro-1/CD44双阳性和双阴性细胞之间基因表达的显著差异。浅蓝色背景的基因表明基因表达的两个比较(Stro-1/CD44双阳性细胞 vs 双阴性细胞,或Stro-1+/CD44+-DES vs VEH)呈负相关。白色背景基因表明两个比较呈正相关。
- 实验设计示意图。从EDC-MMSC和VEH-MMSC中制备无血清条件培养基(CM)。来自成年大鼠子宫肌层的DMC,分别在EDC-MMSC和VEH-MMSC的CM中培养2天。
- MTT检测EDC-MMSC和VEH-MMSC的CM对DMC增殖的作用。
- qPCR检测EDC-MMSC和VEH-MMSC的CM对DMC中β-catenin和β-catenin调控基因(Angpt2、Med12l和Pitx2)表达的影响。*p<0.05,**p<0.01,***p < 0.0001
研究结论
本研究通过RNA-seq、ChIP-seq、RRBS、功能获得/丧失分析和荧光素酶活性实验等多组学分析,表明了外源性雌激素(EDC)直接靶向并影响MMSC的表观遗传学,而MMSC是UF的细胞来源。通过表观基因组分析揭示了调控MMSC转录谱机制的新见解。早期暴露于EDC(如DES)会导致MMSC中组蛋白和DNA甲基转移酶介导的多种基因表达模式发生显著变化,包括各种雌激素应答基因(ERG)。这些基因表达的变化改变了MMSC表征,导致“超雌激素(hyper-estrogenic)”表型,其特征是DNA修复能力降低和激素依赖性子宫疾病(如UFs)的风险增加。
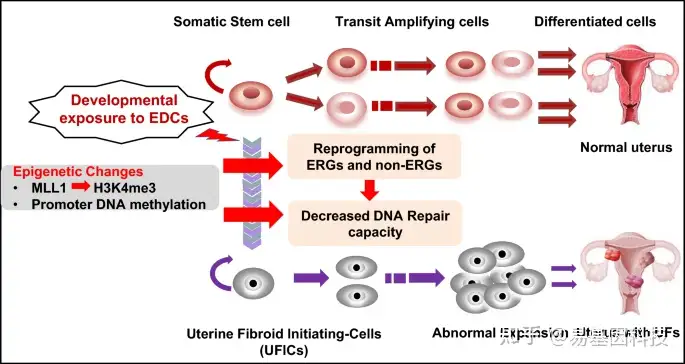
图9:MMSC表观基因组发育重编程模型。
环境风险因素(包括EDC暴露)通过组蛋白修饰和DNA甲基化破坏表观基因组,从而导致MMSC向UF起始细胞转化,并最终导致UF形成
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:
关于易基因简化基因组甲基化测序研究解决方案
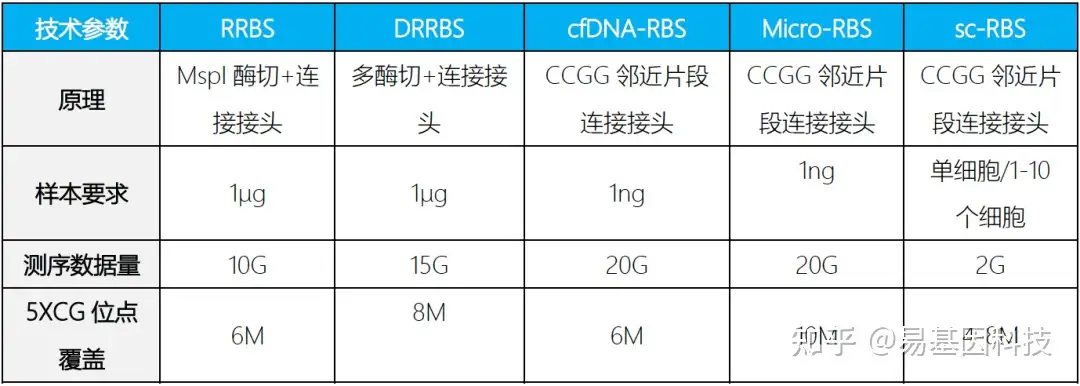
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
易基因提供全面的表观基因组学和表观转录组学、DNA与蛋白互作测序方案,详询易基因:0755-28317900.

参考文献:
Yang Q, Ali M, Treviño LS, Mas A, Al-Hendy A. Developmental reprogramming of myometrial stem cells by endocrine disruptor linking to risk of uterine fibroids. Cell Mol Life Sci. 2023 Aug 31;80(9):274.
相关阅读:
科研进展:ChIP-seq等揭示转录因子NRF1调控原始生殖细胞发育、增殖和存活的表观遗传机制
项目集锦 | 易基因近期染色质免疫共沉淀测序(ChIP-seq)研究成果
亮点研究|oxRRBS+RRBS揭示牦牛下丘脑在神经调节和髓鞘形成中的表观调控机制
项目文章 | RRBS揭示基于DNA甲基化驱动基因的肾透明细胞癌预后模型的鉴定和验证