大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
脑卒中(俗称中风)是导致死亡和长期残疾的主要原因,尤其是对于老龄人来说。脑卒中的平均生存时间为6-7年,许多患者存在身体残疾和晚期认知功能障碍。脑卒中后的神经功能预后取决于许多因素,包括年龄、梗死面积和部位、遗传因素以及脑修复程度。脑卒中后的脑修复包括重建血脑屏障(blood–brain barrier,BBB)结构和功能等。鉴定和表征导致BBB修复不完全的因素和过程是成功修复BBB和改善脑卒中后修复的关键。
近年来,表观遗传学作为组织修复的高阶调控机制,在脑卒中发病机制和脑卒中后修复中发挥着重要作用。表观遗传学指不依赖于DNA序列主要变化的基因表达改变,它依赖于环境因素和基因组之间的互作。三种表观遗传因素对生物体功能至关重要:DNA甲基化、翻译后组蛋白修饰(例如乙酰化、甲基化、磷酸化和泛素化)和非编码RNA。DNA甲基化模式变化是包括脑血管疾病在内的多种疾病的标志。DNA甲基化在脑卒中和调节脑卒中后BBB修复中发挥着重要作用。然而,DNA甲基化与脑卒中后BBB功能和结构完整性之间的相关性仍不清楚。
2023年02月28日,美国密歇根大学医学院Anuska V. Andjelkovic团队在《Fluids Barriers CNS》杂志发表题为“Epigenetics and stroke: role of DNA methylation and effect of aging on blood–brain barrier recovery”的研究文章,该研究通过简化基因组甲基化测序(RRBS)及对应的转录组测序(RNA-seq)分析了幼龄(young)和老龄(old)小鼠脑卒中后BBB修复的甲基化和转录组图谱,强调了可能影响BBB修复的年龄相关过程和因素。

标题:Epigenetics and stroke: role of DNA methylation and effect of aging on blood–brain barrier recovery(在血脑屏障修复中的DNA甲基化作用和衰老影响)
时间:2023-02-28
期刊:Fluids and Barriers of the CNS
影响因子:IF 6.961
技术平台:RRBS、RNA-seq等
研究摘要:
血脑屏障(BBB)功能的不完全修复是脑卒中预后的主要影响因素。脑卒中后BBB如何修复仍然未知。新的证据表明,表观遗传因素在调节脑卒中后血脑屏障(BBB)修复中起着重要作用。本研究旨在评估血栓栓塞性(thromboembolic,TE)脑卒中后脑微血管的表观遗传学和转录组表征,以鉴定限制BBB修复的潜在原因。研究结果表明,与年龄匹配的对照组相比,对从幼龄(6月龄,young组)和老龄(18月龄,old组)小鼠脑卒中后7天分离的微血管进行RNA测序(RNA-seq)和简化基因组亚硫酸氢盐测序(RRBS)分析。比对至注释基因,脑卒中后微血管的DNA甲基化分析鉴定出老龄小鼠中的11287个差异甲基化区域(DMR)和幼龄小鼠中的9818个DMR。这些DMR在编码细胞结构蛋白(如细胞连接和细胞极性、肌动蛋白细胞骨架、细胞外基质)、转运蛋白和通路(如钾跨膜转运蛋白、有机阴离子和无机阳离子转运蛋白、钙离子转运蛋白)、参与内皮细胞过程的蛋白质(如血管生成/血管生成、细胞信号传导和转录调控)基因中富集。甲基化和转录组测序的整合分析鉴定出细胞连接(occludin)、肌动蛋白重塑(ezrin)以及Rho GTPase(RhoA和Cdc42ep4)等信号通路变化。衰老作为异常甲基化的枢纽,通过结构蛋白表达(如claudin-5)的深度变化(高甲基化和抑制)以及参与内皮细胞向间质转化相关基因(如Sox9、Snai1)的激活、抗血管生成和表观遗传调控,影响BBB修复过程。这些结果表明,DNA甲基化通过调节与BBB修复相关的过程在脑卒中后BBB修复中起重要作用,并普遍与增强BBB损伤过程有关。
项目设计:
幼龄C57BL/6雄性小鼠(6月龄)和老龄C57BL/6雄性小鼠(18-20月龄),将血栓栓塞混悬液(8 mg/100μl)注入小鼠颈内动脉。从脑微血管中分离基因组DNA和RNA,进行RRBS和RNA-seq测序分析。
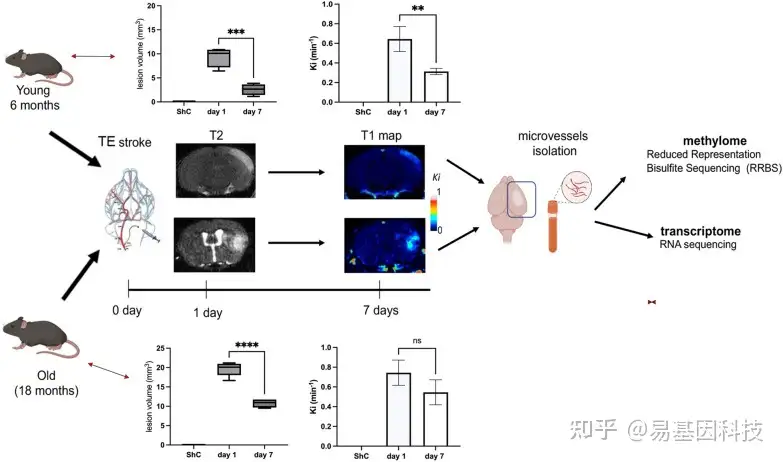
图1:实验流程图
幼龄和老龄小鼠经血栓栓塞(TE)脑卒中处理。在安乐死和微血管分离之前,分别在第1天和第7天进行T2 MRI和T1 MRI以确定梗塞面积和BBB通透性(流入速率常数Ki)。微血管检测甲基化和转录组变化。图表表示平均值 ±SD,n=5,**p<0.01,***p<0.001,****p<0.0001。
实验结果
(1)脑卒中后BBB修复的DNA甲基化谱
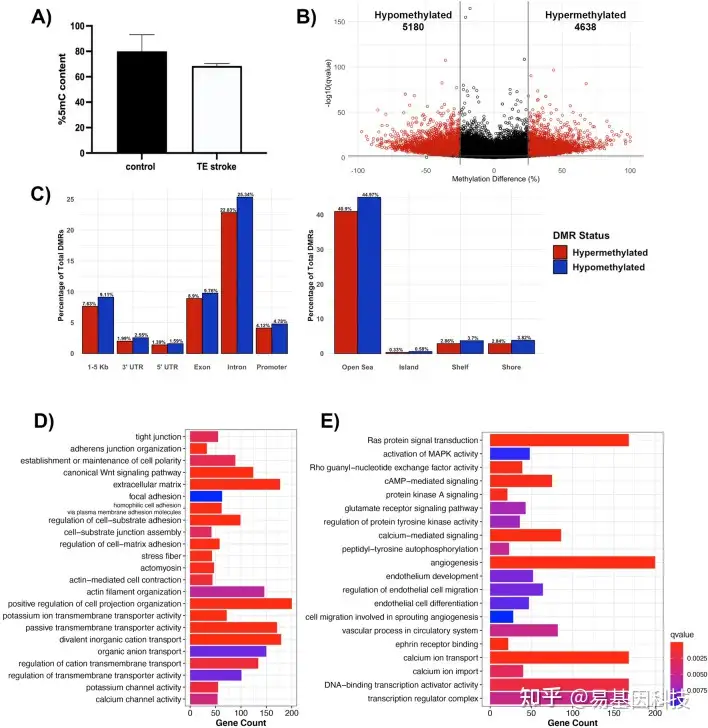
图2:TE脑卒中后血脑屏障修复的DNA甲基化分析
A. 在6月龄的小鼠中,全基因组DNA甲基化检测显示在TE脑卒中后整体甲基化降低趋势。数据表示5mC平均百分比±SEM,n=2
B. 差异甲基化区域(DMR)火山图,x轴和y轴分别显示甲基化差异百分比和-log10(q值)。显著DMR(甲基化差异>25%和q值<0.01)红色显示,无统计显著性DMR以黑色显示。
C. 条形图总结了每个基因组区域(左)和CpG区域(右)的显著DMR百分比。高甲基化DMR百分比为红色,低甲基化DMR百分比为蓝色。
D-E. 结构(D)和信号(E)GO分析的基因过表达数据汇总,x轴显示基因数。
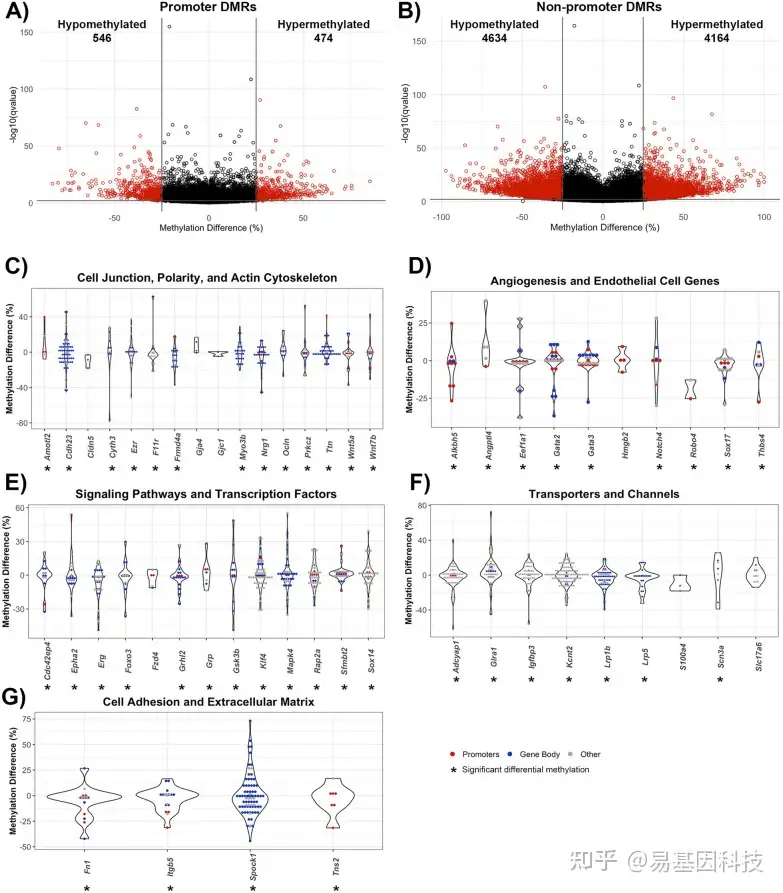
图3:幼龄小鼠(6月龄)脑卒中后BBB修复中DMR的基因组位点。
A-B. 基因启动子区(A)和非启动子区域(B)的DMR火山图
C-G. 细胞连接、极性和肌动蛋白细胞骨架(C)、血管生成和内皮基因(D)、信号通路和转录因子(E)、转运蛋白和通道(F)、细胞粘附和细胞外基质(G)相关基因的差异甲基化。所有DMR,无论统计显著性如何,均表示为与内皮细胞生物学相关的基因,含有显著DMR的基因用星号(*)表示。基因启动子DMR为红色,基因体DMR为蓝色。位于转录起始点上游1-5 kb或无基因组位置的其他DMR为灰色
(2)甲基化组变化对脑卒中后血脑屏障(BBB)修复转录组谱的影响
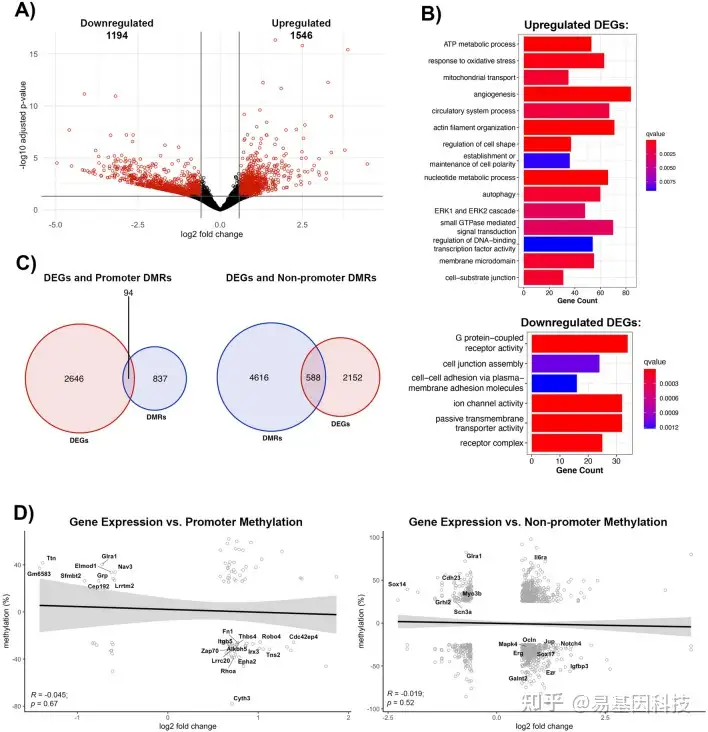
图4:6月龄小鼠脑卒中后BBB DNA甲基化组和转录组图谱比较分析
- 差异表达基因(DEG)火山图。X轴表示log2倍变化,y轴表示−log10(调整后的p值)。显著性DEG(log2FC的绝对值 > 0.58和调整后的p值 < 0.05)为红色,无统计学显著性差异的DEG为黑色。
- 上调DEG(上)和下调DEG(下)基因过表达分析。
- 具有差异甲基化启动子(左)或非启动子区域(右)的DEG数量Venn图。仅统计显著DEG和DMR。如果一个基因包含多个DMR,则该基因仅计一次。
- 基因表达和甲基化发生显著变化的基因,基因表达变化与启动子甲基化(左)或非启动子甲基化(右)之间的Pearson相关性,log2FC位于x轴,甲基化变化位于y轴。启动子区域甲基化(R=−0.045,p=0.67)和非启动子区甲基化(R=−0.019,p=0.52)均与基因表达呈负相关。标记基因与内皮细胞生物学相关
(3)衰老对脑卒中后修复血脑屏障(BBB)DNA甲基化组和转录组谱的影响

图5:衰老对脑卒中后BBB恢复过程中DNA甲基化谱的影响
A. 全基因组DNA甲基化检测表明,老龄小鼠(18月龄)TE脑卒中后分离微血管(BBB)的整体甲基化水平没有变化。数据表示5mC含量的平均百分比±SEM,n=2
B. 老龄TE脑卒中小鼠的DMR火山图,x轴甲基化百分比差异和y轴−log10(q值)。显著DMR以红色突出显示(甲基化差异>25%和q值<0.01),不显著DMR显示为黑色。
C. 每个基因组区域和CpG区域显著DMR百分比条形图。高甲基化DMR用红色表示,低甲基化DMR用蓝色表示。
D-E. 结构(D)和信号(E)的基因过表达GO富集分析。
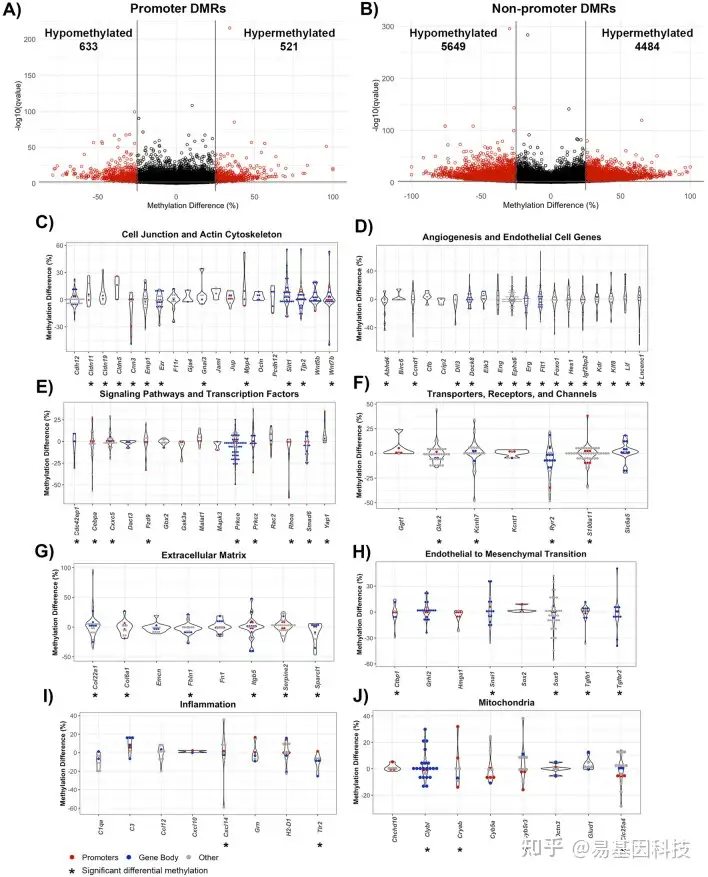
图6:老龄小鼠(18月龄)脑卒中后BBB修复中DMR的基因组位点
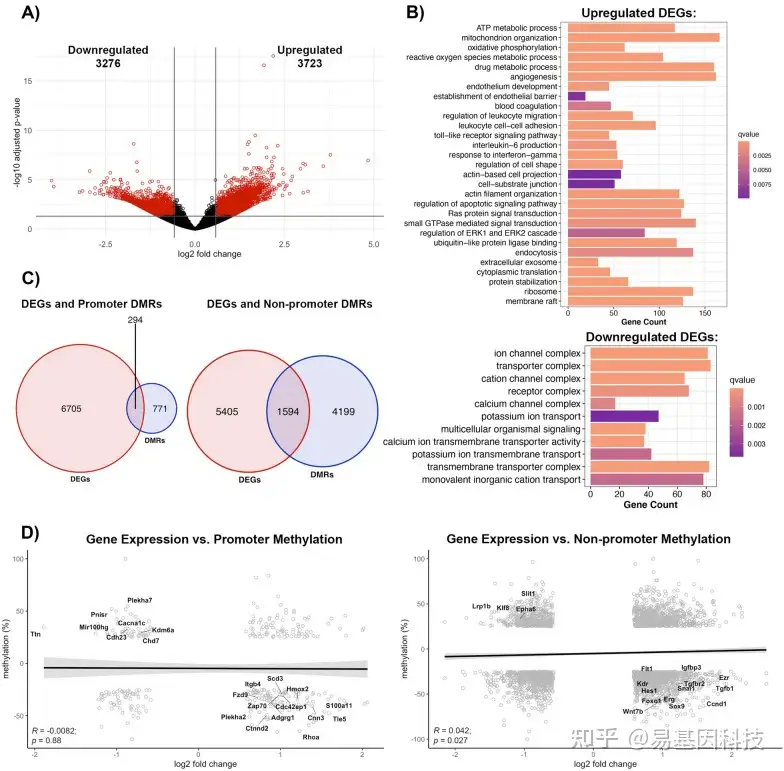
图7:老龄小鼠(18月龄)脑卒中后血脑屏障DNA甲基化组图谱比较分析
(4)幼龄和老龄小鼠脑卒中后BBB恢复的共有和特异性转录组和DNA甲基化组图谱
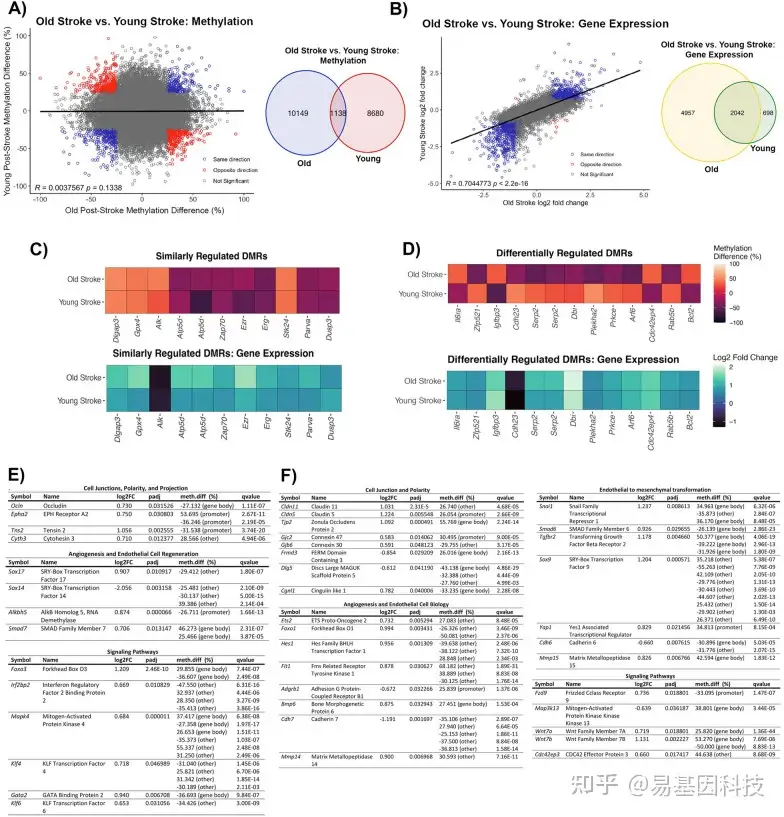
图8:幼龄和老龄小鼠脑卒中后BBB中DNA甲基化变化的共有和特异性DEG
结论:
脑卒中后血脑屏障(BBB)修复过程中存在DNA甲基化变化,可以调节修复BBB特性的过程,同时激活和调节一些导致BBB损伤的因素。本研究通过RRBS、RNA-seq等组学分析研究了不同龄小鼠脑卒中后恢复中的全基因组DNA甲基化模式,定义了BBB恢复的持续过程(即血管生成、屏障维持、通过Rho-GTPase和Wnt/β-catenin蛋白信号以及EndMT进行的信号调控)及其可能限制恢复过程的表观遗传学(甲基化)变化,导致严重的BBB损伤。本研究为脑卒中后血脑屏障和脑血管功能下降的病因提供了新的见解,并为药物发现和靶向治疗开辟了新的途径。
关于易基因简化基因组甲基化测序研究解决方案
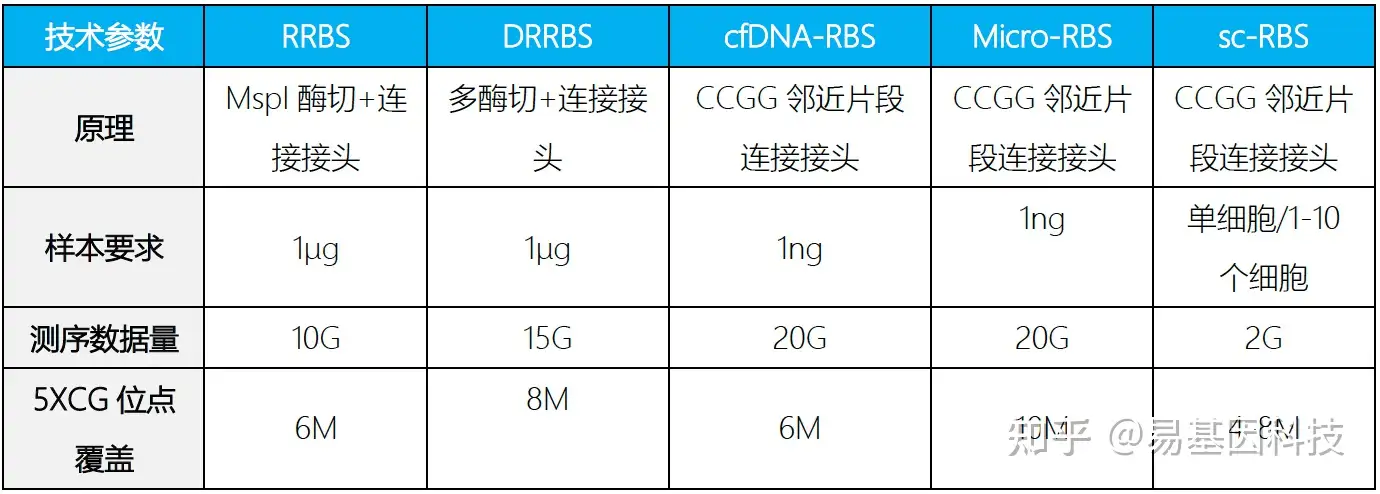
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
易基因科技提供全面的DNA甲基化研究整体解决方案。
参考文献:
Phillips CM, Stamatovic SM, Keep RF, Andjelkovic AV. Epigenetics and stroke: role of DNA methylation and effect of aging on blood-brain barrier recovery. Fluids Barriers CNS. 2023 Feb 28;20(1):14.
相关阅读:
全基因组CpG密度和DNA甲基化分析方法比较(MeDIP、RRBS和WGBS)| 研究综述
3文一览:简化甲基化测序(RRBS)技术优势及研究成果(医学+物种保护+农学)